2023
06-17
06-17
热点综述 | 基于人工智能的多组学分析助力癌症精准医学研究
 AI & 癌症研究多组学技术和人工智能算法的同步发展推动了癌症精确医学的发展。2023年1月《 Seminars in Cancer Biology 》发表了一篇综述文章, 全面总结了基于人工智能的多组学肿瘤分析的最新进展,重点介绍了基于人工智能的多组学技术在癌症诊断、分类、早期筛查、反应评估和预后预测方面的应用。多组学技术随着高通量生物技术的发展,已经开发了多种组学技术来表征不...阅读... 阅 读 全 部 >
AI & 癌症研究多组学技术和人工智能算法的同步发展推动了癌症精确医学的发展。2023年1月《 Seminars in Cancer Biology 》发表了一篇综述文章, 全面总结了基于人工智能的多组学肿瘤分析的最新进展,重点介绍了基于人工智能的多组学技术在癌症诊断、分类、早期筛查、反应评估和预后预测方面的应用。多组学技术随着高通量生物技术的发展,已经开发了多种组学技术来表征不...阅读... 阅 读 全 部 >
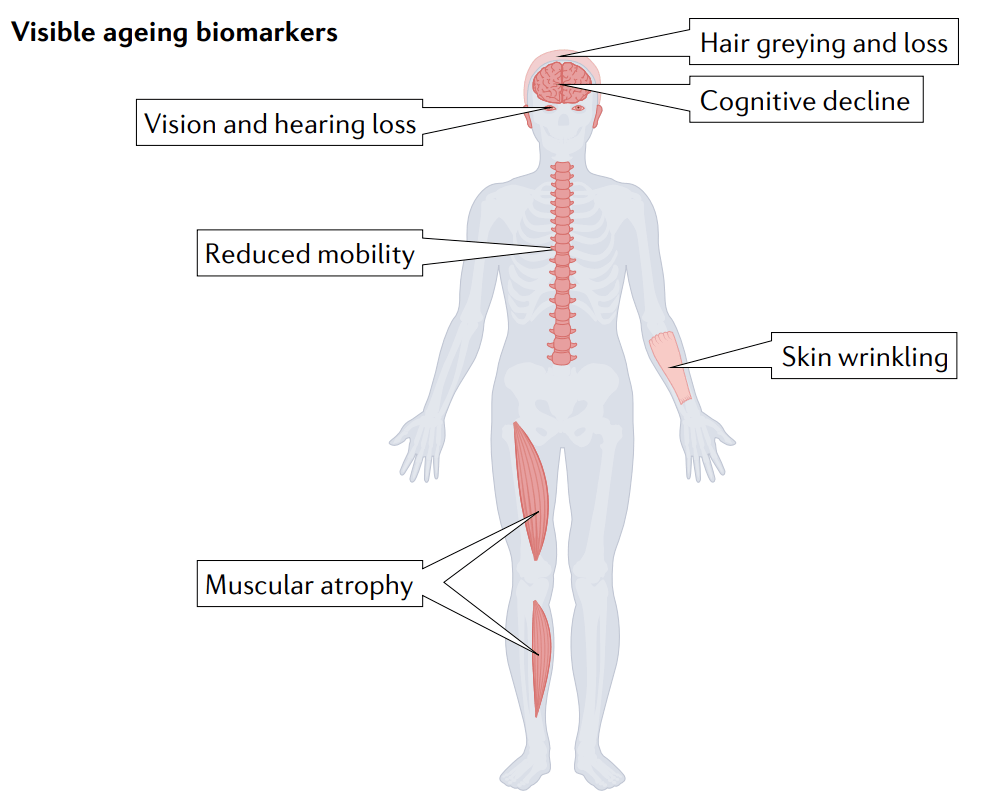 机器学习的兴起为大数据时代掀起新的浪潮。机器学习是人工智能的一个子集,通过泛型算法从数据集中发现模式和相关性并建立逻辑,并根据数据分析结果做出最佳决策和预测。目前机器学习已大量应用于各行各业,助力各产业或科学研究的深度提升。美国加州斯坦福大学的Tony Wyss-Coray团队于2022年6月在Nature Reviews Genetics上发表的一篇名为“Measuring biolo...阅读...
机器学习的兴起为大数据时代掀起新的浪潮。机器学习是人工智能的一个子集,通过泛型算法从数据集中发现模式和相关性并建立逻辑,并根据数据分析结果做出最佳决策和预测。目前机器学习已大量应用于各行各业,助力各产业或科学研究的深度提升。美国加州斯坦福大学的Tony Wyss-Coray团队于2022年6月在Nature Reviews Genetics上发表的一篇名为“Measuring biolo...阅读... 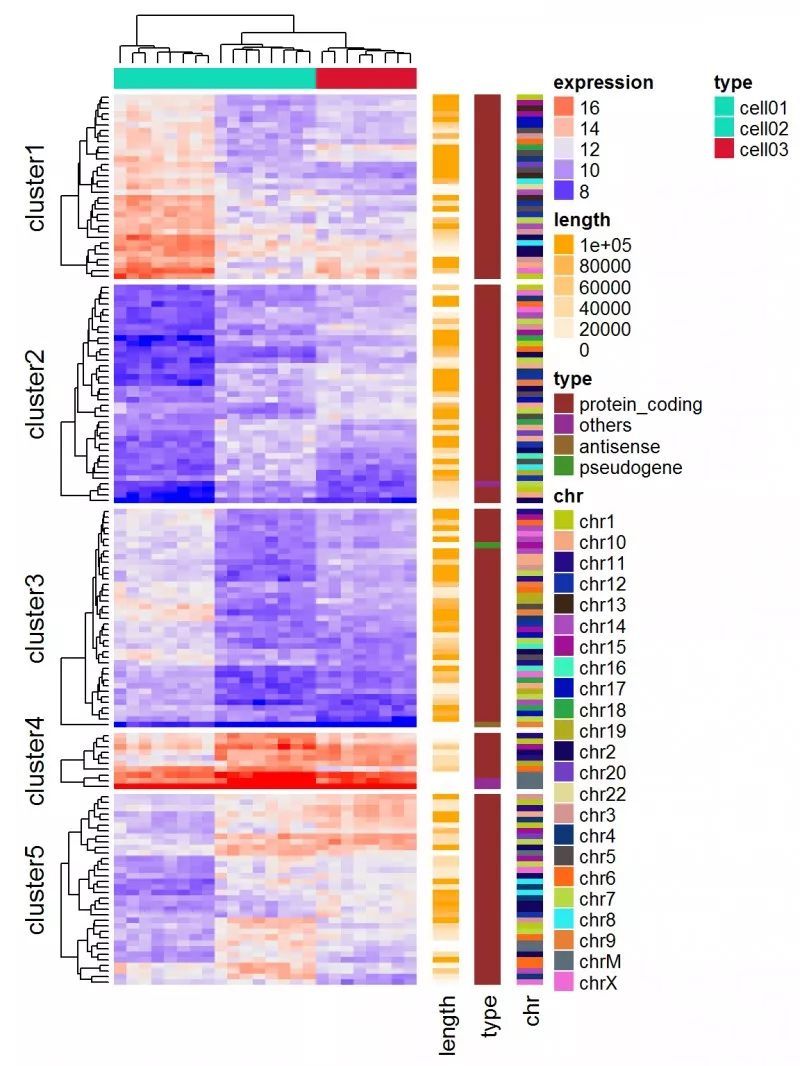 作者简介taoyan:R语言中文社区特约作家,伪码农,R语言爱好者,爱开源。个人博客: https://ytlogos.github.io/简介本文将绘制静态与交互式热图,需要使用到以下R包和函数:heatmap():用于绘制简单热图的函数heatmap.2():绘制增强热图的函数d3heatmap:用于绘制交互式热图的R包ComplexHeatmap:用于绘制...阅读全文>>...
作者简介taoyan:R语言中文社区特约作家,伪码农,R语言爱好者,爱开源。个人博客: https://ytlogos.github.io/简介本文将绘制静态与交互式热图,需要使用到以下R包和函数:heatmap():用于绘制简单热图的函数heatmap.2():绘制增强热图的函数d3heatmap:用于绘制交互式热图的R包ComplexHeatmap:用于绘制...阅读全文>>...  往期我们学习过一个表达趋势折线图+模块热图+GO富集注释的组合类图表绘制,详情可戳:《炫一个模块热图+GO功能富集组合图展示空间功能特征!》 ,后期图表美化主要使用AI完成,如下图。这两天看到另一个比较有意思的R包ClusterGVis,可以同时完成聚类+富集,并生成发表级的组合图表,效果如下。非常方便省事,适合懒人。下面就来学习一番!#相关R包下载与载入:devtools::install_gi...
往期我们学习过一个表达趋势折线图+模块热图+GO富集注释的组合类图表绘制,详情可戳:《炫一个模块热图+GO功能富集组合图展示空间功能特征!》 ,后期图表美化主要使用AI完成,如下图。这两天看到另一个比较有意思的R包ClusterGVis,可以同时完成聚类+富集,并生成发表级的组合图表,效果如下。非常方便省事,适合懒人。下面就来学习一番!#相关R包下载与载入:devtools::install_gi...  正常情况下,我们绘制的常规热图都是直直的,这样更便于我们进行数据的展示和阅读;但上周收到小伙伴的问题:能不能把热图“卷”起来,绘制环状的热图?当然也是可以的,我们先看几个文献中的案例:( BMC Genomics ,2018) ( Microbial Biotechnology , 2017) ( Cell ,2021) 环状热图我们也经常会在论文中看到,用法和热图相同,但更适合于需要展示较多基因...
正常情况下,我们绘制的常规热图都是直直的,这样更便于我们进行数据的展示和阅读;但上周收到小伙伴的问题:能不能把热图“卷”起来,绘制环状的热图?当然也是可以的,我们先看几个文献中的案例:( BMC Genomics ,2018) ( Microbial Biotechnology , 2017) ( Cell ,2021) 环状热图我们也经常会在论文中看到,用法和热图相同,但更适合于需要展示较多基因... 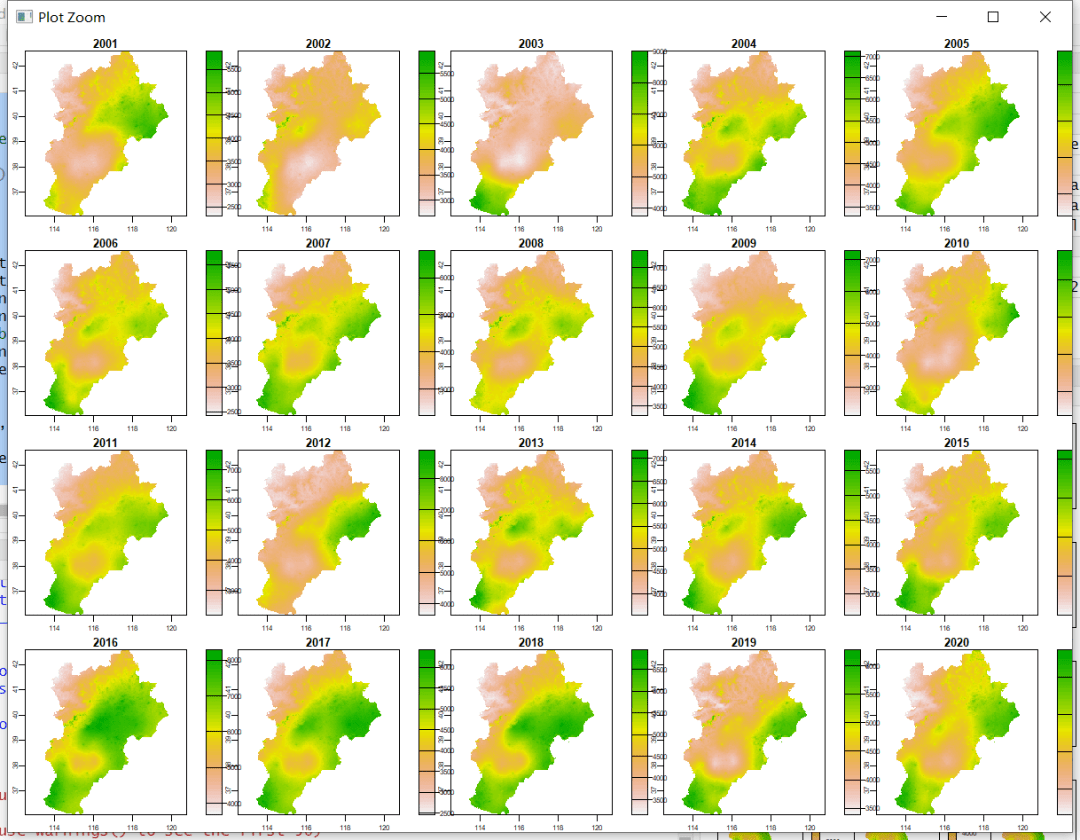 R语言栅格时间序列回归分析——整体和逐像元计算,并行计算前面给大家分享了 2001-2020年1km分辨率中国降水量数据 ,有同学问如何做回归分析,接下来以一元线性回归举例,从整体上和逐像元两种方法来计算。使用的R语言程序包terra 包,栅格计算,支持并行计算tidyverse 包,R语言数据处理可视化必备,内含 ggplot2ggpmis...阅读全文>>...
R语言栅格时间序列回归分析——整体和逐像元计算,并行计算前面给大家分享了 2001-2020年1km分辨率中国降水量数据 ,有同学问如何做回归分析,接下来以一元线性回归举例,从整体上和逐像元两种方法来计算。使用的R语言程序包terra 包,栅格计算,支持并行计算tidyverse 包,R语言数据处理可视化必备,内含 ggplot2ggpmis...阅读全文>>...  大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。在全球范围内,牛为60多亿人提供了重要的营养来源。传染病是养牛生产的主要限制因素,且许多疾病人畜共患,因此与人类健康直接相关。近年来牛的许多复杂性状遗传基础得到了广泛研究,但不同动物的大量重要表型变化仍无法解释,人们越来越关注非遗传变异(包括基因表达、DNA甲基化和染色质)与重要牛表型的潜在相关性。为充分了解牛性状的非遗...阅读全文&...
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。在全球范围内,牛为60多亿人提供了重要的营养来源。传染病是养牛生产的主要限制因素,且许多疾病人畜共患,因此与人类健康直接相关。近年来牛的许多复杂性状遗传基础得到了广泛研究,但不同动物的大量重要表型变化仍无法解释,人们越来越关注非遗传变异(包括基因表达、DNA甲基化和染色质)与重要牛表型的潜在相关性。为充分了解牛性状的非遗...阅读全文&...  生信分析类研究,可以说是近两年火热的科研话题了。随着大数据时代的来临,「拍脑门」想选题的时代已一去不复返,从「别人的数据中」挖掘出自己所需的研究核心已经是大势所趋。更何况,据传说——做生信分析的人可以「不做实验」、「无需编程」、「坐在电脑前」、「轻松发文章」。哇!想想都能笑出声来……忍不住暗搓搓的摩拳擦掌一番,打开一篇生信文章,一窥其真容,看看能不能从...阅读全文>>...
生信分析类研究,可以说是近两年火热的科研话题了。随着大数据时代的来临,「拍脑门」想选题的时代已一去不复返,从「别人的数据中」挖掘出自己所需的研究核心已经是大势所趋。更何况,据传说——做生信分析的人可以「不做实验」、「无需编程」、「坐在电脑前」、「轻松发文章」。哇!想想都能笑出声来……忍不住暗搓搓的摩拳擦掌一番,打开一篇生信文章,一窥其真容,看看能不能从...阅读全文>>...  多序列比对(Multiple Sequence Alignment, MSA),对多个序列进行对位排列。这通常需要保证序列间的等同位点处在同一列上,并通过引进小横线(-)以保证最终的序列具有相同的长度。在生物信息分析中,我们有时需要进行多序列比对,Biopython可以帮我们实现,特别是使用linux系统的同学,biopython值得拥有。两种读取方法Biopython提供了两种方法读取多序......
多序列比对(Multiple Sequence Alignment, MSA),对多个序列进行对位排列。这通常需要保证序列间的等同位点处在同一列上,并通过引进小横线(-)以保证最终的序列具有相同的长度。在生物信息分析中,我们有时需要进行多序列比对,Biopython可以帮我们实现,特别是使用linux系统的同学,biopython值得拥有。两种读取方法Biopython提供了两种方法读取多序......  Python开发的方向太多了,有机器学习,数据挖掘,网络开发,爬虫等等。其实在生信领域,Python还显现不出绝对的优势,生信的大部分软件流程都是用shell或Perl写的,而且已经足够好用了。我选Python是因为我想顺便学点数据挖掘和机器学习的东西,而且Python这些年越来越火,发展势头远超其他脚本语言,所以学它肯定是没错的。一、入门标准入门比较难定义,什么程度才算入门呢?掌握基本的语法,熟...
Python开发的方向太多了,有机器学习,数据挖掘,网络开发,爬虫等等。其实在生信领域,Python还显现不出绝对的优势,生信的大部分软件流程都是用shell或Perl写的,而且已经足够好用了。我选Python是因为我想顺便学点数据挖掘和机器学习的东西,而且Python这些年越来越火,发展势头远超其他脚本语言,所以学它肯定是没错的。一、入门标准入门比较难定义,什么程度才算入门呢?掌握基本的语法,熟... 