对高通量测序数据进行比对,就是将测序得到的reads定位到基因组序列上,对illumina或454得到的short reads比对的软件主要有Bowtie BWA HISAT Tophat。
SAM格式,是序列比对文件的格式。分为头部区和主体区,都以tab分列。
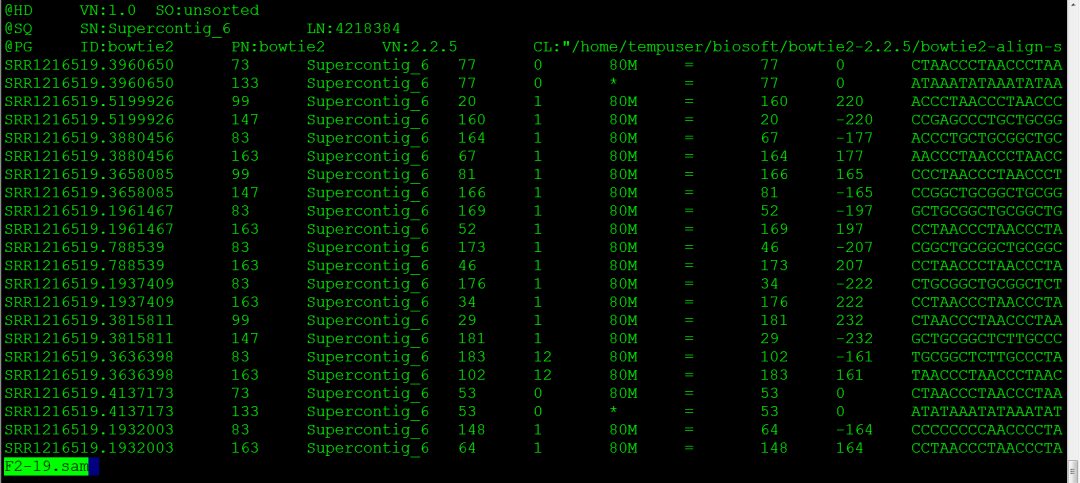
@HD VN:1.0 SO:unsorted
头部区第一行,VN是格式版本,SO是比对的类型,有unknown,unsorted,queryname,coordinate
@SQ SN:Supercontig_6 LN:4218384
参考序列名,SN是参考序列名,LN是参考序列长度
@PG ID:bowtie2 PN:bowtie2 VN:2.2.5
比对所使用的软件
1 QNAME 比对的序列名
2FLAG Bwise FLAG 表明比对类型,第二列的值回答了十一个问题,很重要,可以鉴别污染,提取数据
3RNAME 比对上的参考序列名
4 POS 比对上的最左边的定位
5 MAPQ 比对质量
6 CIGAR 比对结果信息:匹配碱基数,可变剪切等。5M1D75M前5个匹配,I插入,D缺失,后75个又匹配
7NRNM 相匹配的另外一条序列,比对上的参考序列名
8MPOS 1-BASED leftmost mate position
9ISIZE 插入片段长度
10 SEQ 和参考序列在同一个链上的比对序列(若比对结果再负义链上,则序列是其反向重复序列)
11QUAL 比对序列的质量
12可选的行
FLAG值介绍:
1该read是成对的paired reads中第一个
2paired reads中每个都正确比对到参考序列
4该reads没比对到参考序列上
8与该read成对的另一端read没比对上
16该read和参考序列相比,是反向互补的
32该read成对的另一端和参考序列相比,是反向互补的
64在paired reads中,该read是第一条
128在paired reads中,该read是第二条
256次优的比对结果
512没有通过质量控制
1024PCR重复
- 本文固定链接: https://maimengkong.com/zu/902.html
- 转载请注明: : 萌小白 2022年5月3日 于 卖萌控的博客 发表
- 百度已收录
