通常,运行完Cell Ranger得到的a.cloupe文件会得到所有细胞的默认分群结果。但是这些数据并没有进行严格的数据过滤,其中可能包含了很多细胞、死细胞或者状态不好细胞的信息。之前版本的loupe没有数据过滤后重聚类的功能,要实现这些数据的过滤我们一般会借助第三的分析软件比如seurat,现在10x Genomics公司已经对loupe进行了升级,升级到5.0之后只需要简单的几步操作就可以在loupe上实现这些功能,这对没有生信背景的老师可以说是很有必要掌握的技能。
打开loupe之后选择Categories Mode,在这个界面会有一个recluster按钮,点击这个按钮就会进入数据过滤和重聚类的界面。
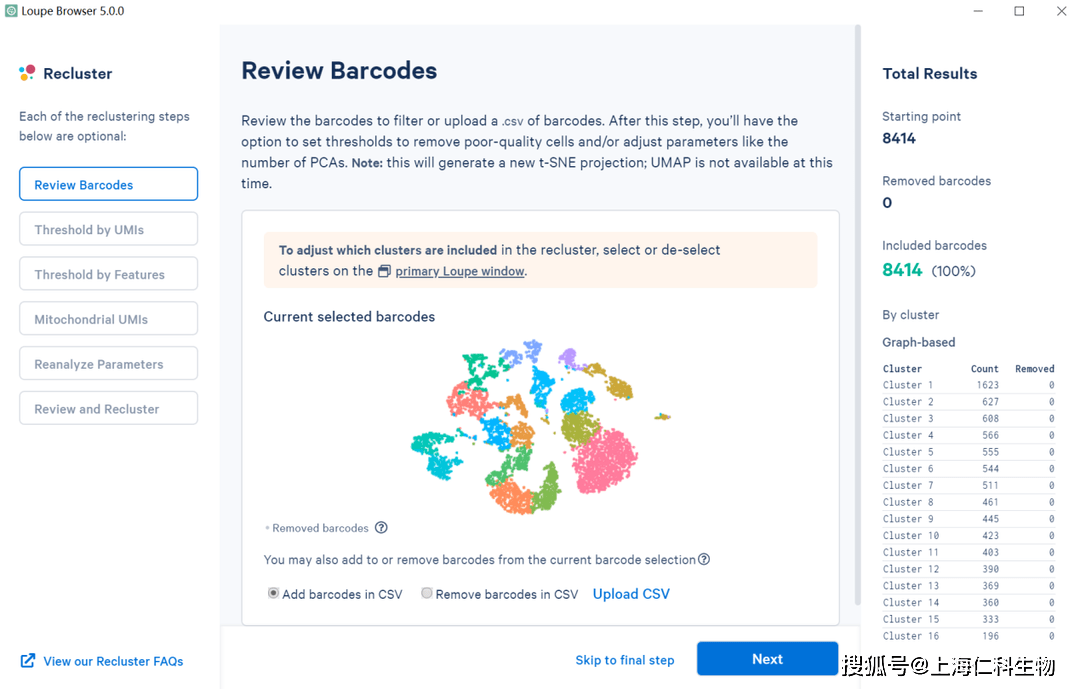
整个界面有三部分组成。最左边的显示了工作流程的当前进度一共包含了六个工作流程,可以根据需要对各个流程和步骤进行随意切换。中间是数据展示窗口,最右边是过滤后的数据统计信息,在reclustering界面的底部,有前进到下一个步骤或者直接前进到最后一步的按钮。我们下面会详细介绍一下每一步工作流程的细节和作用。
1
Review Barcode
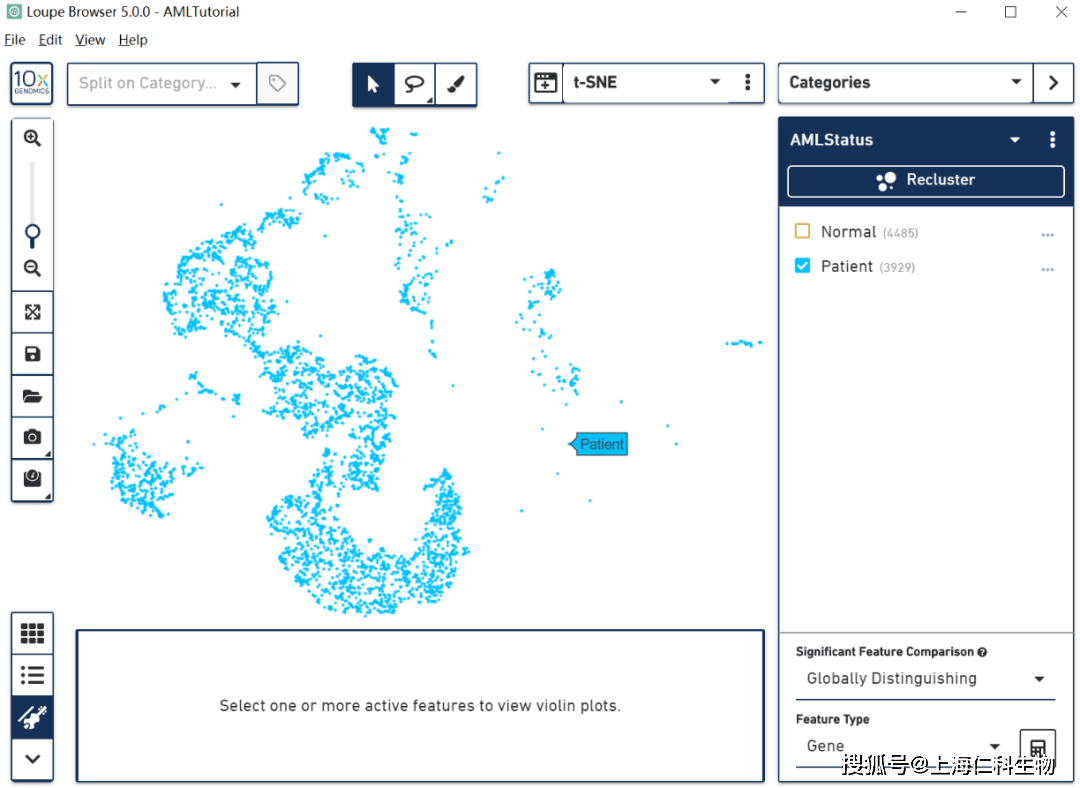
可以通过这一步提取Categories Mode中的任何一个cluster或者分类的数据。例如我们在AMLTutorial数据集中选择Categories模式下的AML status可以看到细胞分为两个部分,一个是病人组,一个是正常组。然后我们选中病人组点击recluster就可以看到我们把正常组的数据给过滤掉了。
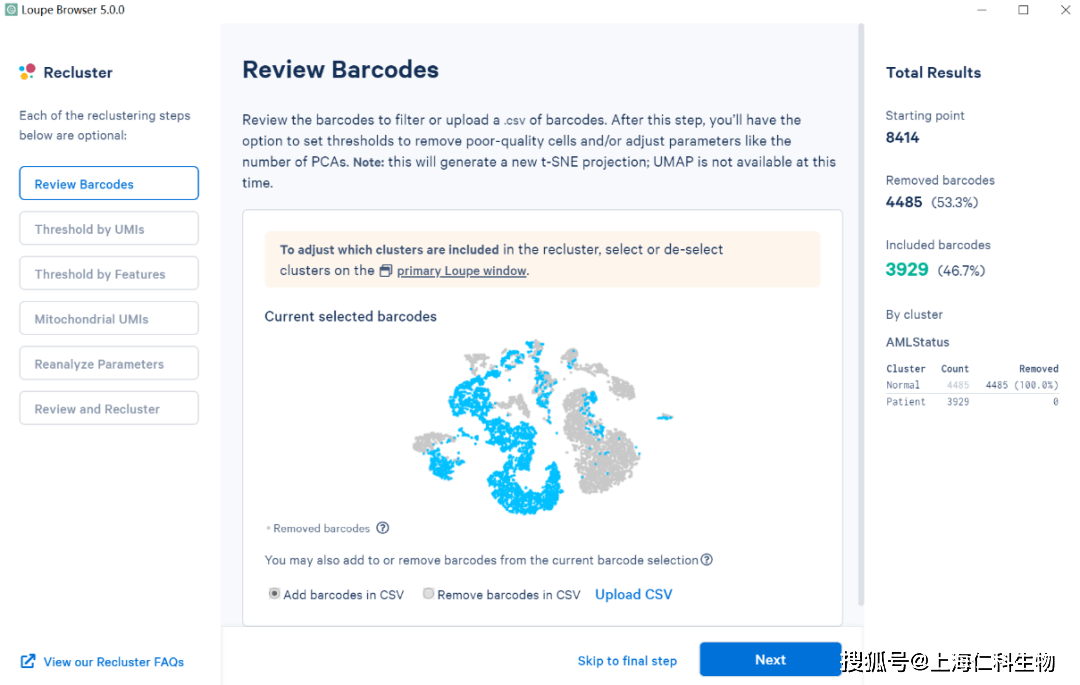
除了这种操作之外我们也可以自定义圈选一些cluster进行recluster或者通过Review Barcode界面下的upload csv按钮来自定义添加或者减少recluster的细胞类群。
2
Threshold by UMIs
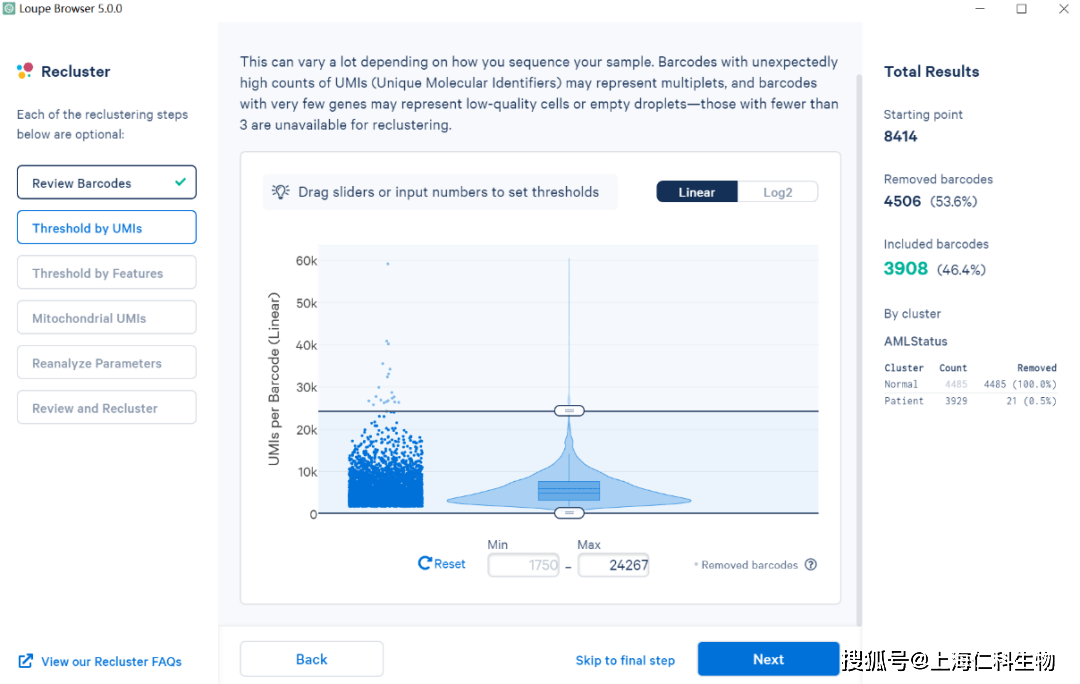
第二步是设定UMI的阈值,通过这一步的操作可以对UMI的阈值进行自定义设定,可以通过滑动图片中的阈值线或者输入阈值范围来进行操作,如下图:
3
Threshold by Features
第三步主要是对检测到的各个细胞中的基因数进行过滤也是可以通过滑动阈值线或者直接输入阈值范围进行数据过滤。通过这一步的操作就可以有效去除双细胞或者表达丰度比较低的细胞亚群。
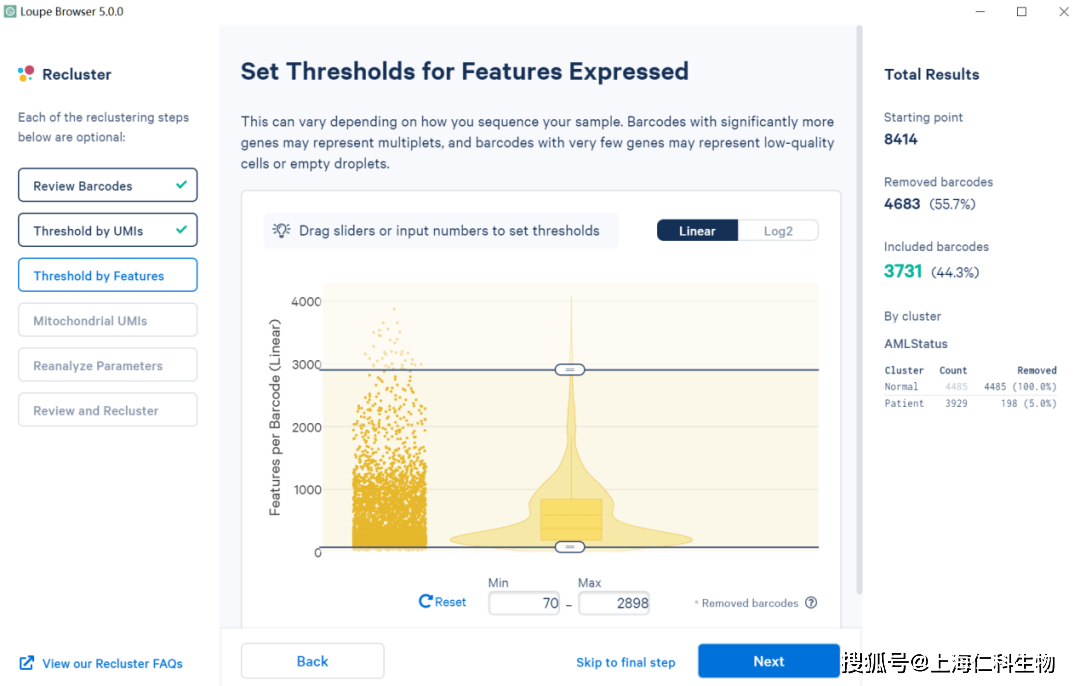
4
Mitochondrial UMIs
第四步是线粒体基因的过滤,通过这一步可以自定义设定线粒体基因的的占比的阈值,过滤掉状态比较差的细胞类群。同时这一步除了可以进行线粒体基因的过滤之外,我们也可以根据自己的研究需求导入需要过滤基因的CSV文件,从而实现其他基因的过滤。
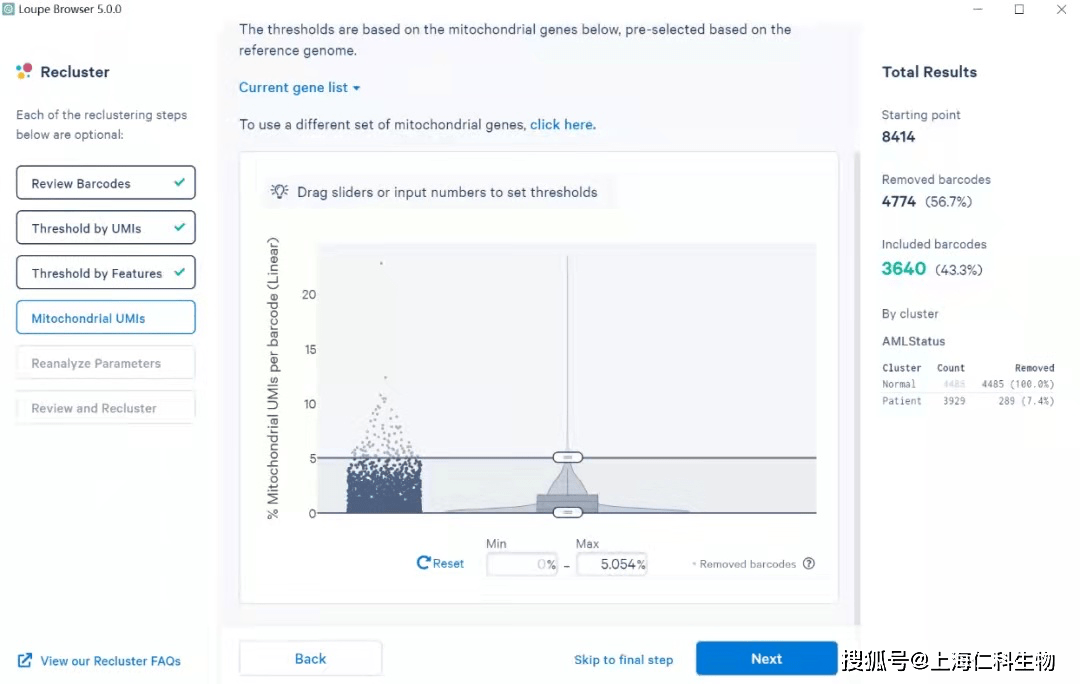
5
Reanalyze Parameters
第五步可以设定聚类的参数可以通过参数的设定将重新聚类的结果分为设定的亚群数量。
6
Review and Recluster
最后一步是对前面所有设定的参数进行数据处理,点击运行后最终实现数据的过滤和重聚类。如果你对最终的结果还是不满意,那么还可以随时回到前面的步骤对相关参数进行修改,然后重新分析聚类。- 本文固定链接: https://maimengkong.com/zu/1516.html
- 转载请注明: : 萌小白 2023年5月3日 于 卖萌控的博客 发表
- 百度已收录
