一.软件介绍
10X genomics 公司不仅为单细胞转录组数据分析提供了配套的 cell Ranger 软件,同时也提供了专门的分析结果查看软件-Loupe Cell Browser,该软件是一个图形界面的软件,操作非常的方便,支持 windows 和 mac 两种操作系统,下载地址如下:https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest
二、操作指南
安装好之后,双击启动,界面如下:

在收到的单细胞测序结果文件夹中找到后缀名为 cloupe 文件,通过左上角 File->Open File,或者箭头所指直接打开,导入后缀名 cloupe 的文件,导入成功后的页面如下:

图片默认展示的是 t-SNE 降维后的 2D-plot,细胞的颜色根据 Graph-Based 聚类结果进行填充,对于聚类得到的 cluster,可以通过对应的颜色框(右击选择 Edit color 或 Edit name)来调节是否在图中显示以及显示的颜色。通过选择 Graph-Based 右侧的三角形下拉按钮可以查看其他聚类的结果,比如 K-means 聚类的结果(下图)。
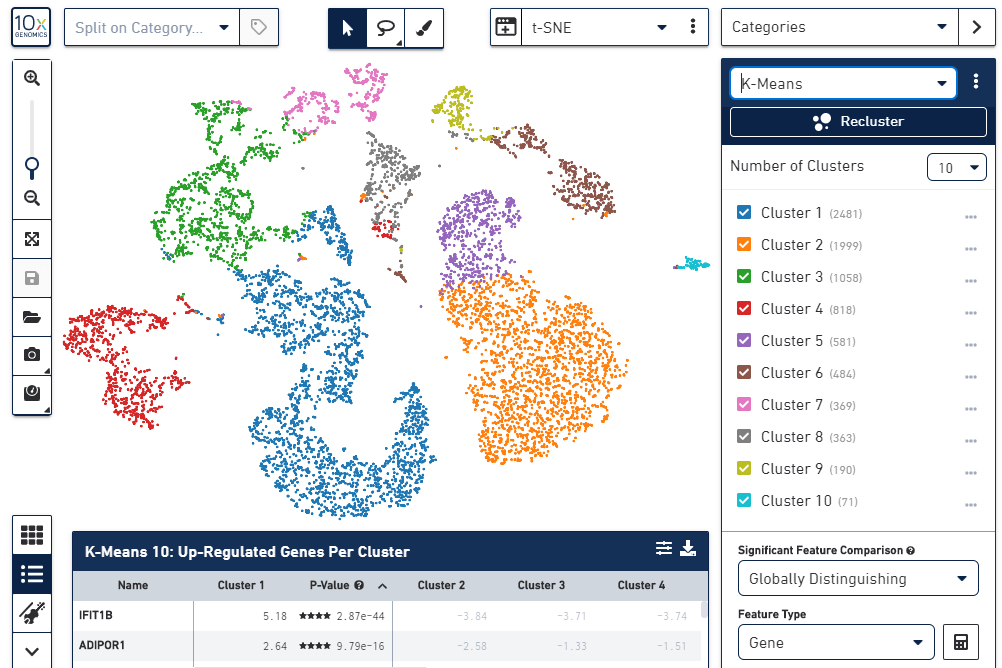
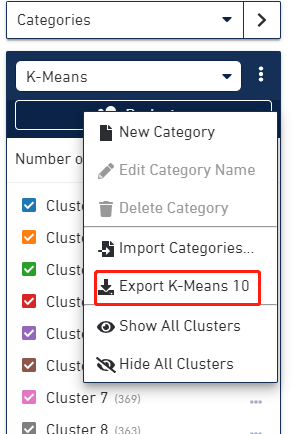
我们也可以点击右侧 Export可以导出每个细胞对应的 cluster 信息的 csv 格式文件。接下来我们把视线转移到中下方的表格,它显示的是feature table, 即每个cluster下显著差异的基因列表,示意如下:

同时也可以点击上图红色标出的部分进行选择和导出差异结果,默认导出 top50 的 genes,可按照实际需要进行选择。
在确定我们感兴趣的差异基因后,我们可以基于结果绘制每个 cluster 的 top marker 的 heatmap 图,通过点击下图红框标出的部分进行热图的展示(左侧红框标出的按钮)和导出完成热图(右侧红框标出的按钮)。

以上这些就是该软件的基本功能,相比网页的显示结果,该软件交互式更强,可以更加方便的探究数据,具体每个按钮的功能注释详见下图。

- 本文固定链接: https://maimengkong.com/zu/1517.html
- 转载请注明: : 萌小白 2023年5月3日 于 卖萌控的博客 发表
- 百度已收录
