超越转录组:单细胞表观基因组学的价值?
长期以来,研究人员一直在基于转录组数据来探索有关复杂细胞群体和过程的生物学见解。样本本身的异质性以及基因表达的细胞间差异促使科学家从批量(bulk)细胞的平均值测定转向单细胞RNA测序,以便更准确地描述复杂的生物系统。然而,单单凭借转录组的信息,也许无法讲述细胞的完整故事,科学家通常还想了解造成这些基因表达差异背后的原因 – 也就是说,从机理上解释通过转录分析观察到的基因表达模式变化。
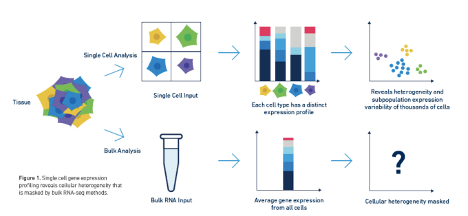
单细胞分析揭示批量细胞分析所掩盖的细胞异质性
转录组在本质上其实是高度协调的基因表达程序的副产物。该程序受到表观基因组、DNA和组蛋白修饰的调控,这些因素通过影响转录因子等DNA结合蛋白与基因组DNA的相互作用,从而激活或抑制基因表达。表观基因组调控增加了生物学的复杂性,不仅驱动了令人着迷的发育相关事件,让有着相同DNA的细胞具有不同的身份并形成多种类型的组织和器官,同时也推动了细胞的异质性,从而促进了复杂的疾病生物学。了解这种调控如何在单细胞中发挥作用,有望揭示不同细胞间转录差异背后的深层次的生物学复杂机制。
也许您是一名癌症研究人员,正在试图定义促进和维持癌症干细胞的调控网络。或者,您准备在免疫检查点阻断疗法后鉴定调控元件在T细胞活化和耗竭中的作用。也许治疗干预对某一特定癌症始终无效,您需要研究和完善细胞类型和细胞状态的注释,以确定肿瘤内部的异质性,甚至在考虑是否是一个被掩盖的细胞亚型在驱动耐药、转移和复发。无论您身处哪个领域,如果您想剖析一个复杂的生物系统并了解单个细胞在该系统中如何受到功能调控,那么单细胞ATAC-seq(scATAC-seq)也许就是您的理想解决方案。
从开放染色质到深入了解基因调控
染色质转座酶可接近性分析(ATAC)是一种通过鉴定开放染色质区域来研究基因组物理结构的方法。该技术使用了一种高活性的转座酶,该酶可以结合在开放染色质区域,在对该区域进行切割的同时插入测序接头,通过该方法获得的测序文库包含了开放染色质区域所富含的序列。
10x Genomics单细胞ATAC-seq工作原理
从开放染色质中可以获得什么信息?
染色质结构的动态重塑是影响哪些基因会发生转录以及何时转录的主要机制之一。当染色质开放时,DNA结合蛋白可以接近调控序列,从而实现转录。scATAC-seq能够提供染色质可接近性的信息,并揭示单个细胞中基因转录活跃的区域。但是,它的功能并不止于此。数十万个调控元件在不同背景下协同作用以协调基因表达模式,而scATAC-seq能够提供这些不同元件的信息,包括其细胞类型特异性、结合位点基序,以及转录因子之类的低表达基因是否有可能开启。
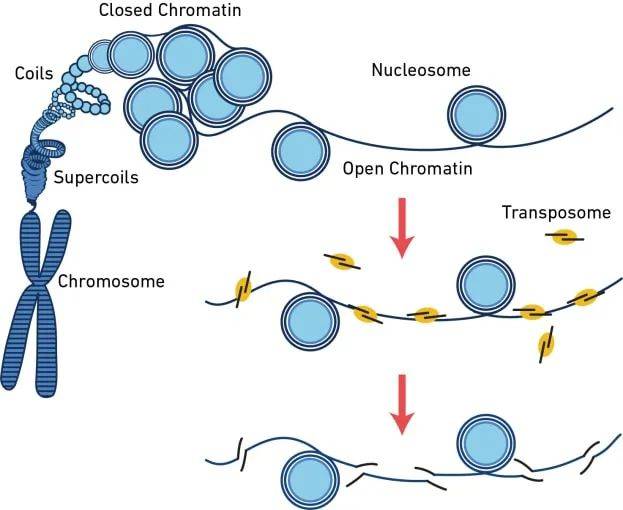
染色质开放区域与基因转录的活跃区域相关。染色质转座酶可接近性分析(ATAC)可以特异性生成染色质开放区域的DNA短片段。将这些剪切位点映射回基因组可以给我们提供一个窗口,以便了解转录因子基序结合、启动子和增强子区域,以及常染色质和异染色质区域。
通过ATAC-seq来定义细胞类型和状态
与单细胞RNA-seq一样,单细胞ATAC-seq也可以对相似的细胞类型和状态进行鉴定和聚类。不过,scATAC-seq数据所用的细胞类型注释方法略有不同。使用scATAC-seq进行细胞注释的最简单的方法是将开放启动子区域作为转录活性的信号。
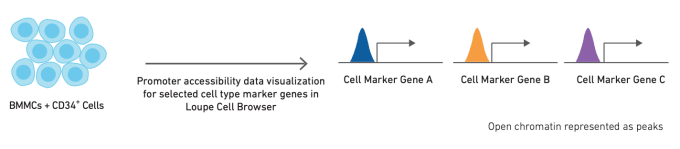
利用细胞类型特异性的顺式调控元件进行注释
此外也可以利用细胞类型特异性的特征集合对细胞类型进行注释,这些集合是根据已分选细胞子集的批量细胞ATAC数据得出的。最后,如果有相同或相似样本的单细胞基因表达数据,那么scRNA-seq数据的注释可作为完善scATAC-seq聚类注释的参考。这些不同策略的相对表现,以及如何使用它们的详细信息,都可以在这篇技术指南中找到(1)。
scATAC-seq提供了与单细胞基因表达数据互补的信息,在某些情况下,甚至可以提供更高的细胞状态分辨率。在对免疫检查点调节剂VISTA的深入分析中,研究人员结合单细胞基因表达、T细胞受体(TCR)图谱分析和单细胞ATAC分析,发现VISTA的缺失会导致记忆样T细胞的扩增,并以静息细胞为代价。此外,ATAC-seq的数据表明,记忆样T细胞簇中TCR效应基因的可接近性增加,说明这些细胞已启动对TCR的应答。欢迎查阅这篇《单细胞测序深化免疫肿瘤学研究》,了解scRNA-seq和scATAC-seq联合使用如何提供强大的多组学分辨率,以帮助研究免疫检查点调控。
推断发育轨迹和鉴定调控元件
scATAC-seq还能够对发育轨迹进行计算推断。染色质可接近性的改变是细胞分化和发育的主要驱动特征。为了改变细胞状态(或细胞类型),细胞必须首先准备好改变其转录程序,而scATAC-seq提供了这些准备过程的读数。发育轨迹的鉴定是基于这样一个事实,即转录程序相似的细胞可能是相关的,开放染色质图谱相似的细胞可以被分在一组。
研究人员可以根据这些关系以数字序列来编排单个细胞,从而确定分化过程中细胞的时间顺序。为了展示如何推断发育轨迹,10x Genomics的科学家对近20,000个骨髓单核细胞和CD34+分选细胞开展了scATAC-seq。这篇中文的应用指南(2)介绍了他们如何推断从干细胞到B细胞、单核细胞和红系细胞的发育轨迹。有了发育轨迹,scATAC-seq数据可帮您实现更进一步的研究,确定不同分化状态下变化并可能决定分化状态的转录因子基序和增强子。
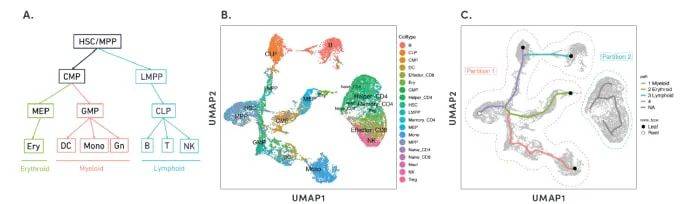
多个造血谱系的发育轨迹。A. 造血过程的细胞等级。B. 人骨髓单核细胞的UMAP投影。C. 细胞分区1组的细胞分区和推断轨迹路径。HSC:造血干细胞;MPP:多能祖细胞;CMP:共同髓系祖细胞;CLP:共同淋巴祖细胞;GMP:粒细胞-巨噬细胞祖细胞;Mono:单核细胞;DC:树突状细胞;Gn:粒细胞;LMPP:淋巴-髓系祖细胞;MEP:巨核细胞-红系祖细胞;Ery:红细胞;B:B细胞;T:T细胞;NK:自然杀伤细胞;
斯坦福大学的研究人员发布了一个重要的应用案例,在其混合表型急性白血病(MPAL)研究中证明了利用单细胞ATAC数据构建发育轨迹的能力。这种癌症是由代表多个造血谱系的细胞组成的。此外,患者样本中的癌细胞显示了与淋巴样细胞和髓样细胞都相关的标志物基因,这导致它们的细胞类型含糊不清。为了弄清这些癌细胞的真正细胞表型和发育起源,研究人员利用寡核苷酸偶联抗体来研究表面蛋白,借助scRNA-seq来研究基因表达,并通过scATAC-seq来研究染色质可接近性,以此绘制出健康人体血液的发育轨迹参考图。然后,他们将癌细胞样MPAL细胞的表型投影到该参考图中,首先将其分为“健康样”或“疾病样”,然后按照最相关的发育状态进一步分类,包括祖细胞样、红细胞样、淋巴细胞样、髓细胞样和T/NK细胞样(3)。

探索基因调控网络
有了绘制开放染色质区域的能力,研究人员还能开始了解细胞内的信息流。转录因子在哪里结合以及它们开启或关闭哪些基因,这取决于DNA上是否存在结合位点,以及该结合位点是否可接近。因此,通过scRNA-seq揭示或通过scATAC-seq推断的转录因子基因活性,以及只能通过表观遗传学方法辨别的靶基因结合位点的可接近性,都对细胞类型特异性的网络有着重要贡献。例如,为了让转录因子1(TF1)调控特定细胞中的TF2,该细胞中的TF1基因必须具有活性,且TF2基因上的TF1结合位点必须可以接近。
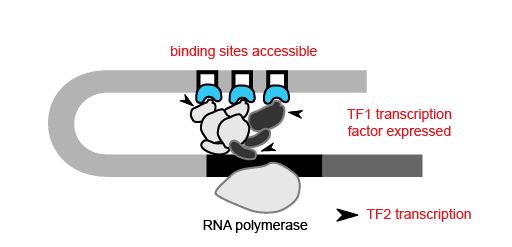
建立基因调控网络。TF2的转录需要TF1(蓝色)的表达,且TF2上的TF1结合位点可以接近。来源:改编自Hupé et al., 2012 (CC BY 3.0)
通过scATAC-seq数据,研究人员可以根据开放染色质上顺式调控元件的基因活性评分来推断特定细胞中转录因子的表达。推定的转录因子结合位点的可接近性可作为转录因子表达数据的补充,以建立细胞类型特异性的调控网络。斯坦福大学的研究人员在上文提到的混合表型急性白血病研究中,利用这种方法及其scATAC-seq数据,将启动子可接近性与靶基因表达相关联,从而确定了调控白血病基因的疾病特异性网络(3)。重要的是,各个细胞谱系和发育状态的转录因子活性以及更广泛的调控网络都可以追踪,而这在单独使用scRNA-seq时颇具挑战性。
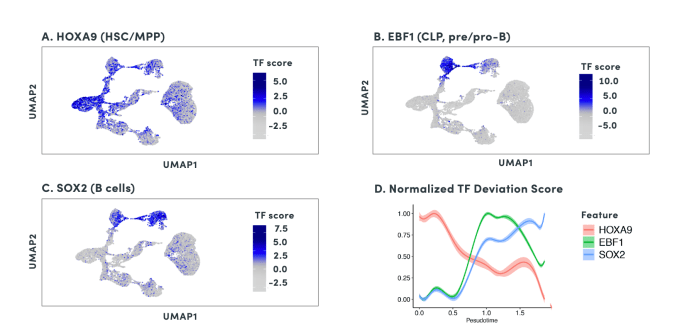
淋巴谱系轨迹中的转录因子动态。A. HSC和MPP富含HOXA9活性。B. CLP和B细胞祖细胞富含EBF1活性。C. B细胞富含SOX2活性。D. 细胞类型特异性的转录因子HOXA9、EBF1和 SOX2分别在淋巴谱系轨迹的早期、中期和晚期被激活。图中所示曲线是拟合到ChromVAR偏离分数的平滑样条,该分数由拟时间维度中的min-max归一化。
若想详细了解如何利用scATAC-seq构建基因调控网络,请参阅这篇应用指南(2)。
单细胞ATAC-seq入门
起初,ATAC-seq提供的这种表观遗传数据似乎令人望而却步。您将直接查看DNA序列片段,而不是对转录本进行计数。然而,Loupe Browser(4)等工具可帮助您以单细胞分辨率获得染色质可接近性的整体视图,而从中获得的见解将加深您对复杂系统的生物学理解,从仅仅知道基因已经打开到了解其如何被激活。
您还在寻找ATAC-seq的基本介绍?下面这个介绍Chromium单细胞ATAC解决方案的网络研讨会可带领您入门。您也可以点击阅读原文访问我们的产品页面,更详细地了解ATAC-seq。
参考资料
- https://support.10xgenomics.com/single-cell-atac/index/doc/technical-note-cell-type-annotation-strategies-for-single-cell-atac-seq-data
- https://www.10xgenomics.com/cn/resources/application-notes/deciphering-epigenetic-regulation-with-single-cell-atac-seq/
- J Granja et al., Single-cell multiomic analysis identifies regulatory programs in mixed-phenotype acute leukemia. Nat Biotechnol. 37, 12 (2019).
- https://support.10xgenomics.com/single-cell-atac/software/visualization/latest/tutorial
- 本文固定链接: https://maimengkong.com/zu/1518.html
- 转载请注明: : 萌小白 2023年5月3日 于 卖萌控的博客 发表
- 百度已收录
