SLAM-seq(thiol(SH)-linked Alkylation for the Metabolic sequencing of RNA)可以理解为“S4U烷基化RNA代谢测序技术”,通过核苷类似物(S4U) 标记和高通量测序方法,实现对新生RNA与原有RNA的定量和识别。SLAM-seq技术有助于检测RNA的半衰期与稳定性变化,鉴定RNA修饰所介导的靶基因变化,以及揭示mRNA稳定性的决定性因素。SLAM-seq是研究基因动态表达的首选,云序生物提供SLAM-seq测序服务,助力客户深入研究基于转录调控的相关机制。
SLAM-seq技术原理
SLAM-seq技术是在培养的细胞中加入4-thiouridine (S4U)核酸类似物,转录时S4U可以与DNA序列上的A碱基互补配对,从而进入新合成的RNA链中。抽提总RNA,加入碘乙酰胺(IAA)使其发生化学修饰。在逆转录过程中发生修饰的S4U与G碱基发生错配生成cDNA单链,导致最终检测结果里原本的T变成了C。通过分析检测 T→C 的转变比例,即可得到新生RNA的信息。
实验流程简述如下:
1)S4U标记:培养的细胞中加入S4U,得到带有S4U标记的新生RNA;
2)加化学修饰:抽提纯化RNA,并加入碘乙酰胺(IAA)诱导S4U发生化学修饰;
3)构建文库:逆转录时S4U与G发生错配,后续PCR中G又与C配对;
4)上机测序:最终测序结果中本来该为T的位点被识别为C;
5)生物信息学分析:检测 T→C 的转变比例,即可得到新生RNA。
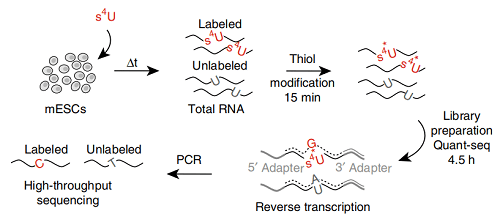 SLAM-seq测序流程图
SLAM-seq测序流程图
云序技术优势
优势1:操作简单
对于常规的新生RNA检测技术,一般需要复杂的标准化方案或者加入spike-in,而SLAM-seq无需繁琐且技术要求苛刻的免疫共沉淀或生物素分离等操作,操作简单。
优势2:RNA用量少
后续分析中只需要检测 T→C 转变比例,即可知道新合成的RNA情况,大大减少了RNA的用量。
优势3:灵敏度高
SLAM-seq可以通过区分已有的RNAs以及新合成的RNAs来扣除背景信号,对新合成的RNAs进行差异分析,提高基因差异表达检测的灵敏度。
优势4:应用方向广
SLAM-seq技术除了可对新合成的转录本进行定量,还有助于检测RNA的半衰期与稳定性变化,评估增强子的活性,鉴定RNA修饰所介导的靶基因变化,以及揭示mRNA稳定性的决定性因素等。
优势5:一站式服务
客户只需提供细胞,云序生物为您完成从样品准备,文库制备,上机测序到数据分析的整套服务流程。
优势6:专业的生物信息学分析
云序生物拥有强大的生物信息学团队,能够满足客户的各类深入数据分析需求。
样本要求
1.样品类型:细胞
2.样品量:
细胞:2×106
3.样品处理:
在细胞培养时,将S4U(100μM)核酸类似物掺入培养基内,培养24 h(文献推荐,具体可调整),倒掉培养基。用PBS清洗2次后,加入TRIzol或RNA保护剂处理,转移至1.5ml离心管,封口膜封好,液氮冻存后-80℃保存(注意已保存的样品不要反复冻融)。
4.样品运输:
如需运输请使用干冰包裹。
案例解析
案例1:SLAm-seq可对转录本稳定性进行检测
题目:Thiol-linked alkylation of RNA to assess expression dynamics
期刊:Nature Methods
高通量测序的基因表达谱揭示了稳定状态下RNA种类的定性和定量变化,但模糊了RNA转录、加工和衰变的细胞内动力学。本研究中作者开发了用于RNA代谢测序的SLAM-seq,SLAM-seq能够快速获取总RNA背景下RNA聚合酶II依赖的基因表达动态。在小鼠胚胎干细胞中验证了该方法,为了直接测量mRNA转录的稳定性,将mESCs进行S4U代谢RNA标记24小时,并在各个时间点制备总RNA。然后对总RNA进行烷基化和定量测序。最终通过mRNA的半衰期测量结果显示,在mESCs中,mRNA的稳定性与mRNA的半衰期测量结果总体上具有良好的相关性。
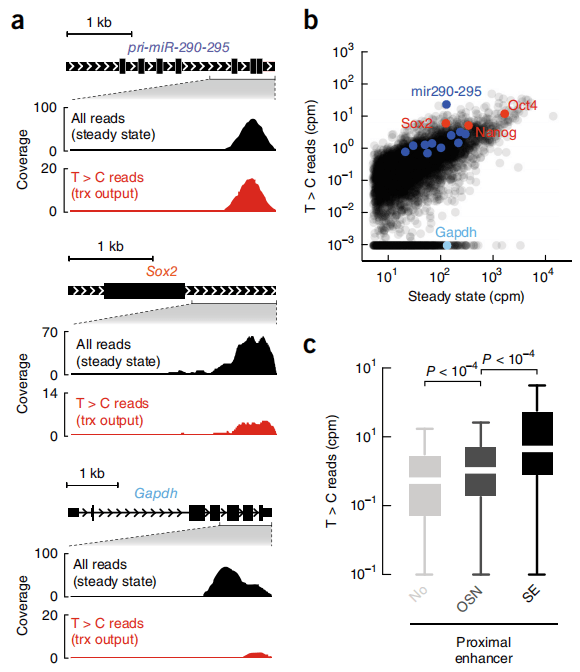 图1:mESCs中多聚腺苷酸转录本的丰度水平
图1:mESCs中多聚腺苷酸转录本的丰度水平
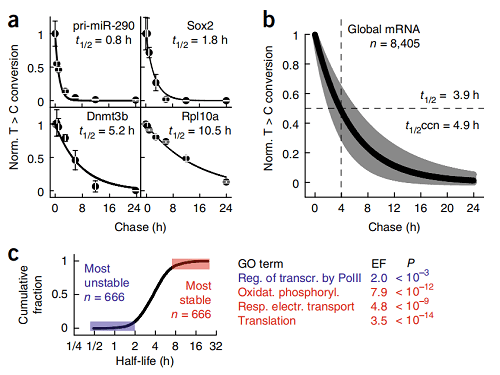 图2:mESCs中全局和转录特异性mRNA的稳定性
图2:mESCs中全局和转录特异性mRNA的稳定性
- 本文固定链接: https://maimengkong.com/zu/1887.html
- 转载请注明: : 萌小白 2025年1月10日 于 卖萌控的博客 发表
- 百度已收录
