1.非编码RNA简介
非编码RNA是一类被认为不具备编码能力RNA,目前已知的已经有十多种,主要包括了:小RNA(sRNA) <40nt、小干扰RNA、miRNA(18-24nt)、长非编码lncRNA(>200nt)、核糖体RNA、核小RNA、核仁小RNA等等。随着人们认知的逐渐深入,发现这些非编码RNA在转录调控上发挥着重要的作用,并且其他部分同样具有编码蛋白的功能。
1.1 lncRNA结构
在非编码RNA中,有一类长度超过200 nt的RNA分子,被称为长链非编码RNA。非编码RNA的来源包括基因间转录本、增强子RNA、正义/反义转录本等。研究发现一些lncRNA与mRNA非常相似,同样具有5'帽子结构和polyA尾巴,区别是不具有开放读码框(open reading frame, ORF)。在lncRNA的形成过程中,lncRNA的转录由RNA聚合酶II/III所引导,并且大多数lncRNA和mRNA一样具有polyA尾巴。然而,某些lncRNA有着不同的加工修饰途径,其加工过程与tRNA形成过程较为相似,其初始转录本的3'端同样会经过RNase P的切割,形成一段类似于tRNA的mascRNA (MALAT1-associated small cytoplasmic RNA)。
1.2 lncRNA功能与特征
lncRNA最重要的生物学功能之一就是可以与特定蛋白相结合,进而发挥相应的作用。有些支架蛋白或RBP在与作为骨架的lncRNA结合后,促使大分子复合物的形成和激活,能够进一步激活下游通路或直接发挥作用。一部分lncRNA可通过与蛋白结合顺式地作用于某些基因的转录起始位点,进而调节基因转录。lncRNA也可以直接与DNA序列结合,从而形成RNA-DNA三股螺旋复合物,使得染色质构象处于开放状态,促进基因的转录。其他lncRNA则具有辅助抑制因子的功能,可与特定转录因子结合,阻碍其与DNA的结合,而抑制RNA聚合酶的活性。此外,lncRNA在染色质结构重建和三维结构中也起着重要作用,可通过与染色质修饰复合物或增强子元件结合,促进激活子活性。此外,反义lncRNA与相重叠的蛋白编码基因可以相互抑制其基因的表达。近年来,有研究表明lncRNA也是一种重要的内源性竞争抑制ncRNA,可作为miRNA的抑制因子,调节其miRNA对靶基因的负向调控作用。到目前为止,已经有大量的lncRNA-miRNA-mRNA作用通路被揭示并研究了其在各种疾病或者发育进程中的作用。尽管大多数lncRNA不具有编码能力,仍然有研究或预测分析发现有少部分lncRNA具有编码能力,可以翻译形成小分子量的蛋白质。
2. lncRNA鉴定
lncRNA预测软件主要有2种方法,一种是直接预测其是否含有ORF元件或同源预测的方法;另一种是基于机器学习的方法,常见的方法包括:线性回归算法、支持向量机(SVM)、或者随机森林、深度学习等。目前主流的方法是采用机器学习算法鉴定lncRNA。
2.1 机器学习鉴定lncRNA
从是否有参考来看可以分为有参类和无参类。
2.1.1 有参考的鉴定策略
CPC是典型的有参类鉴定软件,它基于SVM算法设计,被广泛使用在lncRNA的鉴定上,是目前公司的主要鉴定方法。然而CPC预测lncRNA并没有想象中的这么美好。首先,CPC 本身就不为 lncRNA 鉴定而设计的,它可以根据 BLASTX 的开放阅读框信息和比对结果预测lncRNA ,lncRNA不如mRNA 保守,高比例的 lncRNA 表现出许多类似于蛋白质编码序列的特征 。这会使流程错误地将lncRNA 归类为mRNA。其次,CPC 需要一个高质量且相当全面的数据库,但许多物种的注释信息并不完整,甚至缺少有效的参考基因组。其次,CPC 严重依赖 BLASTX 的输出,但多序列比对工具不能保证最佳比对。最后,极其耗时的比对过程使得在海量数据上使用CPC耗时耗力,需要高昂的运算条件和资源。
2.1.2 无参考的鉴定策略
基于CPC的尴尬使用范围,和不算优秀的可靠性,人们又开发了一系列无参类软件,如CPC2, CNCI, lncRNA MFDL, lncScore, LncADeep, DeepLNC, LncRNAnet, COME, CPAT, lncRScan-SVM, longdist, PLEK, FEElnc, 和LncFinder等。
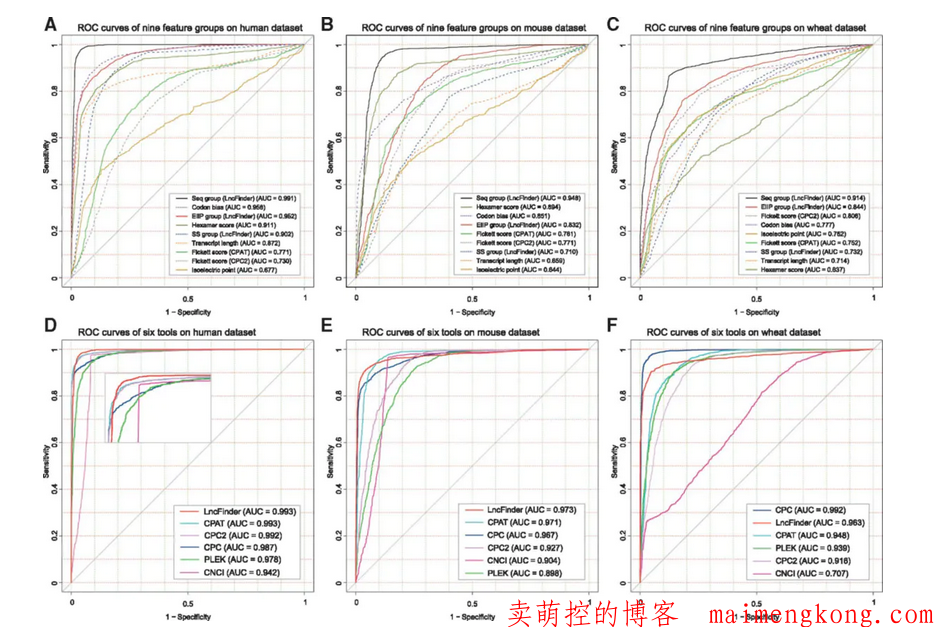
就LncFinder的作者对同类的深度学习软件进行对比,结果如下图所示:
链接:https://www.jianshu.com/p/5cd5555270b8
来源:简书
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。
- 本文固定链接: https://maimengkong.com/zu/1116.html
- 转载请注明: : 萌小白 2022年7月10日 于 卖萌控的博客 发表
- 百度已收录
