各位芝士的朋友好,今天我们继续聊我们的SNP话题,前面两讲我们分享了SNP发生的位置,发生的类型以及SNP的命名,并且特意提到了SNP的两个数据库,今天我们来学习一下这两个数据库的使用。
dbSNP
dbSNP 全称为The Single Nucleotide Polymorphism Database,即单核苷酸多态性数据库,意思是“DNA序列中的单一碱基对(base pair)变异”,也就是DNA序列中A、T、C、G的改变,即基因组的一个特异和定位的位点出现两个或多个的核苷酸可能性,它是人类可遗传的变异中最常见的一种。该数据库是由NCBI与人类基因组研究所(National Human Genome Research Institute)合作建立的,收录了SNP、短插入缺失多态性、微卫星标记和短重复序列等数据,以及其来源、检测和验证方法、基因型信息、上下游序列、人群频率等信息。
dbSNP 网址:https://www.ncbi.nlm.nih.gov/snp/
在第二节我们讲过dbsnp数据库中的snp名字,主要是以rs开头的,这里以rs9923231为例,我们在NCBI的SNP网站上可以轻松查到(https://www.ncbi.nlm.nih.gov/snp/),如下图:
1.在search中输入rs9923231,便进入下面的界面
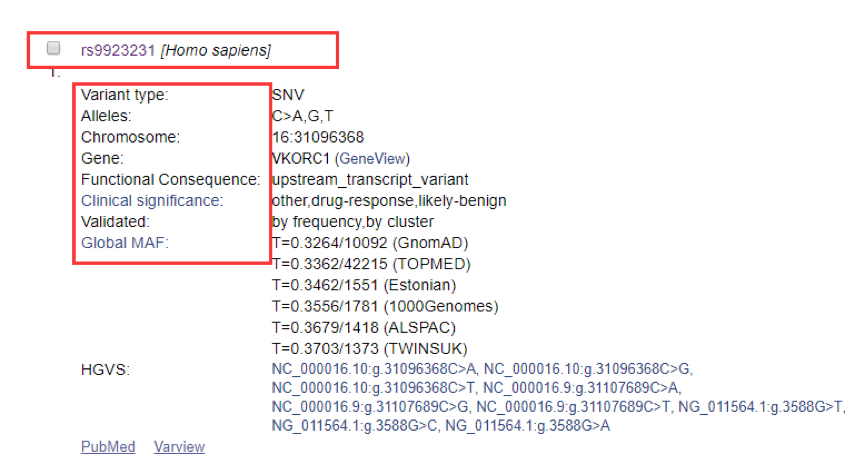
主要有下列信息:分别是Variant type(变异类型)、Alleles(等位基因)、Chromosome (染色体位置)、Gene (位于基因的名字)、Functional Consequence(功能结果)、Clinical significance (临床价值)、Validated(验证类型)、Global MAF(MAF格式文件注释)、HGVS:(HGVS数据库注释)
2.继续点击rs9923231,便出现下面的界面
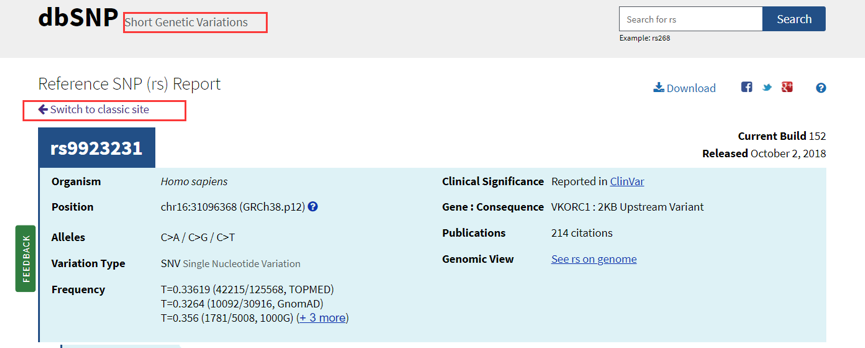
你会发现跳入到新的界面,便是对该位点的详细介绍,这个时候看到一个Switch to class site界面,点击进去发现进入到的是经典的站点,如下:
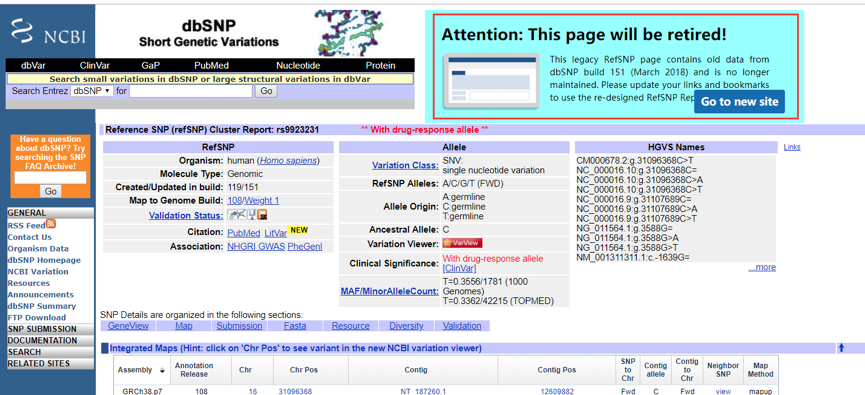
这个提示我们该站点将会停止使用,并推荐我们进入新站点,即我们最开始看到的,那我们就在新站点学习一下该网站使用。
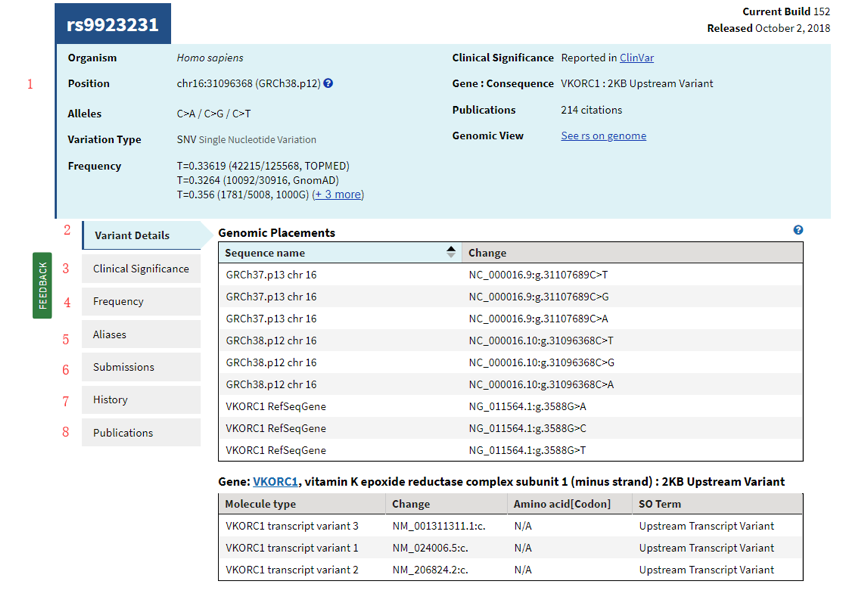
其主要是1-8个部分,1是该snp的基本信息,包括物种,位置,等位基因,变异类型,临床反应等基本信息,2是突变的具体信息,3是该位点的临床显著性。
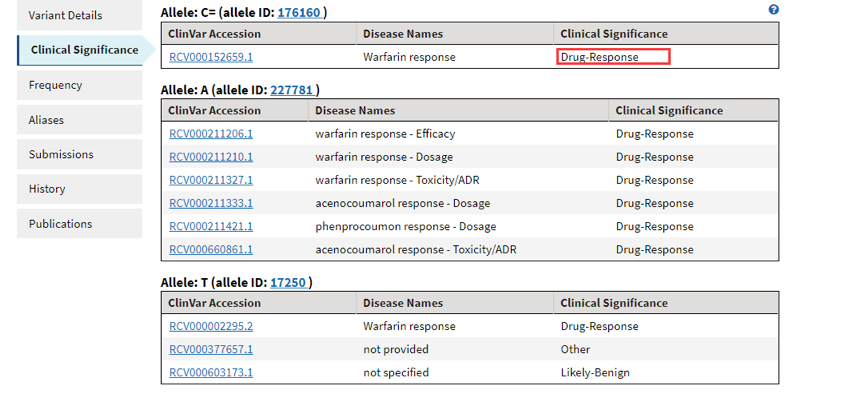
我们可以发现改为点具有临床显著性,即药物依赖性。4.是突变频率,5是等位基因,6是提交到数据库的信息,7.该位点的历史信息8.该位点发表的文章
最后一部分则是对snp位点基因组可视化。
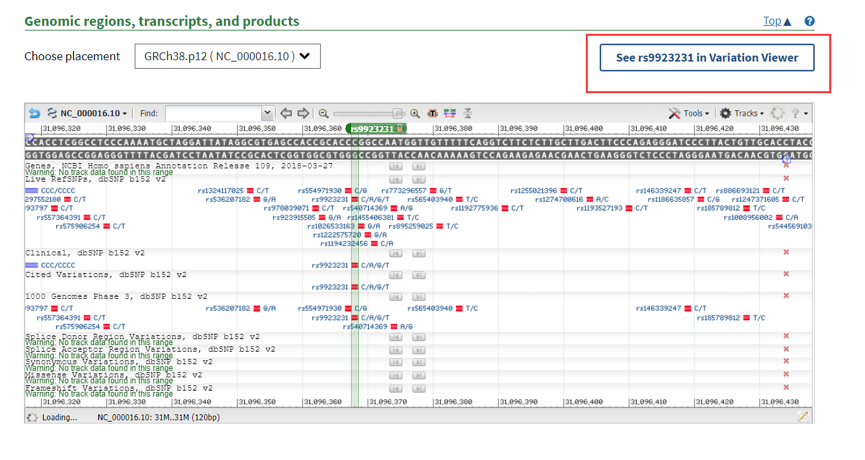
点击红框框信息,如下:
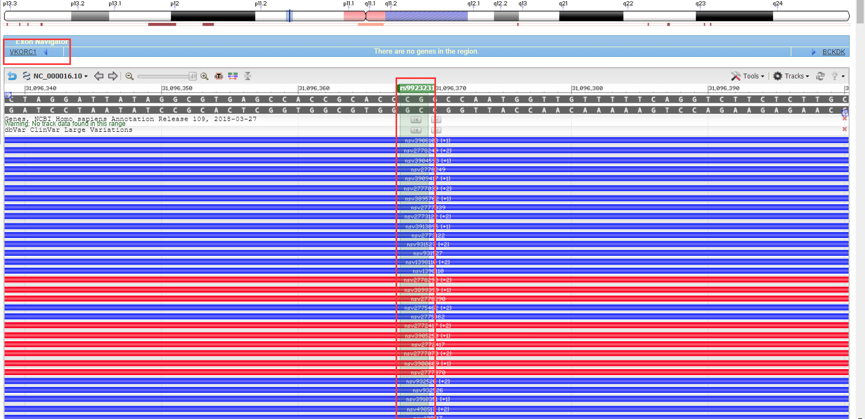
红色框框即是我们的突变位点所在的位置
下面我们来学习一下另外一个数据库HGVS的使用。
HGVS
HGVS 全称为Human Genome Variation Society(人类基因组变异协会),是一个非政府的民间学术组织,其命名规则由HGVS(the Human Genome Variation)、HVP(the Human Variome Project)、HUGO(the Human Genome Organizaion)共同制定。
网站的网址:http://www.hgvs.org/

目前基因检测行业普遍应用HGVS规则对变异进行命名,统一的命名规则方便了各种交流。对于HGVS,其命名规则是我们重点需要关注的,下面我们主要介绍HGVS的命名规则。
HGVS的所有变异命名从三个水平描述:DNA水平、RNA水平、氨基酸水平,并从两个方面进行反映:变异位点的位置和对编码蛋白造成的影响。一般情况下,变异描述的顺序为:参考序列、变异位置、变异类型。
参考序列:
1. 格式
参考序列必须是是NCBI或EBI数据库中的ID,必须同时包含accession和version信息,如NC_000023.10, NC_000023代表编号,10代表版本号。下划线前面的大写字母NC代表参考序列的格式,目前的参考序列格式有:NC_#(例如NC_000023.10),LRG_#(例如LRG_199,LRG_199t1),NG_#(例如NG_012232.1),NM_#(例如NM_004006.2),NR_#(例如NR_002196.1)和NP_#(例如NP_003997 0.1)。
NC_#:代表完整的基因组序列,标记的类别包括基因组、染色体、细胞器、质粒。
LRG_#:Locus Reference Genomic,基因座参考基因组序列。
NG_#:不完整的基因组区域,提供NCBI基因组注释途径。比较有代表性有不转录的假基因或者那些很难自行化注释的基因组簇。
NM_#:转录产物序列;成熟mRNA转录本序列。基因检测报告中最常用此作为参考序列。
NR_#:非编码的转录子序列。
NP_#:蛋白产物;主要是全长氨基酸序列,但也有一些只有部分蛋白质的部分氨基酸序列。
2.文件标识符
参考序列文件标识符应包含入藏号(accession number)和版本号(version number),两者用“.”隔开,除了LRG格式的参考序列之外,其他格式的参考序列均需要版本号。
如:NG_012232 .1
LRG参考序列不包含版本号(例如LRG_199)
3.在参考序列文件标识符和变异位置之间用冒号“:”隔开
如NC_000011.9 : g.12345611G>A。
HGVS建议使用最新的基因组参考序列版本,如NC_000023.10(对于人染色体X,GRCH37 / hg19)。
4. 参考序列类型
DNA
g. = 线性基因组参考DNA序列
o. = 环状基因组参考DNA序列
m. = 线粒体参考序列
c. = 编码DNA参考序列
n. = 非编码DNA参考序列
RNA
r. = RNA参考序列
Protein
p. = 蛋白参考序列
5. 变异位置
g代表基因组,m代表线粒体, p代表蛋白质,这三种参考序列在定位时,都是从1开始计数,写法为g.1, m.1, p.1, 除此之外,不需要任何的修饰符号。
c代表编码蛋白的DNA序列,从起始密码子的第一个碱基开始计数,写法为c.1, 只对exon区间进行计数,终点为终止密码子的最后一个碱基。
一个典型的HGVS命名示例如下:
NC_000023.9:g.32317682G>A
NC_000023.9是NCBI中人类的X染色体的编号,在参考序列之后紧跟着一个冒号,用于分隔参考序列和突变信息,g代表基因组序列,g.32317682代表在基因组上的位置,G>A表示由G碱基突变成A碱基。
如果突变位点在NCBI和EBI中没有合适的参考序列,最终的解决方案就是申请一个LRG编号(http://www.lrg-sequence.org/),在该数据库中对于HGNC定义的gene symbol也出给了对应的LRG编号。
OK,今天就先和大家分享到这,下期再见。
转自:百味科研芝士,Focus科研人的百味需求。
- 本文固定链接: https://maimengkong.com/learn/802.html
- 转载请注明: : 萌小白 2021年11月13日 于 卖萌控的博客 发表
- 百度已收录
