蛋白质是生命活动的基本单位,我们在进行遗传病基因突变为致病性研究时,很重要的一点就是分析该变异是否会影响蛋白质的结构与功能。
熟悉我们检测报告的老师都知道,康旭的蛋白损伤预测采用的是Mutation Taster、SIFT、PolyPhen。之前我们介绍了PolyPhen-2以及SIFT软件,今天小编带大家了解另一款软件Mutation Taster。
Mutation Taster
新一代测序(NGS)项目发现的大部分基因变异要么是内含子的,要么是同源的。这些变异很难解释,因为它们对蛋白质表达和功能的影响往往没有那些错义变异或无义变异那么明显。这里呈现的Mutation Taster,用于评估DNA序列改变的致病可能性。它不仅用于预测氨基酸取代,而且还用于预测内含子和同义改变、短插入和/或删除(indel)突变以及跨越内含子-外显子边界的变异的功能后果。
Mutation Taster包括来自千人基因组项目(以下简称1000G)的所有可公开获得的单核苷酸多态性(SNP)和Indels,以及来自ClinVar和HGMD Public的已知疾病变异。在1000G或HapMap5纯合子状态下发现的四次以上的改变被自动视为中性。在ClinVar中标记为致病性的变异被自动预测为致病,并显示疾病表型。
为了减少假阳性剪接位点预测的数量,Mutation Taster只考虑在现有的内含子-外显子边界上剪接位点的丢失或强度下降。内含子-外显子连接的两个碱基对内的序列变化被认为是剪接位点的丢失。Mutation Taste能够分析跨越内含子-外显子连接的序列变化;这些可能干扰正常剪接,因此具有相当大的致病可能性。
Mutation Taster的应用
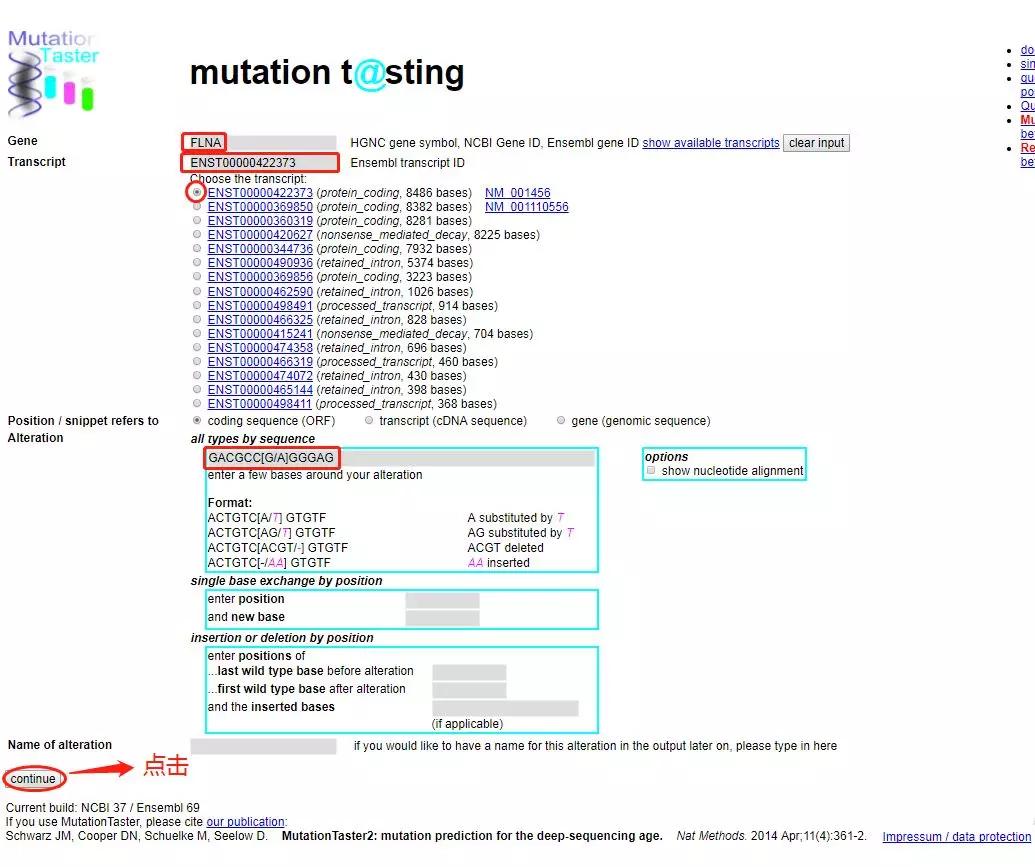
下面提供一个在线服务网址: http://www.mutationtaster.org/,以FLNA基因(NM_001456.3)c.4726G>A p. G1576R为例,简述查询方法。
第一步:准备数据。
进入Ensembl数据库,如下图,输入基因搜索。
网址:http://grch37.ensembl.org/Homo_sapiens/Info/Index
弹出如下界面后,选择人类词条。
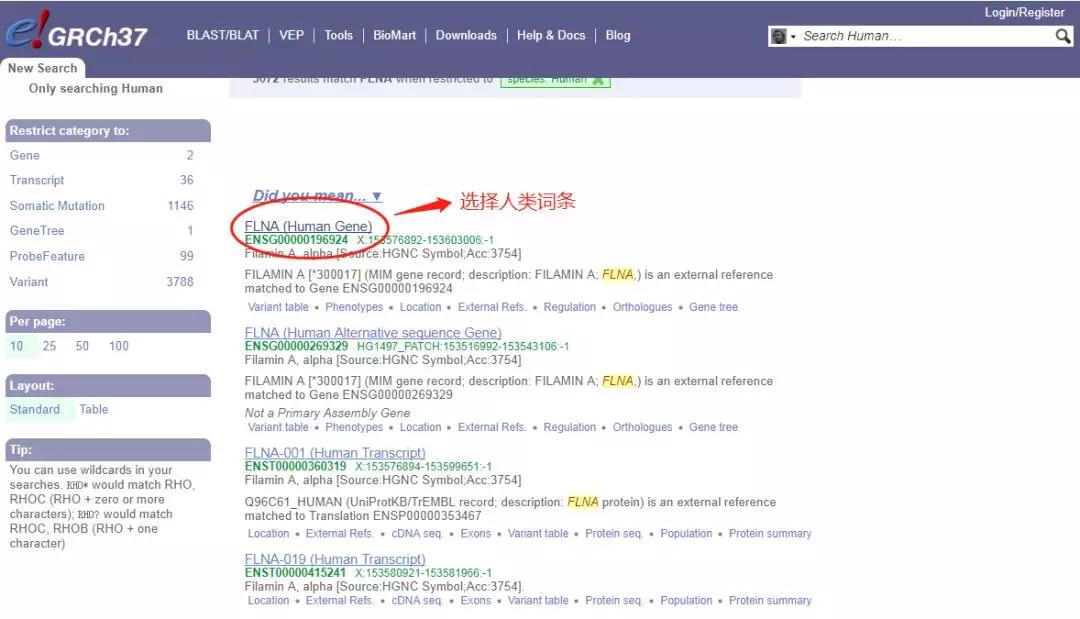
进入FLNA基因界面,其中ENST00000422373.1即为FLNA基因在Ensembl数据库中的ID。点击进入。

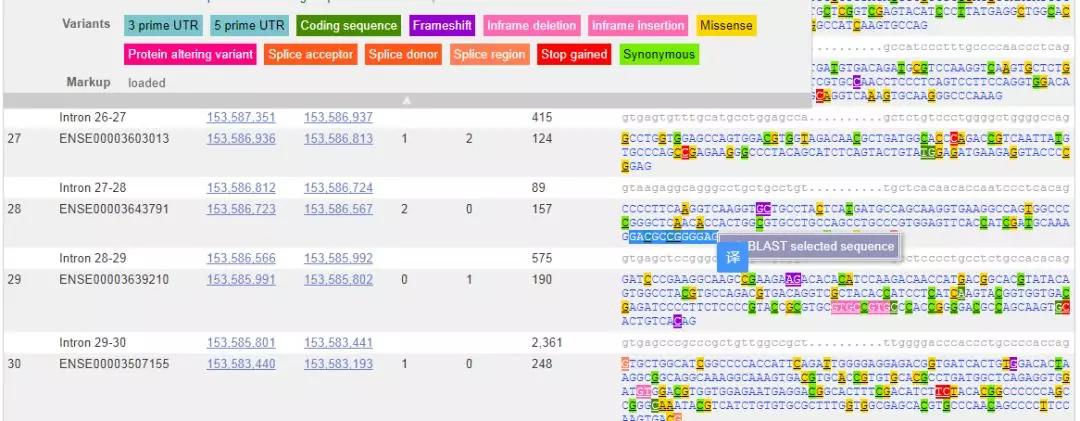
界面会跳转至如下状态,点击外显子,即可看到FLNA基因外显子序列。
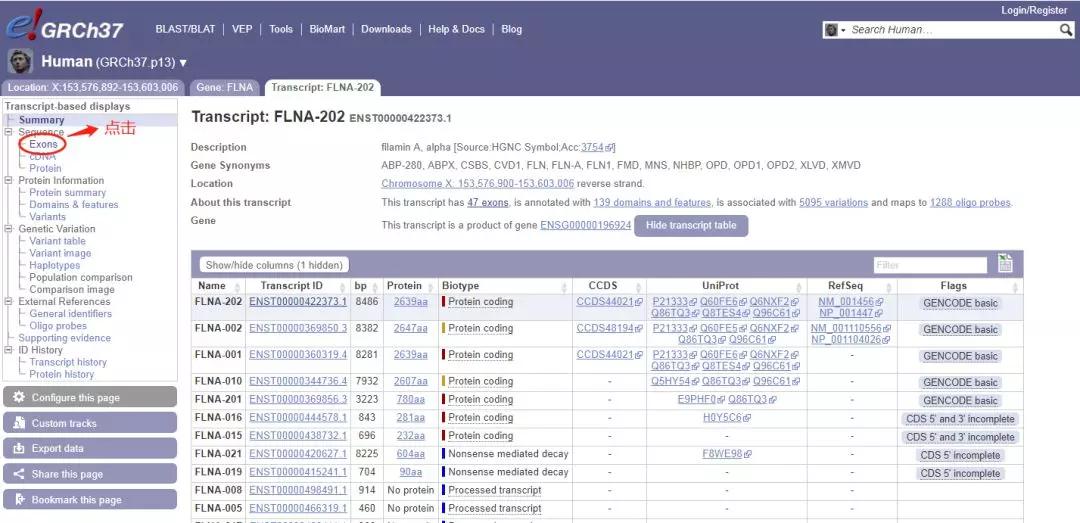
找到FLNA基因(NM_001456.3)c.4726G>A位置。点击碱基即可弹出一下对话框,点击Explorethis variant。
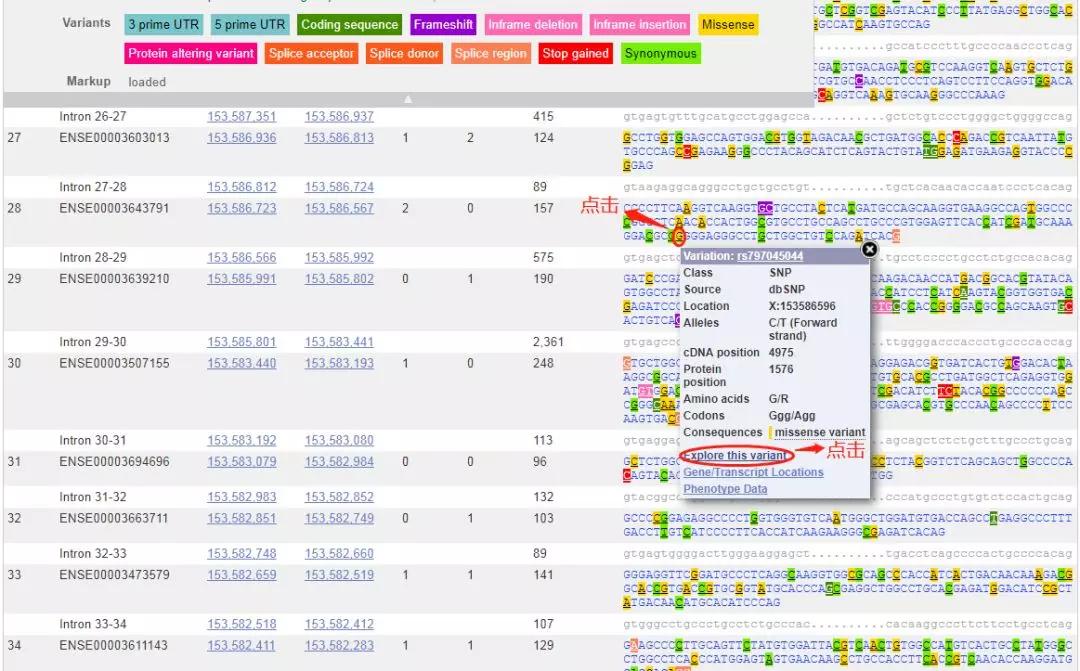
可弹出一下界面,即可看到在各个ID号下的c.。
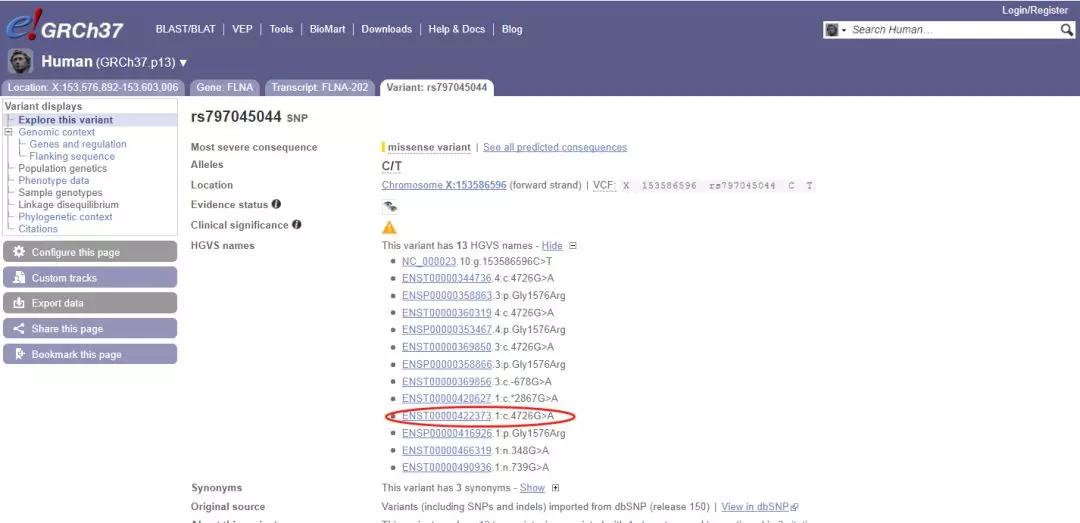
确认后,即可找到变异位点周围的碱基序列,将碱基复制出来,即做好了数据准备。
步骤二:查询蛋白损伤预测。
进入MutationTaster界面
输入内容后如下图,点击continue
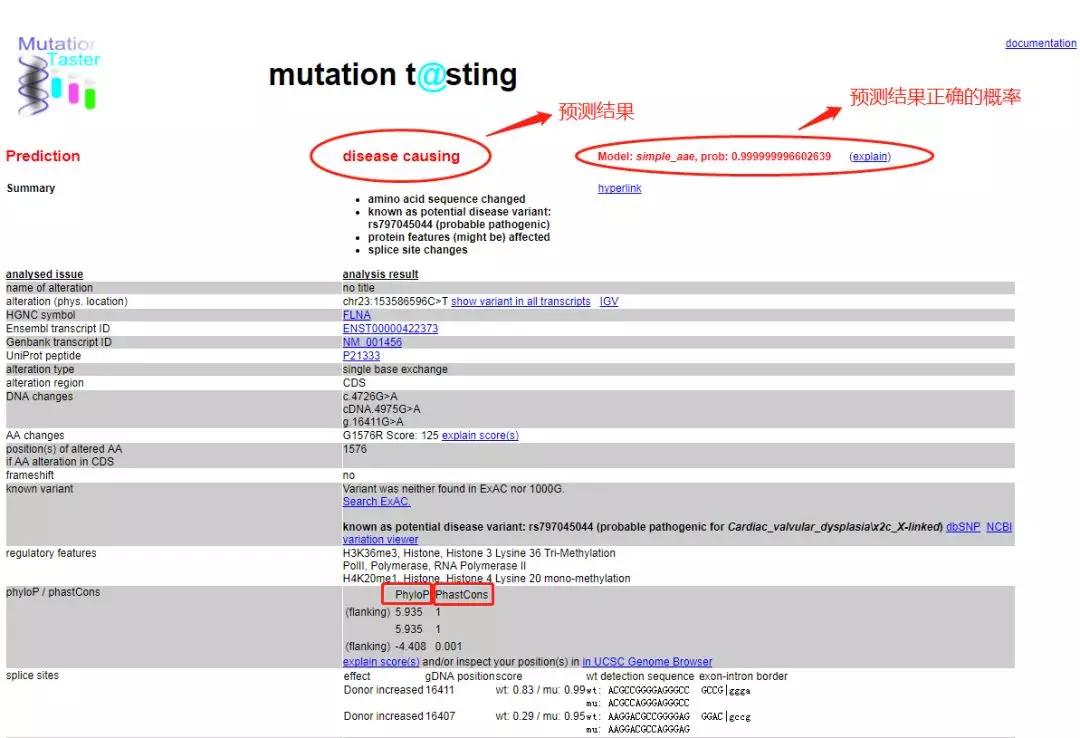
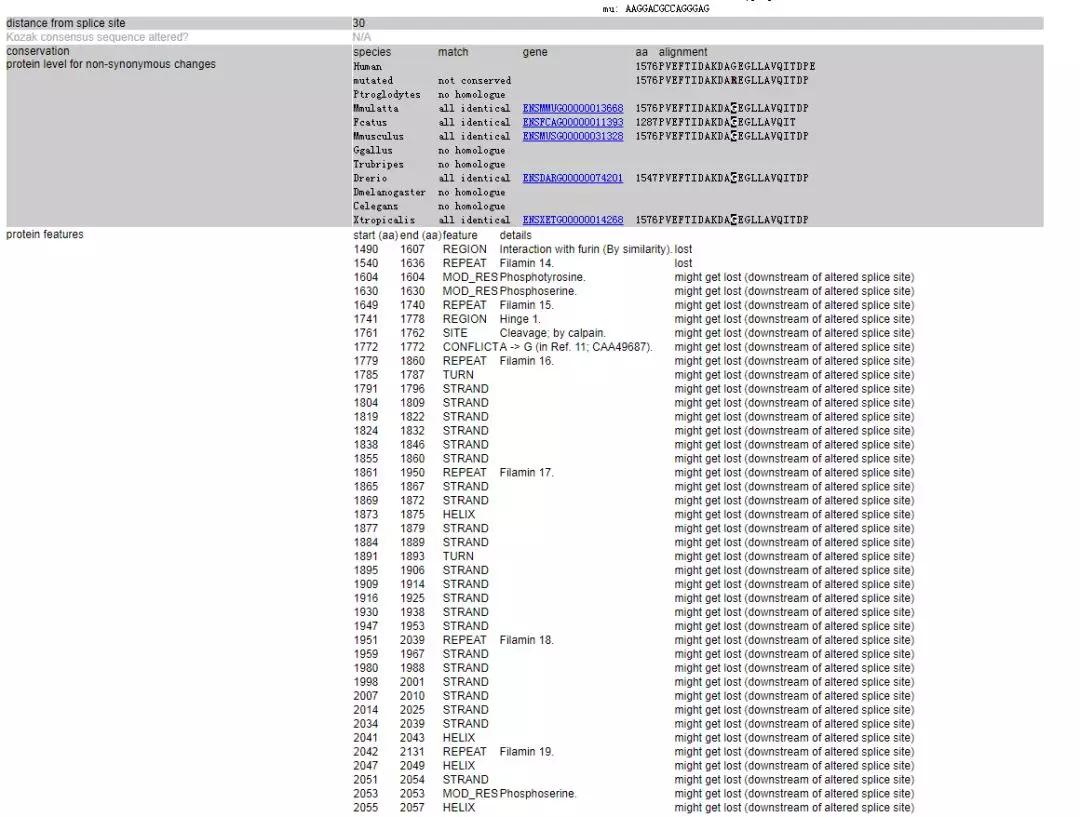
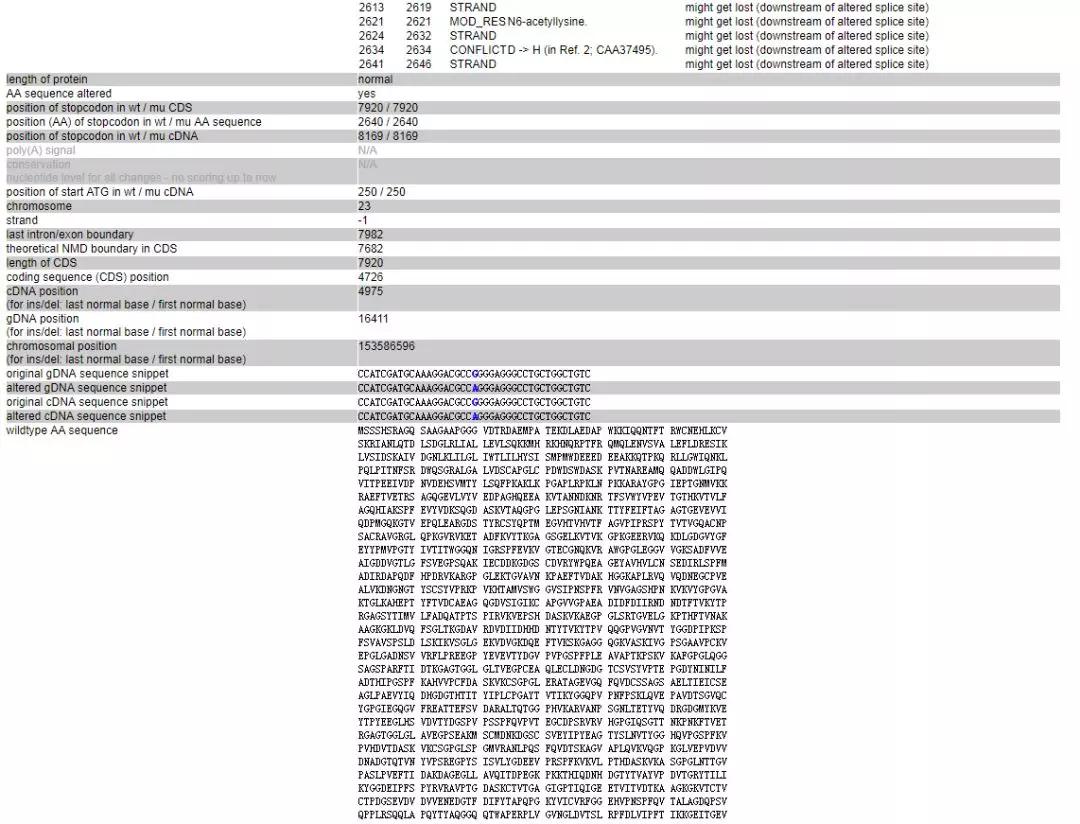
即可弹出如下界面,其中phylop和phastcons都是预测保守性的,他们的区别是一个只看该点在物种上的保守性,另一个是还看该店前后序列的保守性,具体可以看Mutationtaster网站上的相关说明。phylop为正数即保守,数字越大越保守,负数即不保守。phastcons是一个0-1的值,越接近1越保守,越接近0越不保守。


- 本文固定链接: https://maimengkong.com/morejc/803.html
- 转载请注明: : 萌小白 2021年11月14日 于 卖萌控的博客 发表
- 百度已收录
