R语言非常讲究数据的整理,我们在7-13天的R语言学习内容中,着重都是关于R语言的整理,各种方法对数据进行整理,查看,对变量进行转换。dplyr包,主要也用于数据清洗和整理,该包专注dataframe数据格式,从而大幅提高了数据处理速度。这个包对数据处理的方式相对之前的方法,更加简单,是医学数据分析必须要掌握的包。
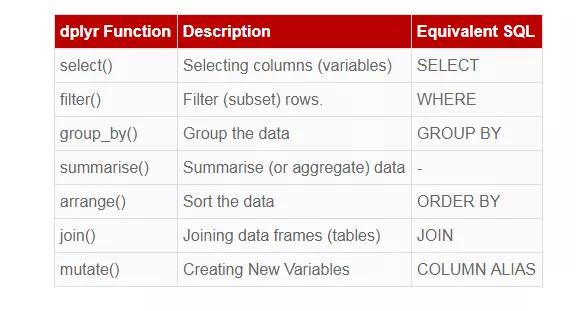
dplyr包存在着上百个函数来帮助进行数据的整理,由于篇幅的关系,本文对主要的函数进行介绍,包括下述五个函数用法:
• 筛选: filter()
• 排列: arrange()
• 选择: select()
• 变形: mutate()
• 汇总: summarise()
• 分组: group_by()
• 合并:join()
首先,安装和导入dplyr
install.packages("dplyr")
library(dplyr)
导入之前数据集
t1<-read.csv("elder1.csv")
t2<-read.csv("elder2.csv")
str(t2)
str(t1)
1 筛选:filter()
按给定的逻辑判断筛选出符合要求的子数据集,之前我们已经通过大量的方法介绍过子集产生的方法,而filter()方法则更直接。
#产生t11数据集,SBP收缩压>=140的子集
t11<-filter(t1,SBP>=140)
#产生t11数据集,SBP收缩压>=140且DBP>=90舒张压的子集
t12<-filter(t1,SBP>=140,DBP>=90)
# 产生t11数据集,SBP收缩压>=140或DBP>=90舒张压的子集
t13<-filter(t1,SBP>=140 | DBP>=90)
#产生t11数据集,SBP收缩压>=140且DBP>=90舒张压的子集
t14<-filter(t1,SBP>=140 & DBP>=90)
str(t11)
str(t12)
str(t13)
# 上述程序,如果用常规R语言,则要
t12<-t1[t1$SBP>=140 & t1$DBP>=90, ]
相对来说更为复杂。
#分类数据转换
t21<-filter(t2,sex==1)
2 排列: arrange()
排序功能,我们之前也学过,比如order(). Arrange 方法其实也更简单。
#根据SBP从小到大进行排序
arrange(t1,SBP)
#根据DBP从大到小进行排序
arrange(t1,desc(DBP))
#根据SBP和DBP,从小到大进行排序,先排SBP,SBP相同时,再排DBP
arrange(t1, SBP, DBP)
#根据SBP和DBP,从大到小进行DBP排序,DBP相同时,再排从小到大根据SBP排序
arrange(t1, desc(DBP), SBP)
3 选择: select()
用列名作参数来选择子数据集,这个功能很有意思,虽然医学数据分析用的不多。
t15 <- select(t1, starts_with('D')) #将D打头的变量名筛选出来
t16 <- select(t1, -DBP, -SBP) #排除DBP和SBP后,剩下的筛选出来
t17<- select(t1, contains('D') #将包括D字母打头的变量名筛选出来
t18<- select(t1, DBP, everything()) #将DBP变量放在数据库最全面一列
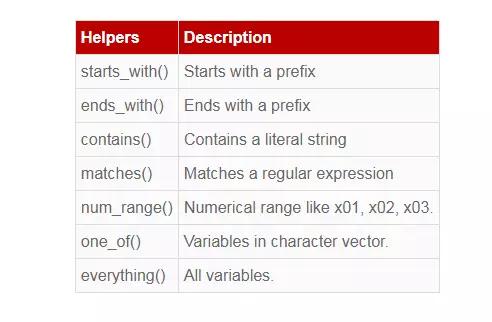
select ( ) 更多的参数设置如下表
4.变形: mutate()
对已有列进行数据运算并添加为新列,这个是非常重要的数据转换功能。
# 计算BMI指数
t19 <- mutate(t1, bmi=weight/(height^2)*10000)
# 可以同时产生多个变量
t110<- mutate(t1, x1=weigh*2, x2=height/100)
mutate( )可以调用的函数很多,以下是部分函数
log(), log2(), log10(): 对值求 log;
lead(), lag(): 返回序列中当前位置前第几个值或后第几个值;
cume_dist(): 计算比当前值还小的值的比例, 相当于计算 density;
ntile():把数据分成若干块, 看每个数据在具体拿一个块;
cumsum(), cummean(), cummin(), cummax(), cumany(), cumall():计算和 (sum), 均值 (mean), 最小值 (min), 最大值 (max), 任何为真 (any), 所有为真 (all);
na_if():把特定地值转换为 NA;
coalesce(): 找出若干列中第一个不为 NA 的值;
if_else(): 向量化的 ifelse 函数的效果.
recode: 把一系列值转换为其他值
case_when: 多条件选择.
比如:
t111<- mutate(t19, lgbmi=lg(bmi)) # 计算BMI指数的对数
t21<- mutate(t2, income=recode(income,"1"=1,"2"=0)) # 对income变量重新赋值
5 汇总: summarise()
summarise()函数以及衍生函数,包括summarise_all, summarise_at,summarise_if主要进行数据的统计描述。
一般情况下,它们需要同时调动以下等其它函数来共同完成。
min():返回最小值
max():返回最大值
mean():返回均值
sum():返回总和
sd():返回标准差
median():返回中位数
IQR():返回四分位极差
n():返回观测个数
n_distinct():返回不同的观测个数
first():返回第一个观测
last():返回最后一个观测
nth():返回n个观测
#求DBP的均数和中位数
summarise(t1, DBP_mean = mean(DBP), DBP_median = median(DBP))
#求DBP和SBP的总个数、均数和标准差,需要调用summarise_at 函数,var()函数,funs() 函数
summarise_at(t1, vars(DBP, SBP), funs(n(), mean, median))
#求定量变量数据的均数和标准差,需要调用summarise_if 函数,var()函数,funs() 函数
summarise_if(t1, is.numeric, funs(n(),mean,median))
#存在着缺失值的时候,计算定均数和标准差,需要调用summarise_at 函数,var()函数,funs() 函数
summarise_at(t1, vars(DBP, SBP),
funs(n(), missing = sum(is.na(.)),
mean(., na.rm = TRUE),
median(.,na.rm = TRUE)))
6 分组: group_by()
当对数据集通过group_by()添加了分组信息后,mutate(),arrange() 和 summarise() 函数会自动对数据库执行分组操作。
group_by()的功能类似于SPSS拆分文件夹的功能,十分地好用。
tt1 <- group_by(tt, sex) # 首先根据性别进行数据库拆分。
tt2<- summarise(tt1, count = n()) # count = n()用来计算次数
或者直接将上述两句整合成一句
tt3 <- summarise_at(group_by(tt,sex) , vars(DBP, SBP), funs(n(), mean(., na.rm = TRUE)))
tt3
7. 数据库合并join()
在之前的内容中,我们介绍过用rbind,cbind,merge等函数进行数据库合并,但利用join()函数,花样更多。
left_join(t1,t2)
right_join(t1,t2)
inner_join(t1,t2,by=c(“”))
full_join(t1,t2, by = c("first", "last"))
semi_join(t1,t2, by = c("first", "last"))
anti_join(t1,t2, by = c("first", "last"))
前4种属于变形连接(mutating joins),后2种属于过滤连接(filtering joins)。
semi-joins基于第二个数据集的信息来过滤第一个数据集的数据。anti-joins找出合并时哪些行不能匹配第二个数据集

8. dplyr 包其它重要函数
由于篇幅的关系,不再一一介绍dplyr 包的函数,这里可以介绍下,有兴趣者可以进一步学习,比如dplyr 包的重命名rename(), 数据集重新再抽样sample(),数据转换transmute()都是是否有用的函数。
DAY14的内容就介绍到这里!
转自:医学论文与统计分析
- 本文固定链接: https://maimengkong.com/kyjc/956.html
- 转载请注明: : 萌小白 2022年6月2日 于 卖萌控的博客 发表
- 百度已收录
