
近几十年,系统生物学慢慢走近科研者的视野,主要着眼于研究细胞信号转导、基因调控网络和生物系统的组成。多组学联合分析可以由表及里,解析直接原因和根本原因,深入阐释作用机理。在多组学的联合应用中,最终都要落脚到功能注释和通路富集分析,其中基于通路富集分析的图形展示丰富多样,通过此分析可找到与研究相关的通路、关键基因/代谢物,以便进行后续的数据挖掘。
小鹿在往期文章中,向大家推荐过一个代谢组学分析工具MetaboAnalyst网站(详情请戳实用锦囊:MetaboAnalyst代谢组学分析工具实操)MetaboAnalyst网站链接: https://www.metaboanalyst.ca/ )。MetaboAnalyst网站主要用于数据筛选和数据标准化处理功能、代谢通路富集等功能。
在上期的推文中我们用MetaboAnalyst快速、简单完成了数据筛选和代谢通路富集图。但拿下图用MetaboAnalyst画出来的气泡图来看,你看出来了吗?似乎缺少点什么?
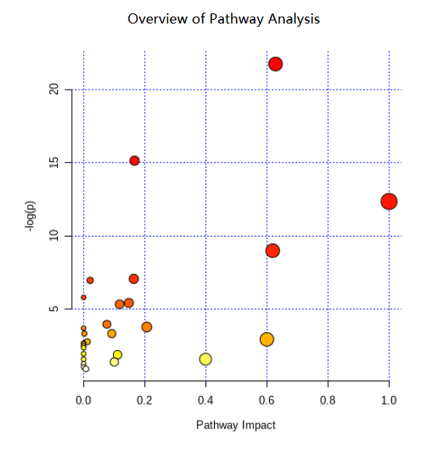
嗨~~发现了吗?这张气泡图有一个显而易见的缺点,那就是没有显示代谢通路的名称。那么,有没有什么方法可以满足我们看到代谢通路名称的需求呢?
答案是肯定的,方法也是有的。它来自贝尔实验室,它最擅长的就是用五彩斑斓的图给你好看,它是可视化的主宰……数据绘图的先锋……分析的神器——R语言!
来,先来看看我们将要完成的目标——为上面的图添加上代谢通路名称!
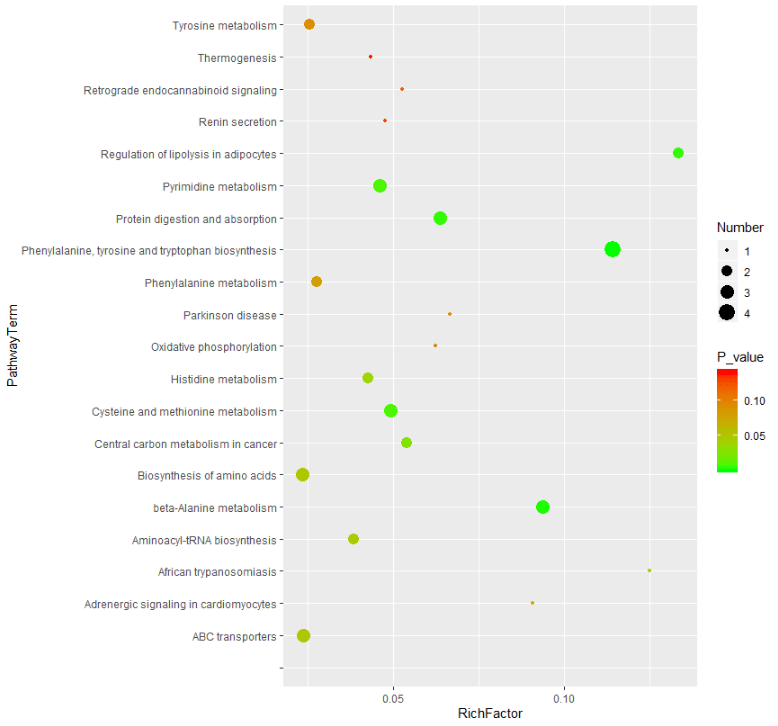
这张气泡图中,左侧是代谢通路的名称。横轴则表示richfactor,指富集因子,是用代谢通路富集结果中的in set除以in background。如果大家用的是MetaboAnalyst的富集结果表格,可以将这个参数替换为Impact。
然后我们用气泡大小来表示, 参与这条通路的差异代谢物数目,用气泡颜色来表示该代谢通路的超几何检验p值。
思路我们已经有了,接下来就是实际操作的部分了!
首先我们要请出我们万能的,无敌的,可视化作图绕不过去的R语言包:ggplot2!
相信ggplot2的作图逻辑,大家已经熟悉了,它的核心理念就是:将绘图与数据分离,数据相关的绘图与数据无关的绘图分离,按图层作图,保有命令式作图的调整函数以及将常见的统计变换融入到了绘图中。
有了气泡图的绘图思路,有了ggplot2的作图逻辑,那么接下来的事情就是水到渠成啦~~
数据处理
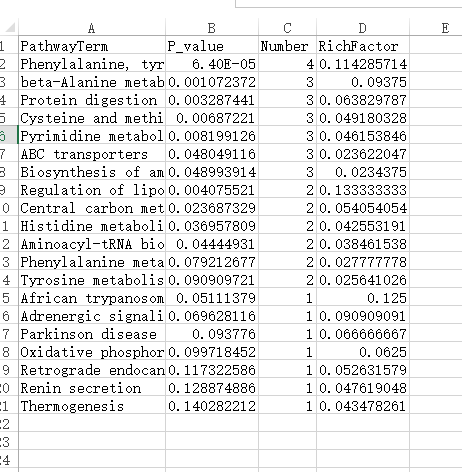
根据我们对气泡图的分析,我们用于作图的数据应该包含以下几列:
1.代谢通路名称(PathwayTerm);
2.横轴的Richfactor或是Impact;
3.参与这条通路的差异代谢物数;
4.代谢通路的超几何检验p值;
最终完成的数据应该是这样的:
包的导入和数据的读取
library(ggplot2)
setwd("G:/R")
#这里用的是csv格式的读入文件,大家可以根据自己的实际情况替换为其他格式的读入文件
pathway = read.table("DE.csv",header=T,sep=",")
作图
#确定使用数据及坐标轴映射关系
pp=ggplot(pathway,aes(RichFactor,PathwayTerm))
#正式画图并添加size和color的参数
pbubble=pp+geom_point(aes(size=Number,color=P_value))
#添加颜色的变化
pbubble+scale_colour_gradient(low="green",high="red")
结束了,画完了,是不是超级简单!
运用ggplot2,不仅能让我们每天都能有新发现,而且可以让我们在代码的运用和变换之间感受无穷乐趣。
可是又有老师会觉得R语言不是我所追求的,我只想快速又简单的画出具有代谢通路名称的代谢图怎么办呢?小鹿还有一个好的分析平台(点击文末阅读原文即可访问),欧易/鹿明生物云平台,极速出图体验,没有更快,只有最快~~快来访问试一试吧!
转自:欧易生物- 本文固定链接: https://maimengkong.com/zu/867.html
- 转载请注明: : 萌小白 2022年4月8日 于 卖萌控的博客 发表
- 百度已收录
