每年的1-3月份是很多老师撰写国自然本子的主要时期,但是每年这个时期都会有老师因为各种各样的原因,导致实验计划延迟而拿不到需要的实验数据,因此需要借助数据库已有的数据做数据分析为基金申请做理论支撑。另外由于很多突发的因素产生,科研人员往往无法及时开展实验工作,因此更需要借助数据库数据,这在今年的新冠肺炎疫情中体现特别明显,在疫情发生的早期,就陆续发表了多篇研究论文,分析讨论新冠肺炎病毒靶向蛋白血管紧张素转化酶2(ACE2)在人体不同组织不同细胞中的表达分布情况,为2019-nCoV在消化系统、呼吸道、生殖系统等的潜在感染途径提供了生物信息学证据,为新冠病毒肺炎的预防和治疗提供参考数据和部分理论依据(多篇单细胞测序文章助力新型冠状病毒感染、传播机制研究)。
近期小编收到很多老师的数据分析需求,基本都是围绕国自然申请和单细胞数据再分析,在和老师沟通过程中,发现有些老师对于数据再分析所需要的原始测序数据下载不甚了解,因此撰写此SOP用于指导数据下载。
GEO简介
GEO(Gene Expression Omnibus)是一个公共存储库,可以存档和自由分发由科学界提交的全套微阵列,新一代测序和其他形式的高通量功能基因组数据,提供了工具来帮助用户查询和下载实验和管理基因表达谱。
GEO数据库具体存放四类数据:GDS、GSE、GSM、和GPL。
GDS号(GDSxxx)对应的一个同一平台的数据集, 包括微阵列和高通量测序产生的数据;
GSE号(GSExxx)对应的是整个研究项目的系列的数据,可能涉及不同平台;
GSM号(GSMxxx)是具体某个单一样品的数据信息,只能是单一平台的数据;
GPL号(GPLxxx)对应平台的信息,例如Illumina测序平台型号、芯片型号等。
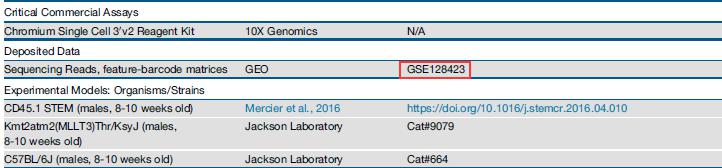
一般高通量测序文章发表时会将原始数据上传至GEO数据库并在文章中提供GSE 号,如果想对某些文章的数据进行在分析,可以在GEO数据库搜索文章中的GSE号。
GEO数据下载
一、进入GEO数据库
1.进入NCBI主页(https://www.ncbi.nlm.nih.gov/)
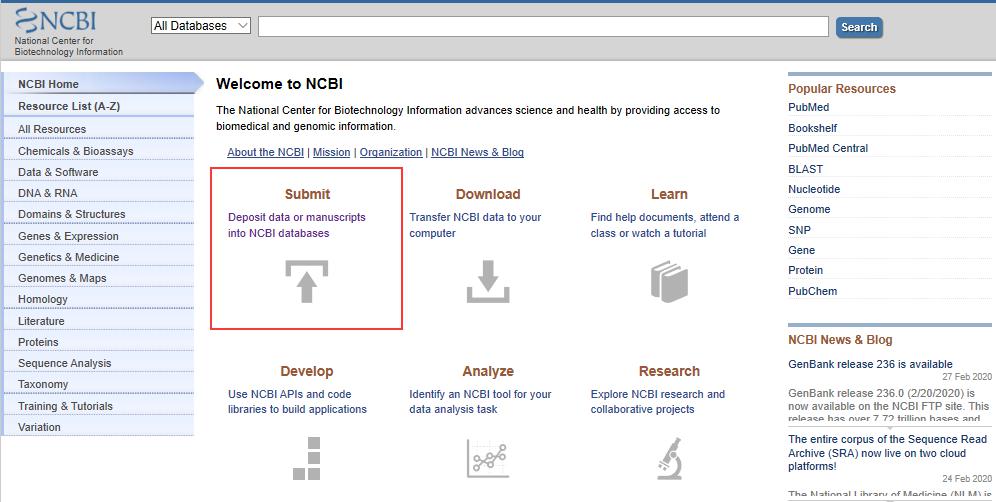
2. 点击Submit进入数据提交页面,下拉至Other Tools
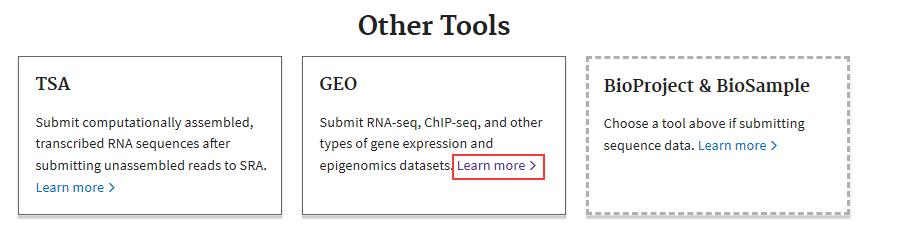
3. 选择GEO,点击Learn more,进入数据提交页面。
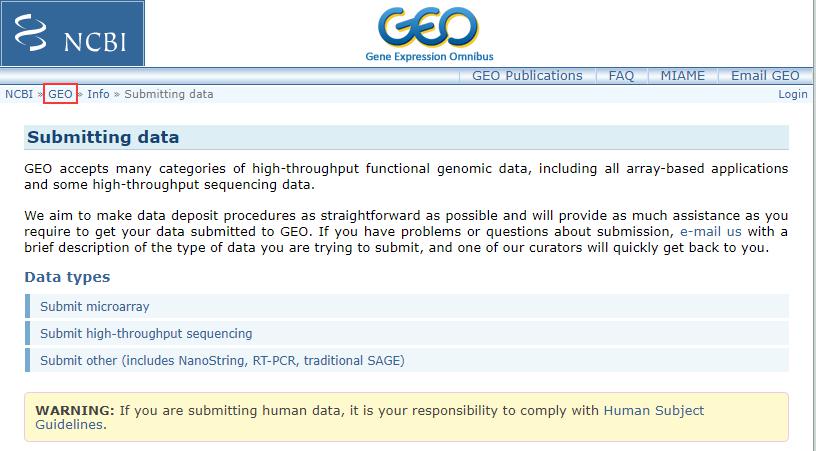
4.进入GEO数据提交页面后,点击“GEO”进入GEO主页(https://www.ncbi.nlm.nih.gov/geo/)
GEO数据下载
1.输入数据集或者样本ID
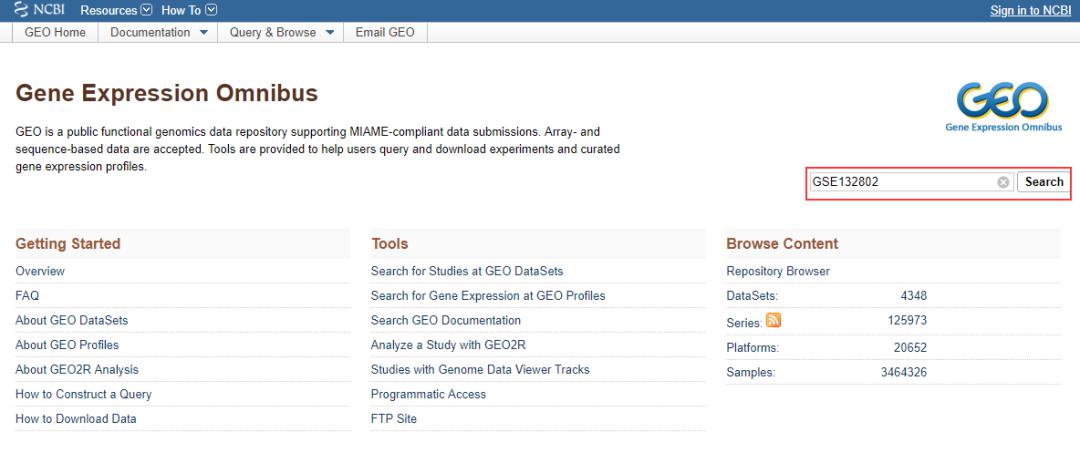
2.进入GSE页面
需要确认此数据是否采用10X Genomics平台(summary可能包含单细胞测序平台信息,若不包含,需搜索并查阅数据来源文章)

3.拉至页面最下方,选择需要下载的样本并点击
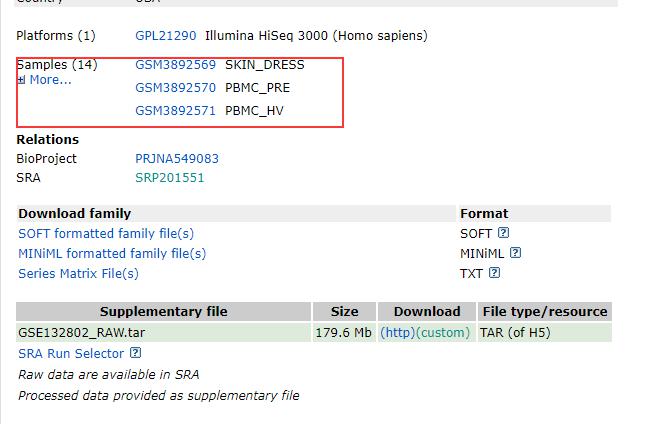
4.进入样本GSM页面
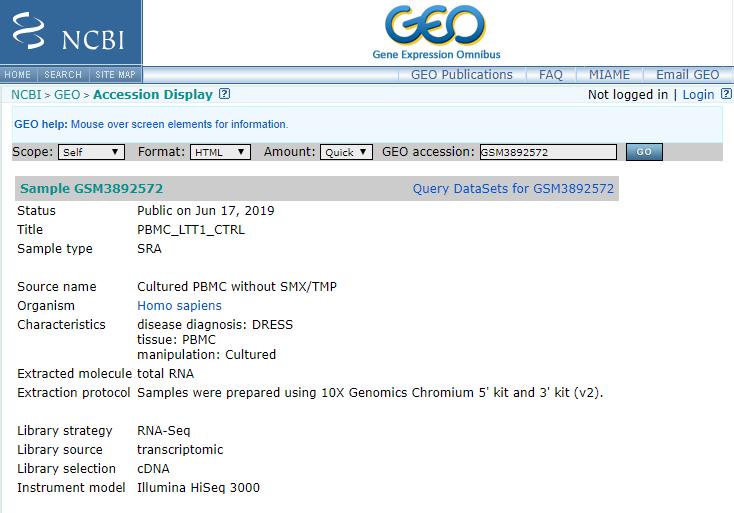
5.下拉并点击SRA数据ID

6.进入SRA页面,下拉至最下方点击数据链接

7.进入SRA数据库,点击Data access
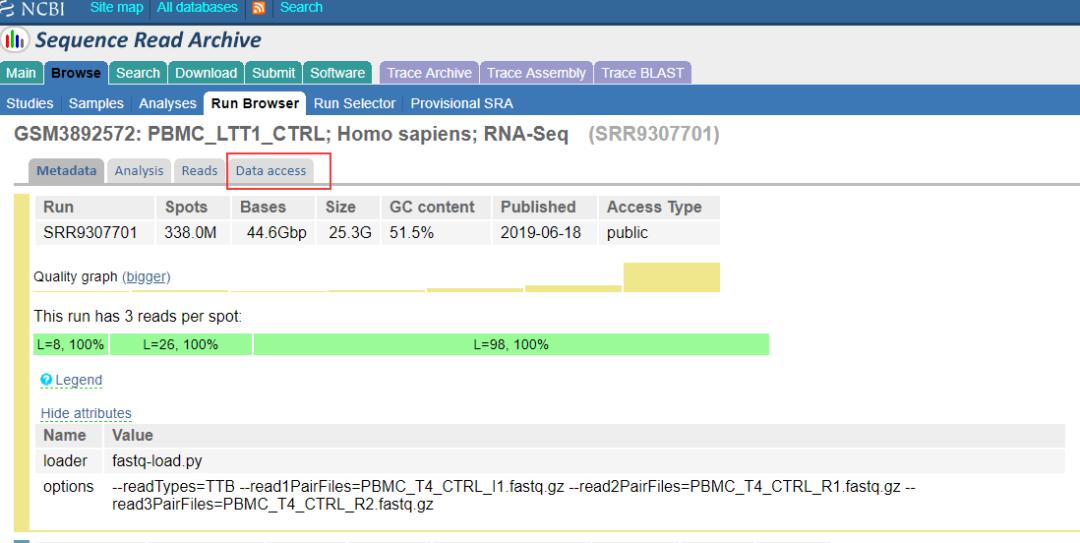
8.进入数据下载页面,下载fastq格式原始测序数据
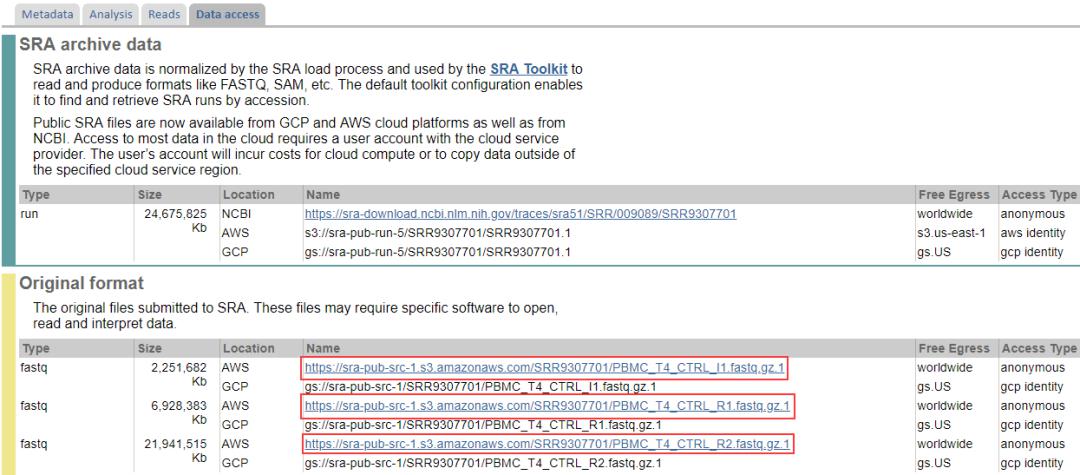
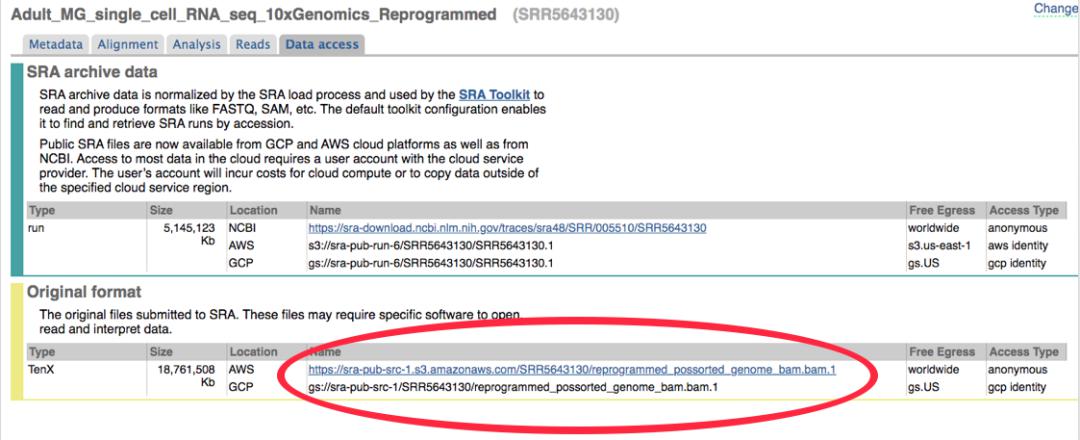
9.BAM数据下载
有时候10x fastq不会被上传到数据库,相反客户会上传bam文件(除了FASTQ文件以外,SRA鼓励提交10x BAM文件),bam是Cell Ranger生成的输出文件之一。如果只有bam文件而无FASTQ文件存在,可以在SRA的“Data access”选项卡中找到bams并下载,下载的bam文件使用10x官方提供的转换工具 bamtofastq(https://support.10xgenomics.com/docs/bamtofastq)将其转换为fastq格式文件。
- 本文固定链接: https://maimengkong.com/zu/1779.html
- 转载请注明: : 萌小白 2024年8月20日 于 卖萌控的博客 发表
- 百度已收录
