用R或Python进行数据分析或数据可视化在生物数据的分析中已经应用得越来越广泛,并为生物学研究解决了大量的问题。但是术业有专攻,在生物学领域深耕的专家学者可能不太熟悉编程技巧,感觉对生信分析一筹莫展,只能找别人帮忙协助分析。那么,有没有方法可以让不熟悉编程技巧的专业研究人员也能自己很方便地进行数据分析,并把数据按自己的思路进行可视化展现出来呢?答案是肯定的,具体怎么操作,且听编者慢慢道来。
首先,本期给大家带来的是Excel协助的生物信息数据分析技巧。Excel是微软Office的其中一个工具,收费。不过,没关系,不想付钱我们可以用WPS的“表格”,和Excel完全兼容,部分功能还超过微软的Excel。
下面我们就从蛋白质组raw文件经过MaxQuant搜库后的结果出发,向您展示如何进行一系列处理后得到差异显著结果。
数据清理
MaxQuant提供的蛋白定量表中包含了部分错误的匹配数据,需要预先去除。我们对结果文件的三列(Only identified by site、Reverse、Potential contaminant)进行处理,即利用Excel的筛选功能,将这三列数据中含有“+”的行剔除。选中这几列,按“Ctrl + Shift + L”(或【点击数据】—【自动筛选】)即可调出筛选功能。
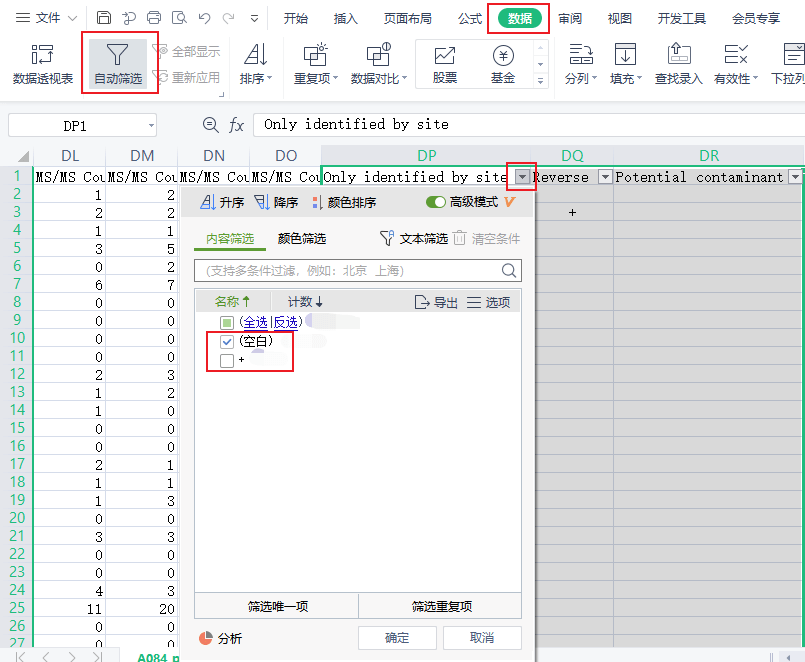
归一化
由于上样量等的差异,每组样本的定量结果可能不能直接比较,需要先进行归一化处理。目前文献报道的归一化方法很多,下面的演示我们采取中值归一化的方法。
(1)数据清理
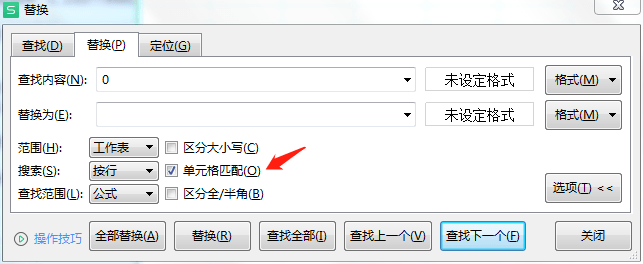
定量表中的0值,一般是指没鉴定到,但在实际样本中是不存在还是定量值太低,不好说,直接都当0看待会导致不必要的偏差。为了在后续寻找中值时不受0值干扰,在进行归一化之前,要先将0替换为空值。操作方法:查找内容为0,替换为空值(不是空格,需要删除格子中所有东西),点击“选项”,选择“单元格匹配”,点击全部替换,即把表格中所有0的单元格替换成空值。如下图:
(2)计算各样本的中值
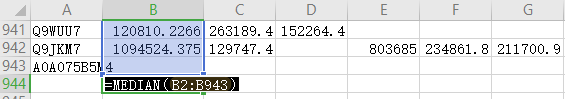
接下来我们需要找到各列的中值,可以用MEDIAN公式寻找中值(如果需要采用总和或最大值归一化,可以用SUM、MAX计算总和或最大值),进行归一化。
在一个样本列的末尾输入“=MEDIAN(该列数据的单元格范围)”,计算出该样本的中位数。然后,鼠标按住该单元格的右下方向右拖动到合适的范围,批量计算出所有样本的中位数。
(3)归一化
在新的列中,用公式“=单元格/中位数所在的单元格”进行归一化。由于中位数所在的单元格的行数是固定不变的。所以我们在行数前加上一个“$”符号,该符号表示的是锁定的意思。然后,鼠标按住该单元格的右下方向右拖动到合适的范围,批量进行归一化。对同一列的批量操作可以在选中单元格后,将鼠标放置在单元格的右下角变成实心“十”后,双击即可对整个列进行操作。

补值
补值是对缺失值的一种估算。在无标定量的MaxQuant结果中,由于我们在搜库时采取了Match between run的方式用一级数据进行定量值的估算,以此依然缺失的数据基本上为定量值很低的结果,我们可以用很低的值进行补值。在此,我们演示用最小值的1/2进行补值。
首先,将归一化后定量结果依然为0的单元格(在归一化的公式操作中,空值经公式计算后得到0值)替换成空值后,找到最小值。可以通过“=MIN(单元格范围)”公式计算最小值,或者选中全部定量列,在下方状态栏中显示最小值。
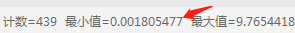
然后取最小值的1/2进行补值,操作步骤如下:
按“Ctrl + G”调出”定位”,选择“空值”,点击“定位”,则把所有空值的单元格都定位上,直接输入要补的值,填入到当前选中的单元格中,然后按“Ctrl+Enter”即可对所有空值进行补值。
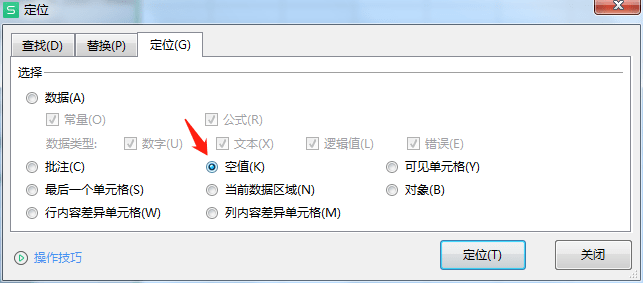
注释信息的匹配
在某些情况下,我们的定量表里可能只有“Accession”信息,但在实际分析中还需要Gene Symbol的信息。我们可以从数据库中提取所有蛋白对应的Gene Symbol信息表,但需要提取我们的鉴定列表的Gene Symbol则需要用Excel 的VLOOKUP函数进行匹配。
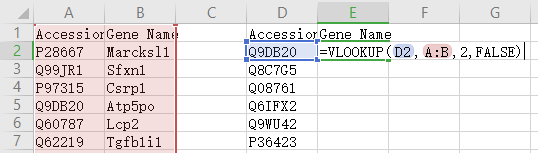
如图,左侧是所有蛋白的Accession和Gene Symbol(也称Gene Name)的对应关系,右侧是我们想要匹配的蛋白列表。用“=VLOOKUP(查找值,数据表,列序数,匹配条件)”。在这里,查找值是D列的第二行(D2);数据表的范围是A列到B列(A:B);列序数,我们想要匹配的是A,B两列的第二个(2);匹配条件,我们选择精确匹配(FALSE),如果希望忽略大小写可以模糊匹配(TRUE)。完成后,将鼠标放在该单元格右下角,变成实心“十”字后双击补全,即可获得所有蛋白Accession对应的Gene Symbol。
统计检验
完成了前面的归一化、补值后,我们需要进行不同分组数据的分析以便获得差异表达结果。在此,我们采用生物学研究中常用的T-test进行统计检验,并计算差异倍数(Fold change,FC)。
(1)统计检验(t-test)
采用Excel的公式“=TTEST(第一组数值,第二组数值,尾数,类型)”,计算出pValue。本演示数据是2组3重复,所以第一组数据是B2:D2,第二组数数据是E2:G2;尾数选择2,代表双尾;类型选择2,代表双样本等方差假设。随后对整列进行补全操作,即可得到所有蛋白的差异的pValue。
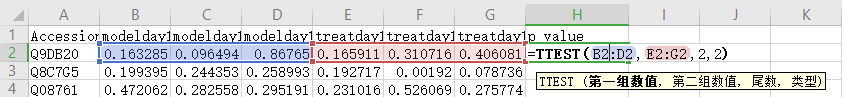
(2)计算差异倍数(FC)
计算差异倍数FC(即2组数据平均值的差)。通过函数“=AVERAGE(第一组单元格范围)/AVERAGE(第二组单元格范围)”,获得FC值,并补全列,即可得到所有蛋白的FC。

为上下调蛋白着色
Excel还给我们提供了一种很直观的可视化方式,即可在设定条件后,自动以不同颜色展示不同的结果。
在这里我们卡pValue<0.05和log2(FC)>1为上调,pValue<0.05和log2(FC)<-1为下调。log2的计算可以通过公式“=LOG(数值单元格,底数)”计算。
(1)筛选pValue
通过Excel的筛选功能,筛选出pValue小于0.05的数据。
(2)自动着色
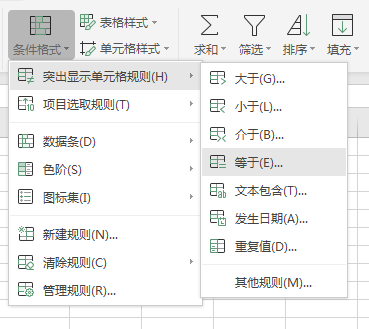
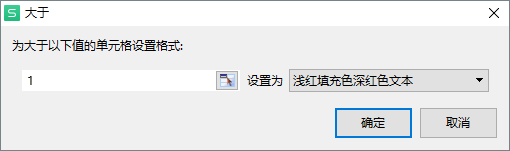
选中 “log2 ( FC ) ” 这列,点击【开始】 -- 【条件格式】 -- 【突出显示单元格规则】,选择大于,数值选择 1 ,设置为 “ 浅红填充色深红色文本 ” ;同样的操作步骤选择小于,数值选择 -1 ,设置为 “ 绿填充色深绿色文本 ” 。
效果如下图所示:
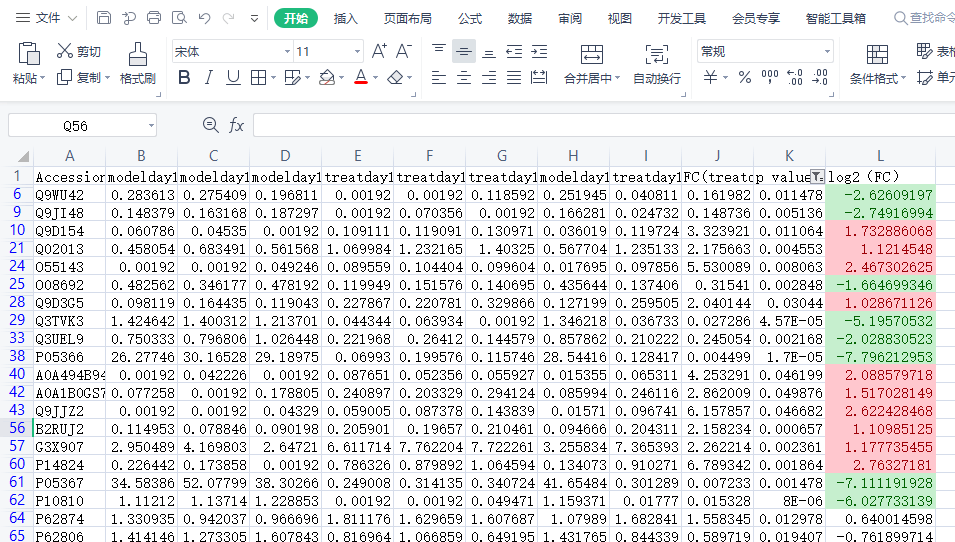
至此,我们用Excel完成了从MaxQuant搜索的原始搜库结果,经过数据清理、归一化、补值,最终进行差异筛选和不同颜色展示的整个过程。怎么样,是不是很简单。生信分析,我们不用编程也可以办到的。
当然,我们只是演示了一种最基础的数据处理策略,如果需要更复杂的方法,还需要发掘Excel的其它功能,或者采用其它软件辅助的方法实现。- 本文固定链接: https://maimengkong.com/zu/1687.html
- 转载请注明: : 萌小白 2024年2月11日 于 卖萌控的博客 发表
- 百度已收录
