CRISPR/Cas9是一项可对基因组特定靶基因进行编辑的DNA操控技术,该系统由sgRNA和Cas9蛋白组成,Cas9蛋白在sgRNA的引导下对靶位点处的DNA双链进行剪切,并产生一个平末端的双链DNA缺口,进而启动DNA损伤修复机制,通过非同源末端链接(Non-homologous end joining,NHEJ)或同源重组(Homologous recombination,HR)的方式将断裂上下游两端的序列连接起来。
目前,CRISPR/Cas9基因编辑技术在疾病基础研究、靶点验证、药物分子的高通量筛选、以及遗传性疾病的治疗等领域得到了越来越广泛的应用。sgRNA在CRISPR/Cas9基因编辑系统中具有准确识别靶基因序列的作用,其效果可影响编辑的效率、是否发生脱靶等,甚至对最终基因编辑的效果产生决定性作用。因此,设计合理有效的sgRNA是我们实现基因编辑的重要基础。
sgRNA的设计流程

sgRNA的设计原则
1. sgRNA的长度:S. pyogenes II型CRISPR系统(SpCas 9)一般为20nt。
2. sgRNA序列的碱基组成:基因特异的sgRNA 模板序列为位于PAM 序列前,PAM序列的特征为NGG (N可以为任意核苷酸),所以选择3'末端含有GG的sgRNA,这样可以构成PAM序列。同时,sgRNA的序列应避免以4个以上的T结尾,GC%含量最佳为30%-70%(40%-60%)。
3. sgRNA的序列与On-target和Off-target的匹配数都应尽可能的高,一般大于60,认为是可用的。
4. 如果构建U6启动子或T7启动子驱动sgRNA的表达载体,需要考虑sgRNA的5'碱基为G或GG,来提高其转录效率。
5. 全基因的脱靶效应分析,需要考虑脱靶位点的错配碱基数,最多不超过5个。
6. 如果想要造成基因移码突变,需要尽量靠近基因编码区的ATG下游,最好位于第一或第二外显子上。
sgRNA设计示范
此处以查询人类WNT10B基因为例。在NCBI Gene数据库中输入需要查找的人类WNT10B基因,查找的结果显示多个与WNT10B相关的基因,这些基因包括了不同物种的WNT10B同源基因。因而查找时需要注意该基因在NCBI的登录号与种属描述等。点击查询人类WNT10B基因,显示出该基因的基本信息(图1)。
 (图1) NCBI网站查询WNT10B基因信息
(图1) NCBI网站查询WNT10B基因信息
找到该基因的mRNA and Protein(s) 相关信息,查询可知WNT10B的mRNA 编号为 NM 003394.4。如果该基因可以通过可变剪接产生多个mRNA亚型的话,在下图中会依次记录(图2)。
 (图2)NCBI网站查询WNT10B基因mRNA信息
(图2)NCBI网站查询WNT10B基因mRNA信息
使用SnapGene软件对基因序列信息进行分析,如下图所示,WNT10B基因一共包含4个外显子,其中粉色区域为信号肽所在序列,该序列在蛋白质翻译加工过程中会被切除,sgRNA设计时应避免此段序列,最左边的的外显子序列也不参与蛋白质的翻译,也应该避免采用此段序列,所以最佳的sgRNA靶向序列应采用图中蓝色选定区域(图3)。
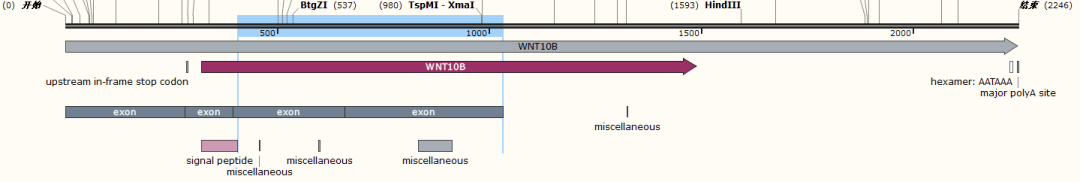 (图3)WNT10B基因结构组成图示
(图3)WNT10B基因结构组成图示
选取上图中序列作为sgRNA的靶序列,在sgRNA设计网站输入靶基因信息和设计相关信息。
现在有多种sgRNA设计网站可供选用,常用的设计网站包括:
1. CRISPOR(http://crispor.tefor.net/)
2. GPP Web Portal (https://portals.broadinstitute.org/gppx/crispick/public/)
3. E-CRISP (http://www.e-crisp.org/E-CRISP/)
4. CHOPCHOP (http://chopchop.cbu.uib.no/)
此处我们选择张锋实验的CRISPOR(http://crispor.tefor.net/)来进行sgRNA的设计,输入上文选择的靶序列,选定基因对应的物种信息和想要使用的Cas9和PAM种类后点击提交(图4)。
 (图4)sgRNA设计网站界面
(图4)sgRNA设计网站界面
网站生成的sgRNA序列和相关信息如下图所示,信息包括sgRNA的位置,网站给出的特异性评分,切割效率评分,潜在的脱靶结果和脱靶位点相关信息(图5)。
 (图5)网站生成的sgRNA方案和分析
(图5)网站生成的sgRNA方案和分析
sgRNA脱靶分析
网站会生成多个备选sgRNA结果,我们需要根据信息筛选出最佳的sgRNA,上图中根据生成的sgRNA信息,我们可以从特异性评分,剪切效率评分,潜在脱靶情况和脱靶位点信息几个方面进行分析,选取最佳sgRNA。上图中第一个和第五第六个sgRNA被标注为 inefficient,所以我们可以优先考虑第二第三和第四个sgRNA作为备选方案。
除了该网站自带的脱靶信息分析以外,根据选择的sgRNA,我们也可以通过脱靶分析网站对sgRNA进行脱靶分析。
推荐使用CCTop(https://cctop.cos.uni-heidelberg.de:8043/)在线网页进行预测。将sgRNA序列输入,选定相应种属基因组进行分析。并且对获得的遗传材料进行检测。挑选前10个潜在脱靶位点,通过PCR测序验证是否脱靶。如果实验要求较为严格的,则需要通过全基因组测序鉴定脱靶情况(图6)。
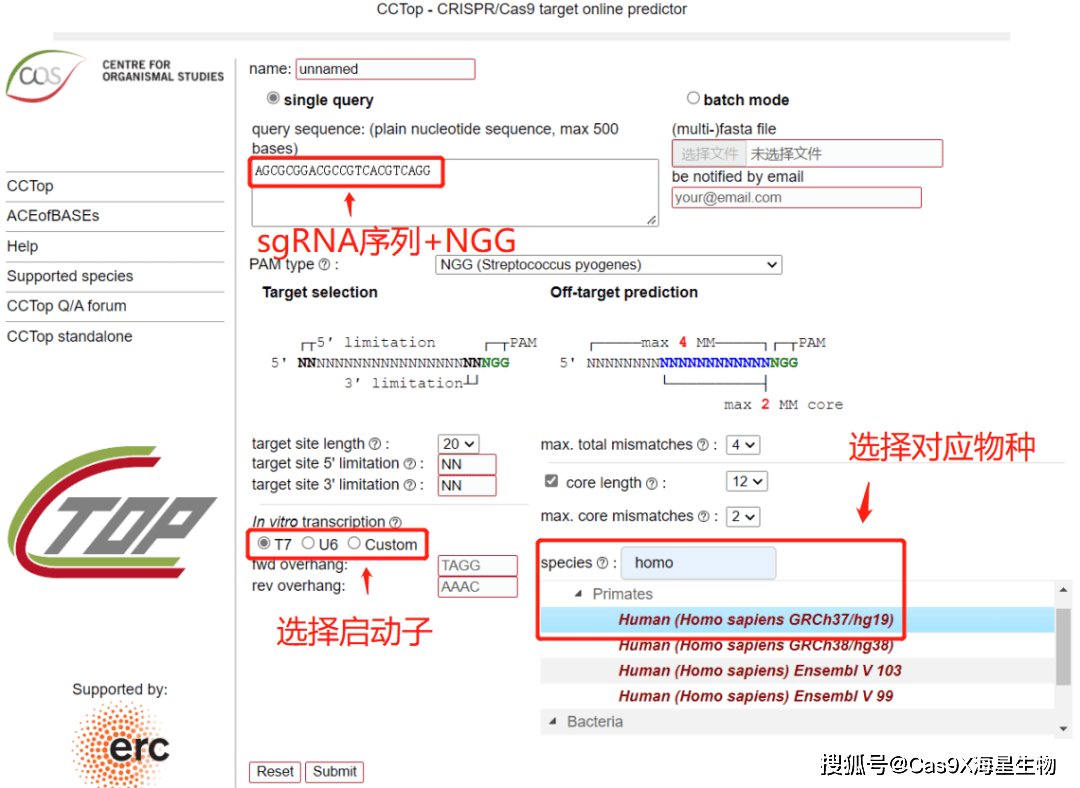 (图6)脱靶分析网站
(图6)脱靶分析网站
输入sgRNA+NGG序列和对应信息后,点击提交,得到如下结果图
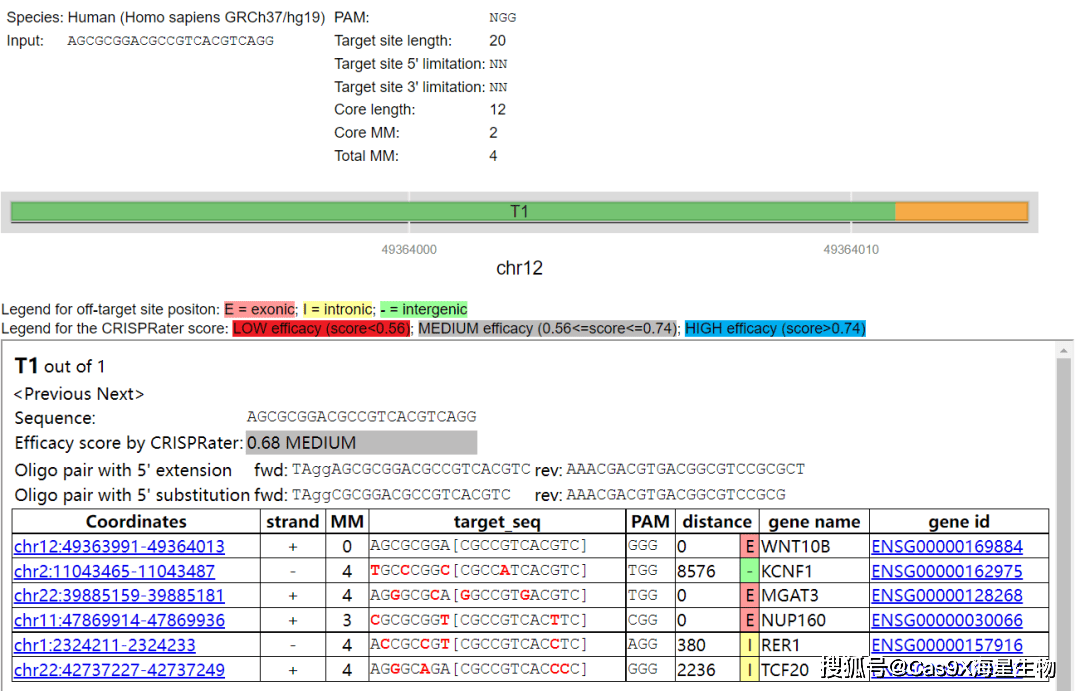 图7 脱靶分析结果
图7 脱靶分析结果
根据图中信息即可分析sgRNA的剪切效率,潜在脱靶位点信息等(图 7)。
除上文提到的脱靶效应分析网站外,之前提到的E-CRISP和CHOPCHOP这两个sgRNA设计网站也包含脱靶分析功能。
sgRNA在CRISPR-Cas9基因编辑系统中具有准确识别靶基因序列的作用,其效果可影响编辑的效率、是否发生脱靶等,甚至对最终基因编辑的效果产生决定性作用。因此,设计合理有效的sgRNA是我们实现基因编辑的重要基础。sgRNA包括20nt的靶向序列,crRNA,linker loop 和 tracrRNA几个部分共同组成(图1)。通常我们所说的sgRNA设计指的就是针对20nt的靶向序列进行设计,而选择合适的靶向序列就是sgRNA设计的核心工作。
- 本文固定链接: https://maimengkong.com/kyjc/1686.html
- 转载请注明: : 萌小白 2024年2月10日 于 卖萌控的博客 发表
- 百度已收录
