其中freebayes,bcftools,gatk都是把所有的snp细节都call出来了,可以看到下面这些软件的结果有的高达一百多万个snp,而一般文献都说外显子组测序可鉴定约8万个变异!
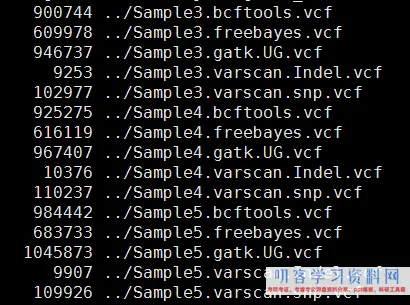
这样得到突变太多了,所以需要过滤。这里过滤的统一标准都是qual大于20,测序深度大于10。过滤之后的snp数量如下
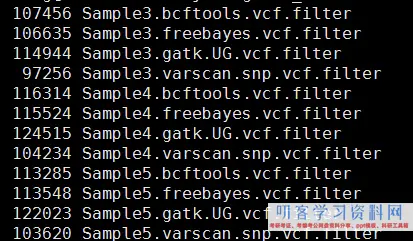
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$1″}’ Sample3.bcftools.vcf >Sample3.bcftools.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$1″}’ Sample4.bcftools.vcf >Sample4.bcftools.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$1″}’ Sample5.bcftools.vcf >Sample5.bcftools.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]”}’ Sample3.freebayes.vcf > Sample3.freebayes.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]”}’ Sample4.freebayes.vcf > Sample4.freebayes.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]”}’ Sample5.freebayes.vcf > Sample5.freebayes.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]”}’ Sample3.gatk.UG.vcf >Sample3.gatk.UG.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]”}’ Sample4.gatk.UG.vcf >Sample4.gatk.UG.vcf.filter
perl -alne ‘{next if $F[5]<20;/DP=(\\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]”}’ Sample5.gatk.UG.vcf >Sample5.gatk.UG.vcf.filter
perl -alne ‘{@tmp=split/:/,$F[9];next if $tmp[3]<10;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[3]”}’ Sample3.varscan.snp.vcf >Sample3.varscan.snp.vcf.filter
perl -alne ‘{@tmp=split/:/,$F[9];next if $tmp[3]<10;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[3]”}’ Sample4.varscan.snp.vcf >Sample4.varscan.snp.vcf.filter
perl -alne ‘{@tmp=split/:/,$F[9];next if $tmp[3]<10;print “$F[0]\\t$F[1]\\t$F[3]\\t$F[4]:$tmp[0]:$tmp[3]”}’ Sample5.varscan.snp.vcf >Sample5.varscan.snp.vcf.filter
这样不同工具产生的snp记录数就比较整齐了,我们先比较四种不同工具的call snp的情况,然后再比较三个人的区别。
然后写了一个程序把所有的snp合并起来比较
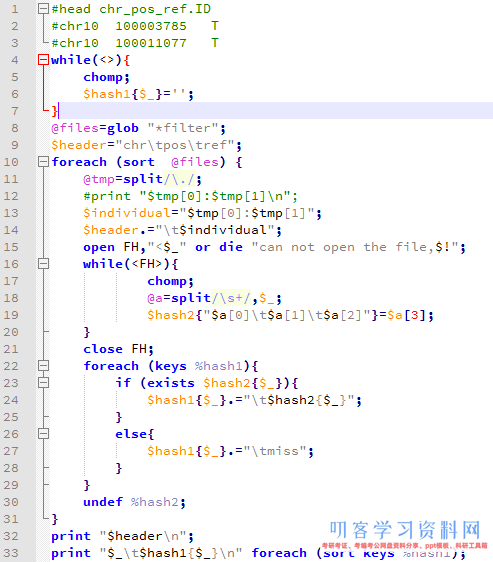
得到了一个很有趣的表格,我放在excel里面看了看 ,主要是要看生物学意义,但是我的生物学知识好多都忘了,得重新学习了 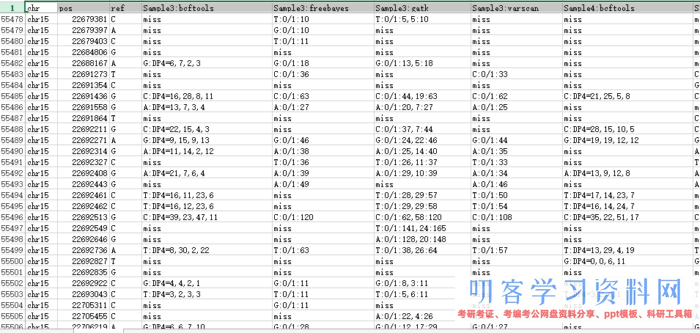
- 本文固定链接: https://maimengkong.com/zu/1398.html
- 转载请注明: : 萌小白 2023年3月5日 于 卖萌控的博客 发表
- 百度已收录
