摘自生信草堂
今天的文章中我给大家介绍环状RNA预测的另一款重要的软件——CIRCexplorer2. 该软件是由中国科学院上海生命科学研究院生物化学与细胞生物学研究所陈玲玲研究组和计算生物学研究所杨力研究组共同开发的,他们团队已经在《Cell》, 《Mol Cell》和《Genome Res》等国际顶尖杂志发表多篇环状RNA研究文章。
CIRCexplorer2继承了CIRCexplorer主要功能并增加了很多新的特性。它支持TopHat2/TopHat-Fusion, STAR, MapSplice, BWA and segemehl等多种RNA aligners, 并且能够精确地注释预测到环状RNA。更为重要的是,该工具可以侦测到多种环状RNA可变剪切事件,并且能够de nove组装环状RNA全长转录本。图1描绘了CIRCexplorer2的工作流程:
图1:The schematic diagram of CIRCexplorer2
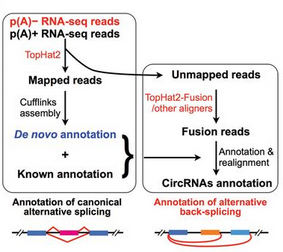
下面给大家介绍CIRCexplorer2的工作流程和命令行参数
-
安装
CIRCexplorer2是基于Python编写的,因此可以用pip install工具直接安装在本地服务器上。代码如下:
git clone https://github.com/YangLab/CIRCexplorer2.git
cd CIRCexplorer2
pip install -r requirements.txt
### install scipy according to http://www.scipy.org/install.html
python setup.py install
在CIRCexplorer2 documentation中还提供了几种可选择的安装方式,读者有兴趣的话可以参考:http://circexplorer2.readthedocs.io/en/latest/
Tophat-Fusion pair-end alignment
尽管CIRCexplorer2有自己的基因组比对模块(CIRCexplorer2 align),并且支持TopHat2/TopHat-Fusion,STAR, MapSplice等多种RNA aligner,但对于双端测序数据(pair-end sequencing reads)来说,只支持TopHat-Fusion。因此,我们只介绍基于TopHat-Fusion的比对数据。
$tophat --library-type fr-unstranded -o tophat_fusion_out -p 15 --fusion-search --keep-fasta-order
--bowtie1--no-coverage-search ${bowtie_index} read1.fq read2.fq
###TopHat参数介绍###
--library-type fr-unstranded :标准Illumina文库类型。
-o :结果输出目录,包含accepted_hits.bam等主要输出文件
-p :多线程
--fusion-search :允许fusion mapping
--keep-fasta-order :保持与基因组fasta文件相同的顺序
--bowtie1 :采用bowtie1算法进行比对
--no-coverage-search :不允许基于coverage的算法寻找junctions
2.Parsing for Circular RNA Fusion Junction Reads
$CIRCexplorer2 parse --pe -t TopHat-Fusion tophat_fusion/accepted_hits.bam –o circ_out 2> CIRCexplorer2_parse.log ### CIRCexplorer2 parse参数介绍###
--pe :Parse paired-end alignment file (only for TopHat-Fusion).
-t :指定aligner
3.Annotating for Circular RNAs
$ CIRCexplorer2 annotate -r hg19_ref_all.txt -g hg19.fa circ_out > CIRCexplorer2_annotate.log
### CIRCexplorer2 annotate参数介绍###
-r :基因注释文件
-g :基因组fasta文件
经过以上4个步奏,CIRCexplorer2会生成一个annotate目录,里面包括circ_fusion.txt文件,即是环状RNA注释文件,其内容包括以下几列:
|
Field |
Deion |
|
chrom |
Chromosome |
|
start |
Start of circular RNA |
|
end |
End of circular RNA |
|
name |
Circular RNA/Junction reads |
|
score |
Flag of fusion junction realignment |
|
strand |
+ or - for strand |
|
thickStart |
No meaning |
|
thickEnd |
No meaning |
|
itemRgb |
0,0,0 |
|
exonCount |
Number of exons |
|
exonSizes |
Exon sizes |
|
exonOffsets |
Exon offsets |
|
readNumber |
Number of junction reads |
|
circType |
Type of circular RNA |
|
geneName |
Name of gene |
|
isoformName |
Name of isoform |
|
index |
Index of exon or intron |
|
flankIntron |
Left intron/Right intron |
参考文献:
A combined strategy to identify circular RNAs (circRNAs and ciRNAs) (Zhang et al., Complementary Sequence-Mediated Exon Circularization, Cell (2014), 159:134-147)
Zhang XO*, Dong R*, Zhang Y*, Zhang JL, Luo Z, Zhang J, Chen LL#, Yang L#. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs. Genome Res, 2016, 26:1277-1287
生信草堂
浙大生信博士团队倾力打造的一个科研人员学习交流的公众微信平台。我们致力于科研社区服务,分享最前沿的科技进展,提供生信分析方法,解读经典分析案例,公众数据库的挖掘和临床数据统计分析。在此我们欢迎各位的加入!
- 本文固定链接: https://maimengkong.com/zu/1309.html
- 转载请注明: : 萌小白 2022年12月15日 于 卖萌控的博客 发表
- 百度已收录
