1. Enhancer 与 Super-enhancers
1.1. 增强子(Enhancer)
所谓增强子(Enhancer),位于结构基因附近,是一类非编码DNA顺式作用元件,在真核生物的发育过程中通过结合转录因子、辅因子以及染色质复合物作用于启动子,可以激活或增强基因的转录。简单说:增强子是能够增加启动子活性从而增加基因转录频率的DNA序列。
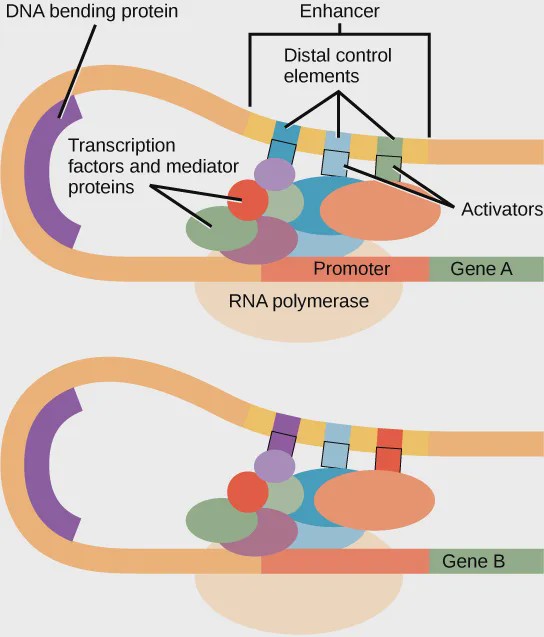

图1. 增强子及复合物结构示例图
增强子通常具有以下特点: ① 在转录起始点5'或3'侧均能起作用; ② 相对于启动子的任一指向均能起作用; ③ 发挥作用与受控基因的远近距离相对无关; ④ 对异源性启动子也能发挥作用; ⑤通常具有一些短的重复顺序。
增强子分为以下两种类型: ⑴ 细胞特异性增强子:能够在特定的细胞或特定的细胞发育阶段选择性调控基因转录表达的增强子称为细胞特异性增强子。例如,B细胞免疫球蛋白重链基因或轻链基因的增强子,只有在胚胎干细胞分化为B细胞时,才能对Ig基因起正调控作用。此外,α-类和β-类珠蛋白基因簇上游非编码区中均存在红细胞系特异性增强子。 ⑵ 诱导性增强子:在特定刺激因子的诱导下,才能发挥其增强基因转录活性的增强子称为诱导性增强子。如激素反应元件(HRE)及金属应答元件(MRE)
1.2. 超级增强子(Super-enhancers)
除了增强子是调控细胞基因时空表达关键的顺式作用元件外,2013年,Richard A. Young 实验室基于当时增强子的研究,提出了超级增强子(Super-enhancers, SEs)概念[1]。超级增强子是具有转录活性增强子的一个大簇, 富集高密度的关键转录因子(Master transcription factors)、辅因子(Cofactor)和增强子表观修饰标记(Histone modification marks)(见图1)。在功能上超级增强子能够驱动控制细胞身份基因的表达,可以用来解释细胞类型特异的表达模式,在发育生物学、癌症等疾病致病机理研究中显示出巨大的应用潜力[1-4]。胚胎干细胞中的多个转录因子(Oct4、Sox2、Nanog、Klf4、Esrrb、Nr5a2、Prdm14、Tcfcp2l1、Smad3、Stat3、Tcf3)富集在超级增强子上,在之前的研究中发现这些转录因子在胚胎干细胞中起着十分重要的作用。
Richard A. Young曾激动地预言道:“‘超级增强子’具有广阔的研发前景和价值,必将成为下一个药物研发的黄金靶点!” 因此开展肿瘤相关超级增强子的研究,将有助深入解开肿瘤发病机制,并且可用于指导抗肿瘤药物的高效研发,具有重要的社会意义和经济价值。

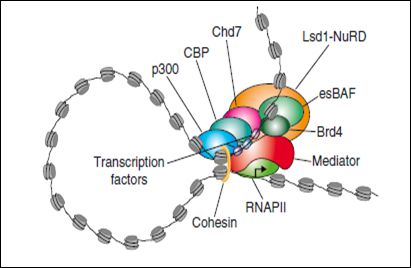
图2. 超级增强子及复合物结构示例图
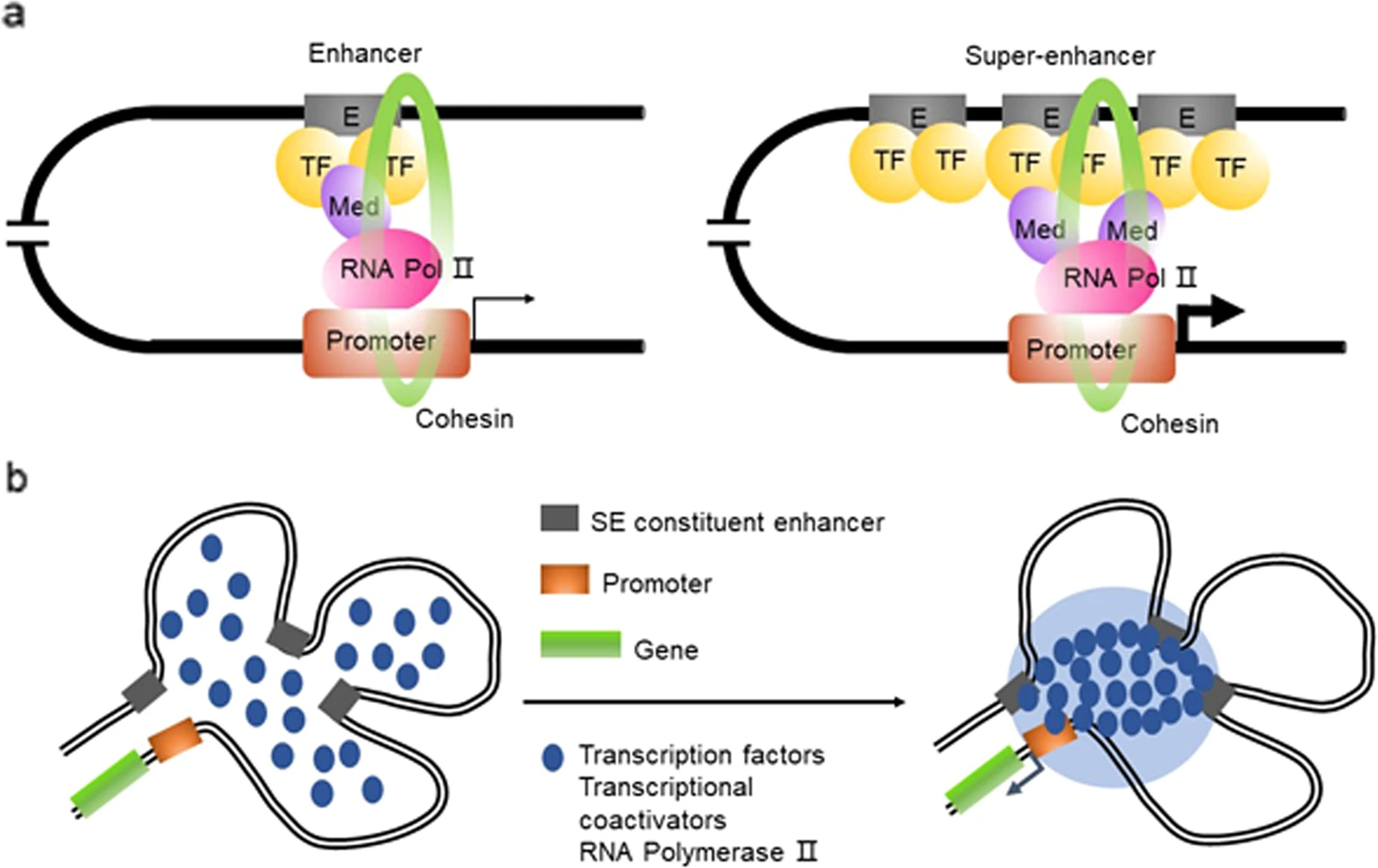
图3. 增强子与超级增强子及复合物结构对比示例图
-
(a) Enhancers and SEs are occupied by a high density of transcriptional regulators, including transcription factors, coactivators, and the RNA pol II complex.
-
(b) A phase separation model of SE activation. High-density interactions between transcriptional regulators form phase-separated multimolecular complexes at the SE locus, leading to the transcription of SE-driven genes.
2. 超级增强子的鉴定
目前对增强子鉴定,主要采用染色质免疫共沉淀技术(ChIP-seq) 针对活性增强子相关联的因子或组蛋白修饰进行检测,如转录因子、转录辅激活因子(如Mediator、p300)、组蛋白修饰H3K27ac 和H3K4me1等。活性增强子通常同时含有H3K27ac 和H3K4me1 修饰,而静态增强子(poised enhancer)一般同时具有H3K4me1 和H3K27me3 组蛋白标记[4-6]。在此基础上,超级增强子依据增强子转录活性标记分子结合水平强度的差异进行鉴定。在分析方法上,首先对所得增强子进行缝合。主要依据在基因组范围内,这些单个增强子实体间如在12.5 kb 范围内,则合并为单个实体,即缝合增强子(Stitched enhancer)。最后,确定超级增强子和普通增强子之间的阈值。缝合增强子和其余的单个增强子按照ChIP-seq所测信号水平的强度排序,绘制获得一张曲线图,该曲线上斜率为1 的切线的切点所得的信号值为区分超强增强子和普通增强子之间的阈值,高于该值为超级增强子,其余的则称为普通增强子(Typical enhancer)[1,2](见图2,图3)

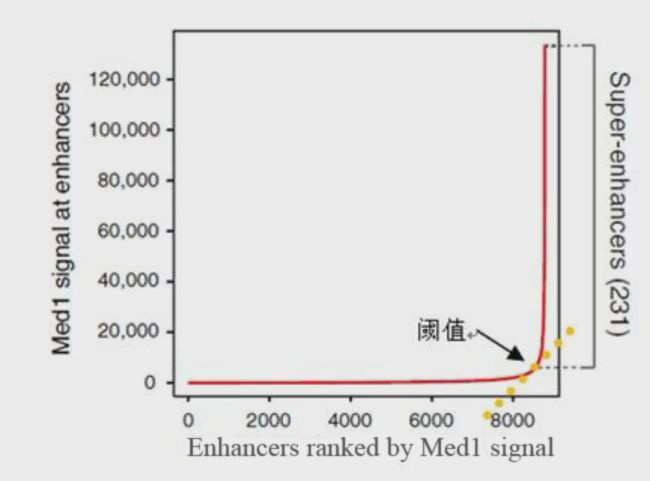
图2. 确定超级增强子和普通增强子之间的阈值[1]
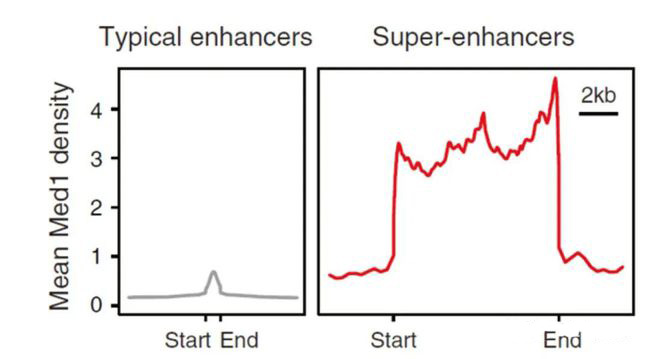

图3. 普通增强子与超级增强子示例图[3]
总结下来,识别增强子有以下几种方法:
-
(1) Chip-seq/Chip-chip识别转录因子结合位点,
-
(2) 增强子特异的因子也可用于识别增强子,如EP300,EP300 的结合位点常用于预测增强子;
-
(3) RNA 聚合酶 Ⅱ 能与数千个增强子结合,所以 RNA 聚合酶 Ⅱ 的最大亚基POLR2A 也可用于寻找增强子位点;
-
(4) 脱氧核糖核酸酶 Ⅰ 超敏位点 (DHS) 代表开放染色质区域,许多覆盖有增强子;
-
(5) 甲醛辅助分离调控元件 (FAIRE) 结合测序,能识别大量激活调控元件,包括增强子;
-
(6) 一些组蛋白修饰模式反应了不同的染色质状态,如H3K4me1 和 H3K27ac 的结合广泛用于增强子的标记;
-
(7) 增强子序列能够转录,转录的增强子RNA(eRNA) 也是激活增强子的标志;
-
(8) 染色体 3D 构象的方法 (e.g. 5C and ChIA-PET, Capture-C) 也能提供增强子 - 启动子相互作用的信息。
鉴定出超级增强子之后,可以根据基因位置预测超级增强子所调控的编码蛋白基因和非编码RNA的表达,通过结合转录组测序技术,可以对超级增强子和肿瘤异常高表达的mRNA、lncRNA、circRNA、miRNA进行关联分析,从而推断关键的超级增强子,进一步锁定肿瘤相关的基因,有利于指导下一步的抗肿瘤功能研究。
3. 其他资料
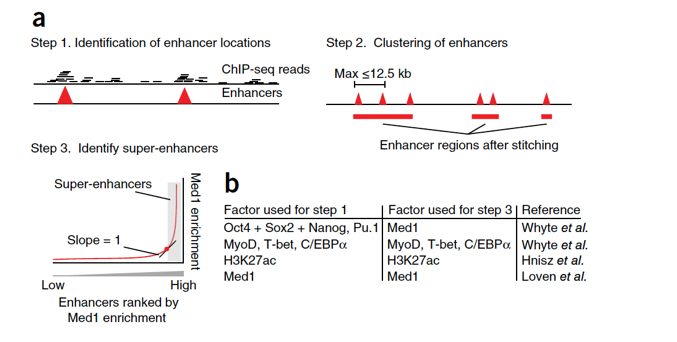

定义超级增强子。
-
(a) 说明了定义超级增强子的三个步骤。
-
步骤1:通过调用细胞类型特异性主转录因子的芯片序列数据来定义增强子基因座。
-
步骤2:将12.5kb范围内的增强子组合成缝合的增强子区域。下一步将使用缝合的增强子区域和12.5 kb范围内没有伙伴的增强子来识别超级增强子。
-
步骤3:计算每个增强子区域(缝合和单个增强子)的Med1的ChIP seq信号。所有增强子区域都是根据y轴上的Med1富集度沿x轴排列的。超级增强子定义为结果曲线拐点右侧的区域(以灰色突出显示)。
-
(b) 表中总结了步骤1和3中用于定义不同论文中的超级增强子的所有因子组合,表明在没有Med1芯片序列数据的情况下可以定义超级增强子。
一些案例
文献:Assessing eRNAs associated with a cytokine-sensing mammary super-enhancer | bioRxiv:
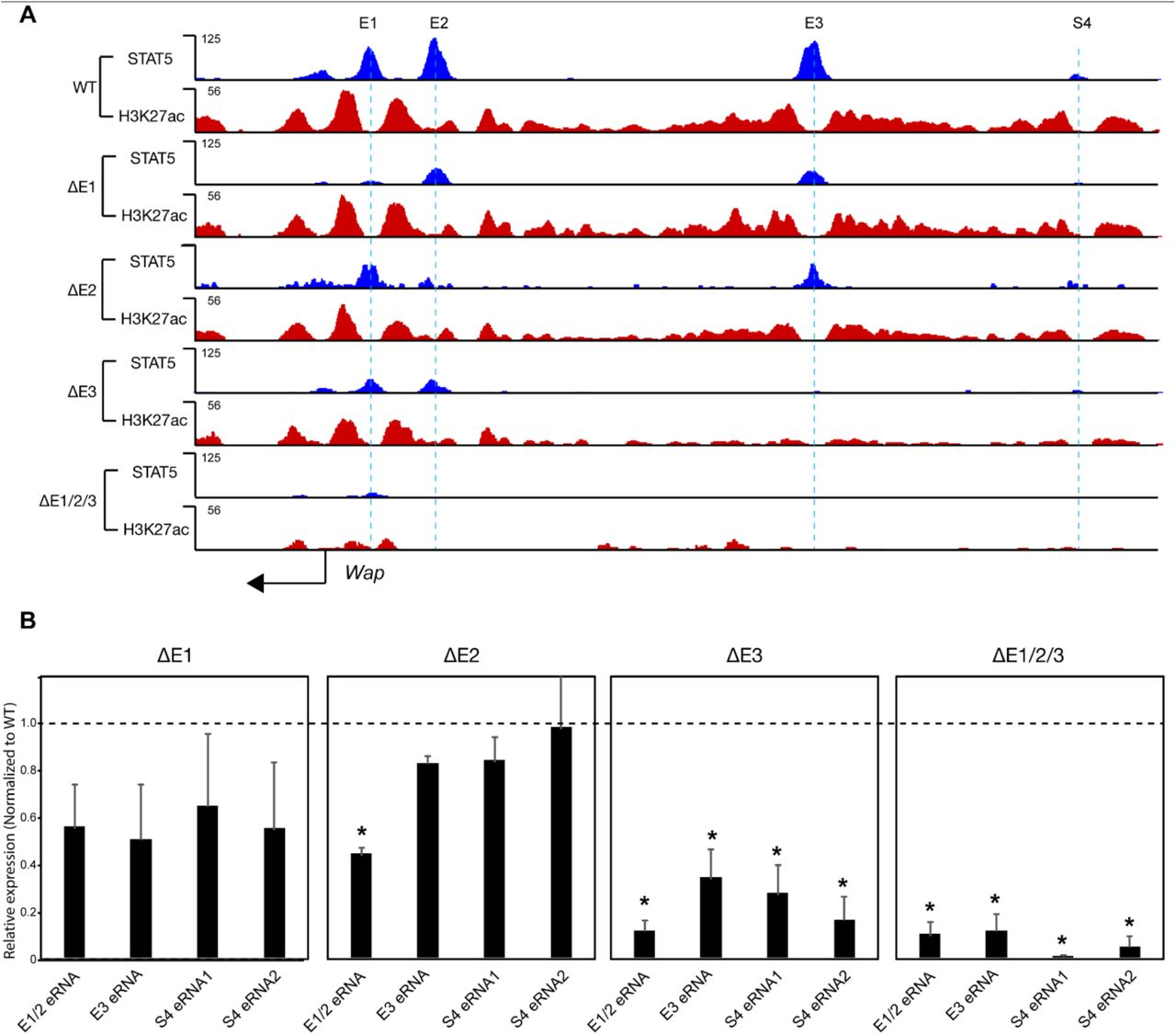
Wap超增强子组成元素中STAT5结合位点的删除导致STAT5结合,H3K27Ac和增强子转录的减少。
-
(A)携带单个或多个增强子缺失小鼠的L1乳腺组织中的ChIP-seq显示了增强子之间的协同作用,以有效募集STAT5。在位点消除了H3K27Ac所有三种增强子的丧失。
-
(B)通过Taqman测定法测量野生型和突变小鼠中的eRNA的表达
- 本文固定链接: https://maimengkong.com/zu/1282.html
- 转载请注明: : 萌小白 2022年11月19日 于 卖萌控的博客 发表
- 百度已收录
