在某一项研究中,我们通过转录组或者蛋白组学研究了干预组和对照组的表达谱,获得了数千个基因的变化信息,如何从这些表达谱中发掘有用的信息?初步的生物信息分析可能已经给我们提供了它们之间的差异倍数,P值等等。接下来我们可以使用在线工具对基因表达的分布进行分析。这里提供一个最近更新的简单又快捷,不需要安装软件的在线分析工具:WEB-based GEne SeT AnaLysis Toolkit。
0 1
首先点击网站
http://www.webgestalt.org/
如Figure1所示 第一行显示的是不同的分析类型,从左边起第一个是ORA (Over-representation analysis,过度表达分析 ) 检查符合选择标准的基因,并确定该列表中是否存在统计学上过度表达的基因集。第二个是我们最常用的GSEA (基因富集分析,Gene Set Enrichment Analysis)(红色圈圈)。GSEA用来确定先验定义的一组基因是否在两种生物学状态(例如表型)之间显示出统计学上显着而且一致的差异。第三个是NTA(Network Topology-based Analysis,基于网络拓扑的分析)。第四个是2019年新增加的功能磷酸化位点分析。
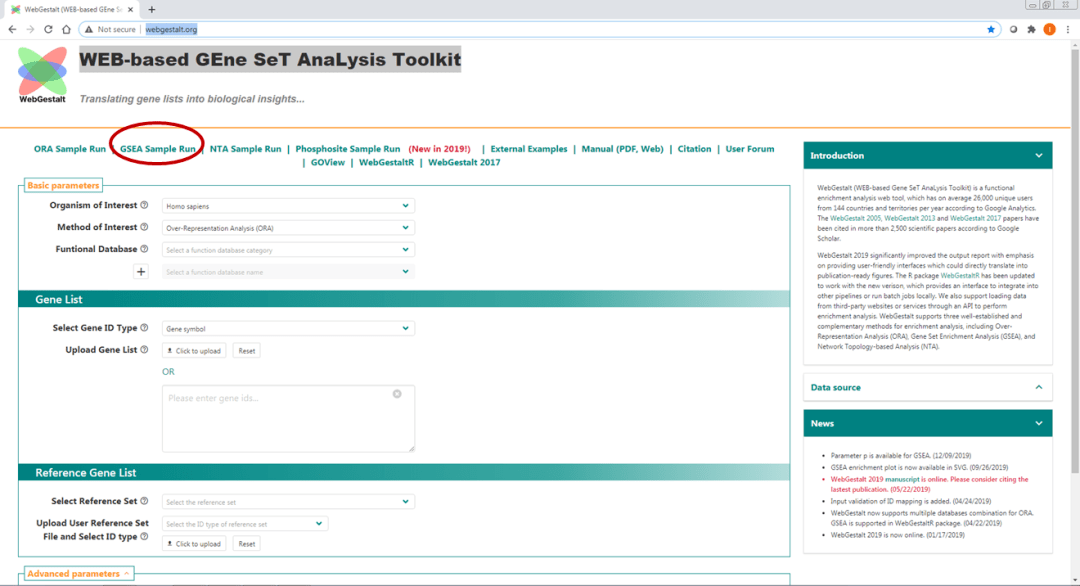
Figure1
0 2
我们首先看GSEA 。如Figure2所示, 首先在基本参数(basic parameters)中进行选择。第一行是物种,Homo Sapiens为人类,也可以在下拉菜单选择Mus (小鼠),Rat(大鼠)等。第二行是方法的选择,可以选择刚才介绍的ORA,GSEA,NTA。在这里我们选择GSEA为例。第三行是功能基因组数据库,可以选择Geneontology(GEO ),在第四行继续选择GEO下面的亚组,比如Biology process (生物过程),Cellular component(细胞定位),Molecular function(分子功能),在这里我们以Biology process为例。
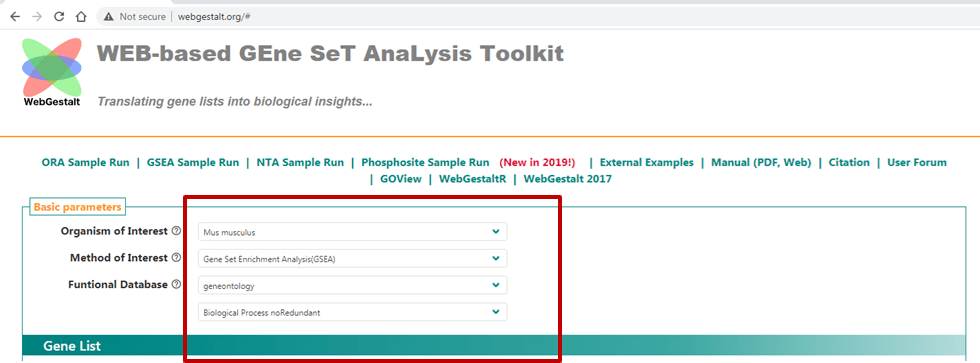
Figure 2
0 3
当然,您可以选择其他数据库,如Figure3所示, 比如在Funtional Database一行中选择pathway, 就可以在第四行进一步选择KEGG,Reactome,PANTHER等其他通路数据库。
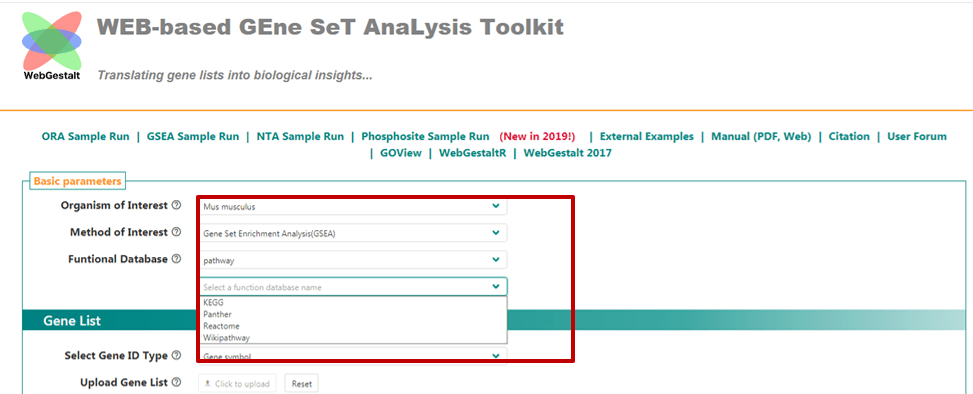
Figure 3
0 4
接下来就是输入基因名称了(Select Gene ID Type)。一般默认输入基因名称(gene symbol) , 如Figure4所示, 当然也可以从下拉菜单选择输入NCBI的基因ID等等。接下来可以上传基因名称文件,也可以从Excel拷贝数据到空白框中,作GSEA分析需要两列,第一列是基因名称,第二列是差异表达的倍数(log2FC)。
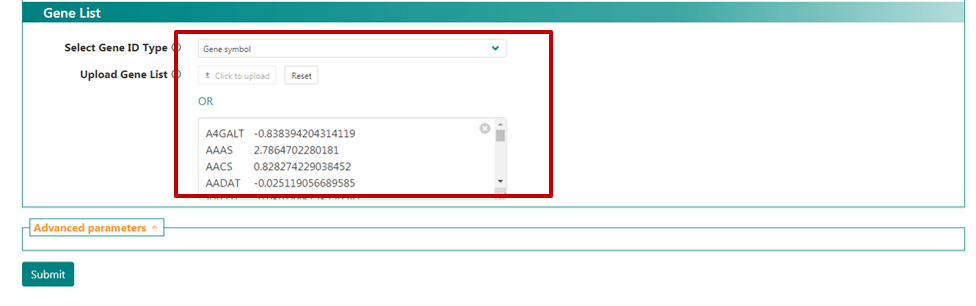
Figure4
0 5
接下来是高级参数。如Figure5所示 ,一般只需要修改Significance level(显著性差异), 可以选择显示有统计学差异的FDR值或者Top但不一定有统计学差异的。在这里我们选择FDR<0.05 。
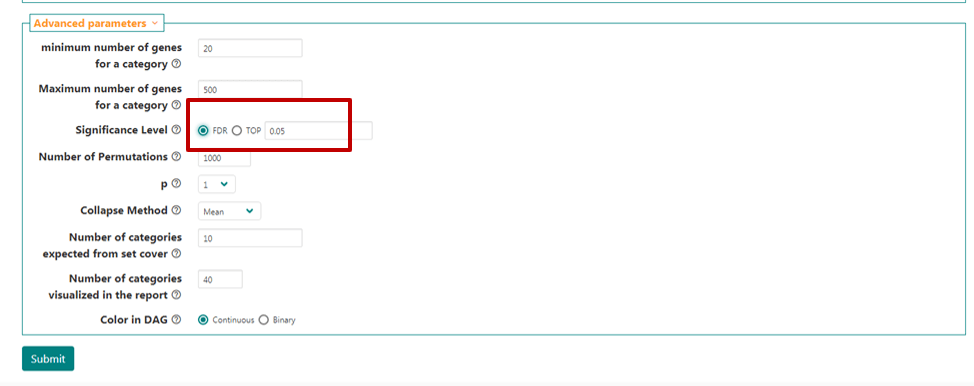
Figure2
0 6
点击提交,等待数分钟,就得到可以直接发表的图了。如Figure6所示 ,这里根据上述参数,显示了5个正相关类别和1个负相关类别。深色提示均为FDR<0.05. 点击右键,可以下载图片。
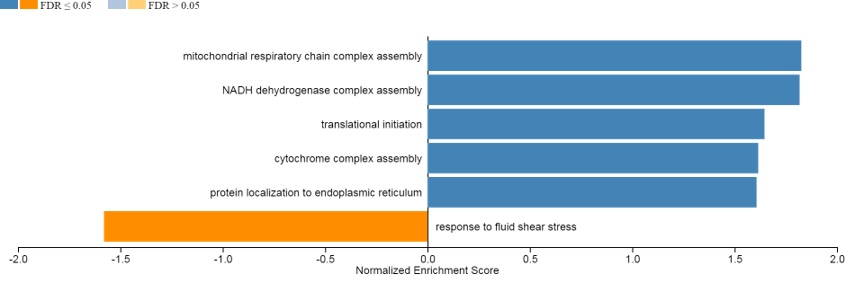
Figure6
0 7
点击某一个通路,可以看到对应的富集分析的图。比如第一个是线粒体呼吸链复合体装配,如Figure7所示 。
Figure7
0 8
下面更是具体显示了整条功能通路的得分,如Figure8所示, FDR,以及每个基因的得分情况。点击标题右边的下载按键可以将这条通路的具体基因以Excel的格式下载下来。
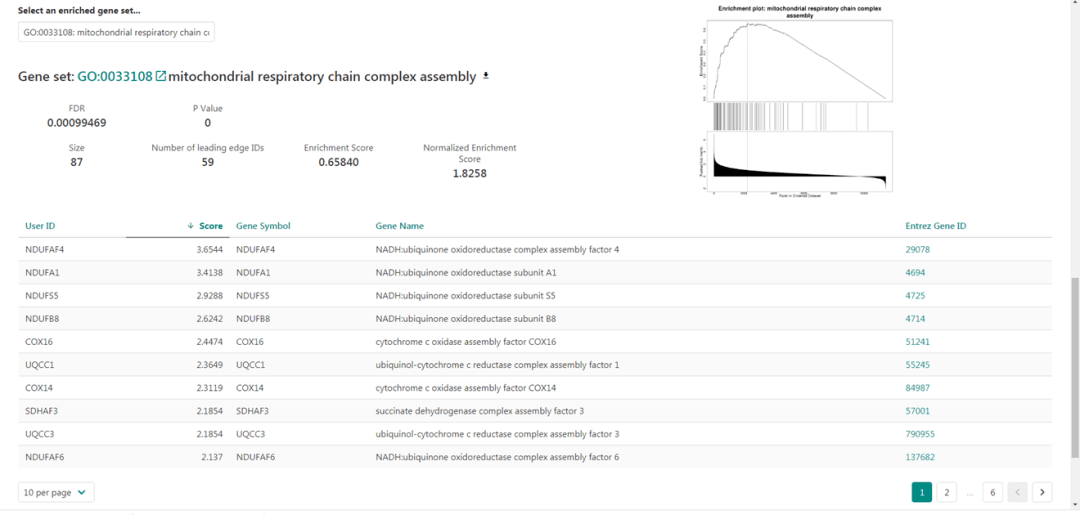
Figure8
怎么样,是不是5分钟搞定?如果您想进一步了解这个数据库的方法学,可以参考:
WebGestalt 2019: gene set analysis toolkit with revamped UIs and APIs .Nucleic Acids Research, Volume 47, Issue W1, 02 July 2019, Pages W199–W205,https://doi.org/10.1093/nar/gkz401
END
转自:医学方- 本文固定链接: https://maimengkong.com/zu/1251.html
- 转载请注明: : 萌小白 2022年10月6日 于 卖萌控的博客 发表
- 百度已收录
