ESPript:轻松绘制多序列比对结果,打造出版级Figure!
在上一期文章中,我们介绍了如何在线高效完成蛋白序列比对,并获得了 .fasta 或 .fa 格式的比对结果文件。本期,我们将通过 ESPript 工具,将这些比对结果转换成清晰、美观的图,让研究成果轻松达到出版级别。
什么是ESPript?
ESPript (Easy Sequencing in PostScript) 是一个专为学术研究者设计的在线工具,可以将序列比对结果可视化,并结合二级结构信息呈现,为序列相似性分析和发表提供强大支持。
ESPript 的主要特点包括:
-
高质量图表:支持序列比对和二级结构信息结合的可视化,结果清晰直观。 -
完全免费:针对科研和教育用途开放,用户无需安装软件,直接在线操作。 -
支持多种输入文件:可兼容 Clustal、FASTA、PIR 等常见格式,同时支持导入 PDB 文件标注结构信息。 -
高分辨率输出:支持 PDF 和 PNG 格式,方便插入到论文或报告中。
开发与维护团队:由法国 CNRS / 里昂大学的 Patrice GOUET 和 Xavier ROBERT 主导开发,当前版本为 ESPript 3.x。
ESPript 3.x 的官方网站是:https://espript.ibcp.fr/ESPript/ESPript/index.php
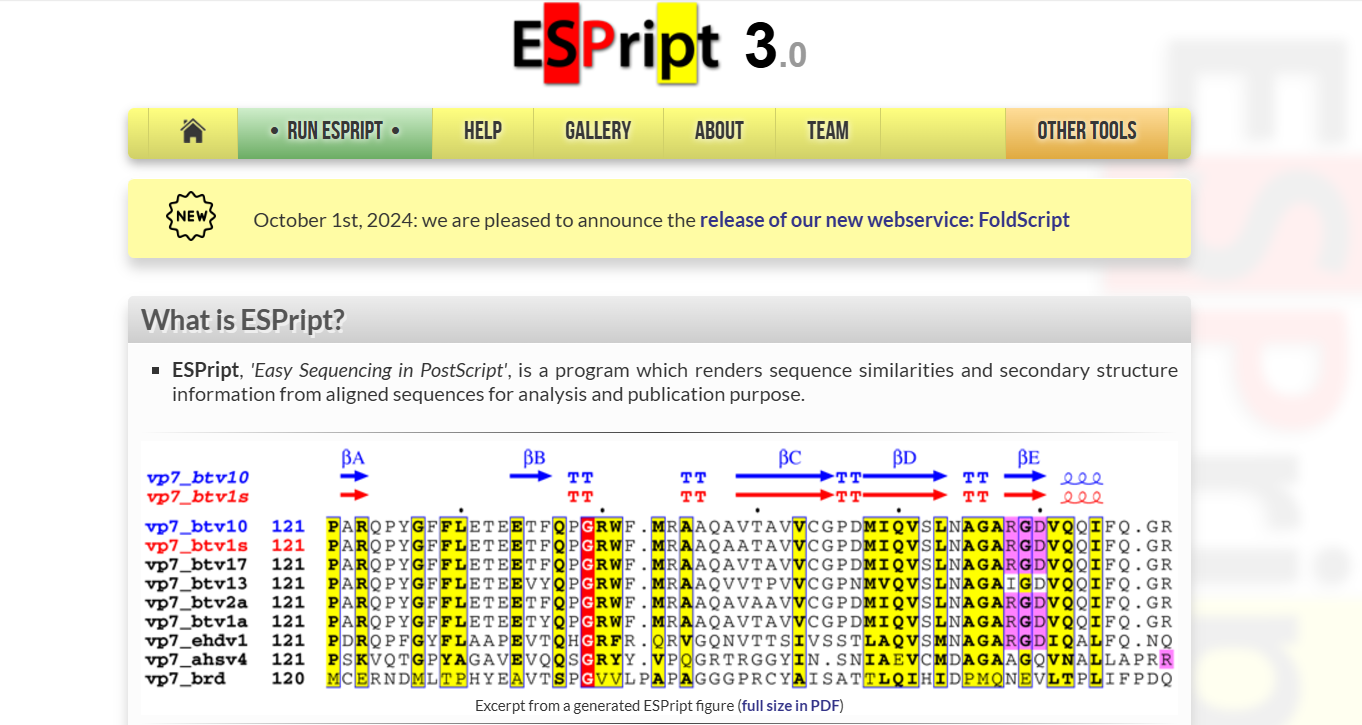
美化序列比对结果的详细步骤
1. 上传序列比对文件
-
访问 ESPript 官网,点击首页绿色按钮 “RUN ESPript”,进入文件上传界面。 -
点击红框中的 “选择文件” 按钮,上传上次获得的 T2R41蛋白的多物种序列比对结果文件。
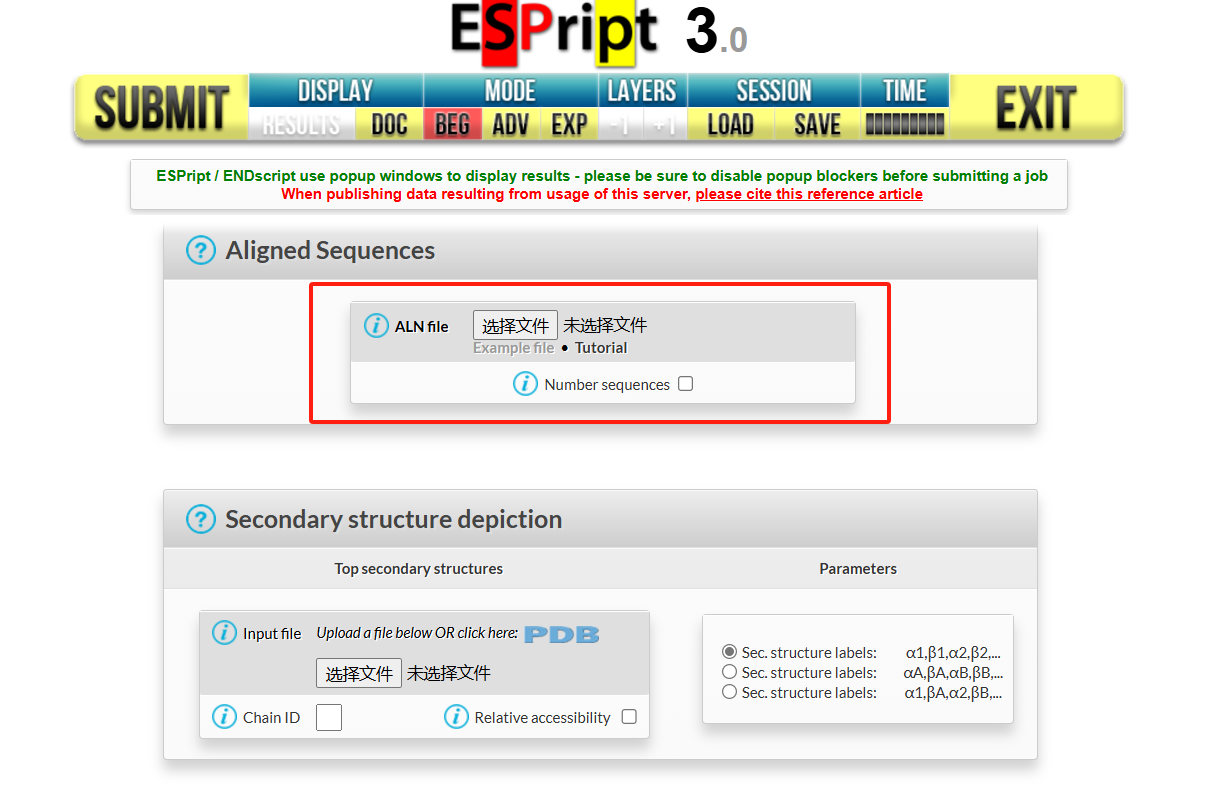
2. 设置比对图表参数
在设置界面中,按需调整以下参数:
(1)Alignments output layout
设置比对图的整体风格:
-
颜色模式:选择 B&W(黑灰白)配色,经典、专业,适合正式发表。 -
字体大小:默认值为 7,如果序列较长,可稍作调整,减小字体大小。 -
列数(Number of columns):决定每行显示的氨基酸数量,建议保持默认值,后续可以根据结果调整。
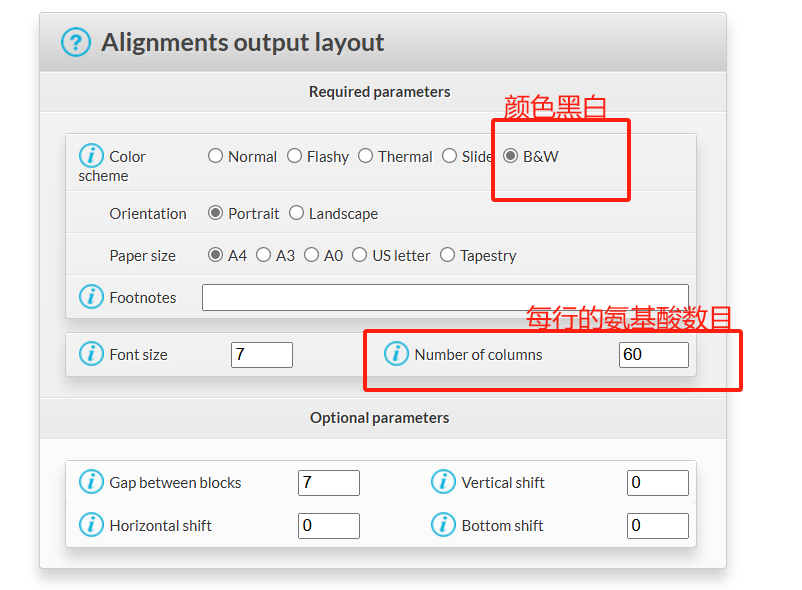
(2)Alignments output files
-
选项中可以选择输出文件格式,这里推荐默认选择 PDF 格式,方便生成高分辨率PDF文件。
3. 提交任务并生成结果
-
所有选项设置完成后,点击页面左上方 “SUBMIT” 按钮提交绘图任务:
提交后,页面会跳转到结果窗口,可在 “RESULTS” 板块找到绘图输出文件。
4. 下载比对图表
-
在结果页面中,点击 PDF 文件链接(如 pdf[15 Kb]),下载比对结果。以下是生成的图表示例:

结果中,如果某个位置氨基酸在物种间保守是黑色背景,白色字,我们可以看出我们比对的这个蛋白在目前选择的三个物种间并不是十分保守。
Tips: 如果初次结果显示序列保守性较低(如上图白色区域较多),可以调整比对的物种范围。例如,在哺乳动物中重新选择物种后生成的比对结果如下:

可以看到,T2R41蛋白在哺乳动物中深黑色区域更多,更加保守。
小结
使用 ESPript,可快速将蛋白序列比对结果转换成出版级别的专业图,并通过合理设置进一步优化输出效果。无论是论文发表还是会议展示,ESPript 都能为您的科研成果提供有力支持。
- 本文固定链接: https://maimengkong.com/kyjc/1838.html
- 转载请注明: : 萌小白 2024年11月17日 于 卖萌控的博客 发表
- 百度已收录
