一文教你快速完成多物种蛋白序列比对及进化树构建!
当我们研究一个蛋白时,探索其在不同物种间的保守性很关键。这不仅能帮助我们理解蛋白的功能和进化历史,还能为进一步实验设计提供线索,如保守性等。在本期文章中,我们将教你如何通过Uniprot网站,快速完成多物种蛋白序列比对和进化树构建。无需下载任何工具,全程在线完成,简单高效!
步骤一:打开Uniprot并检索目标蛋白
打开uniprot网站:https://www.uniprot.org/
输入你的目标蛋白名称。例如,我们以 T2R41 为例。直接点击 Search 按钮开始检索。
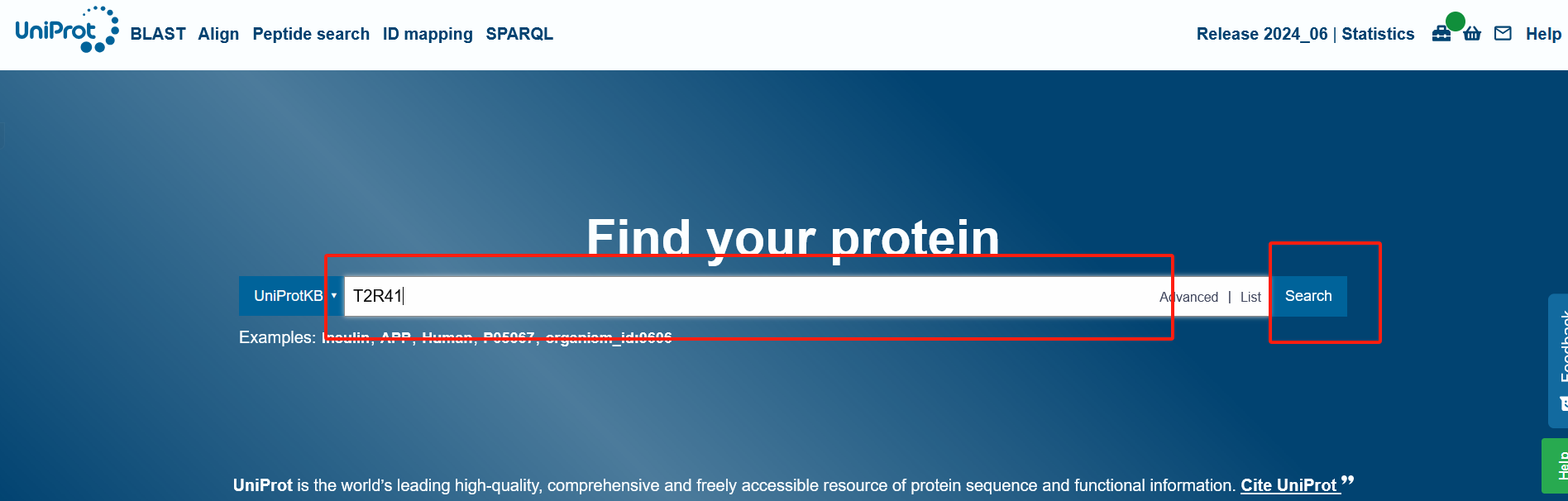
步骤二:选择物种
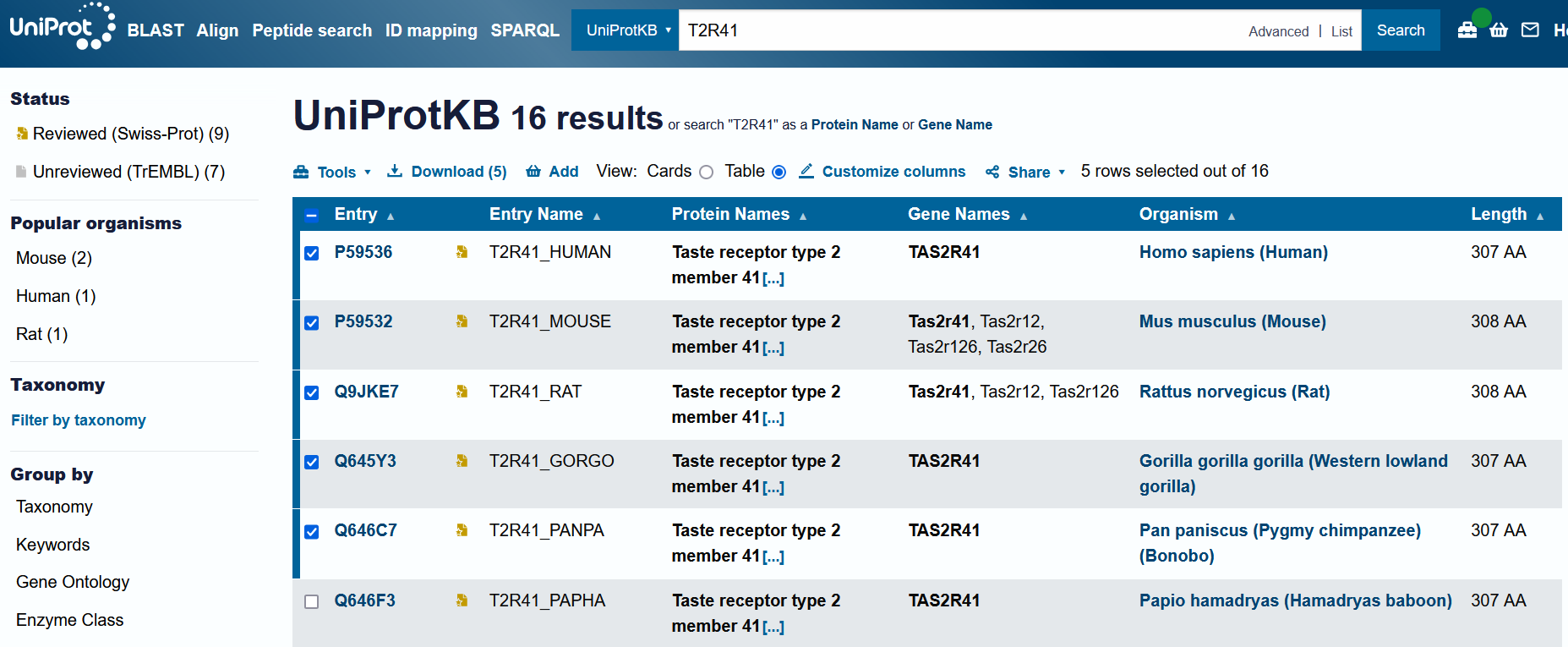
Uniprot会返回多个物种中T2R41的蛋白序列信息。你可以看到人类、小鼠、大鼠、猩猩等多个物种的蛋白数据。
我们选择5种物种进行分析,如下图:
步骤三:进入Align工具
在Tools中选择Align工具并点击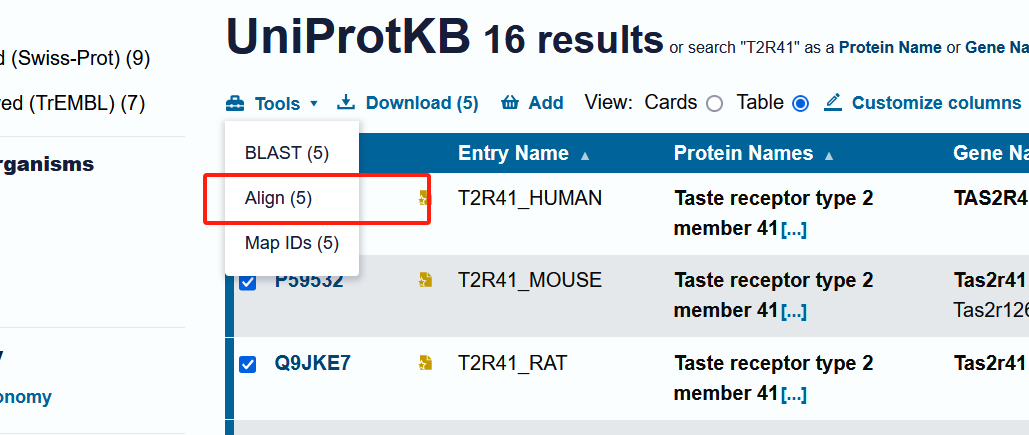
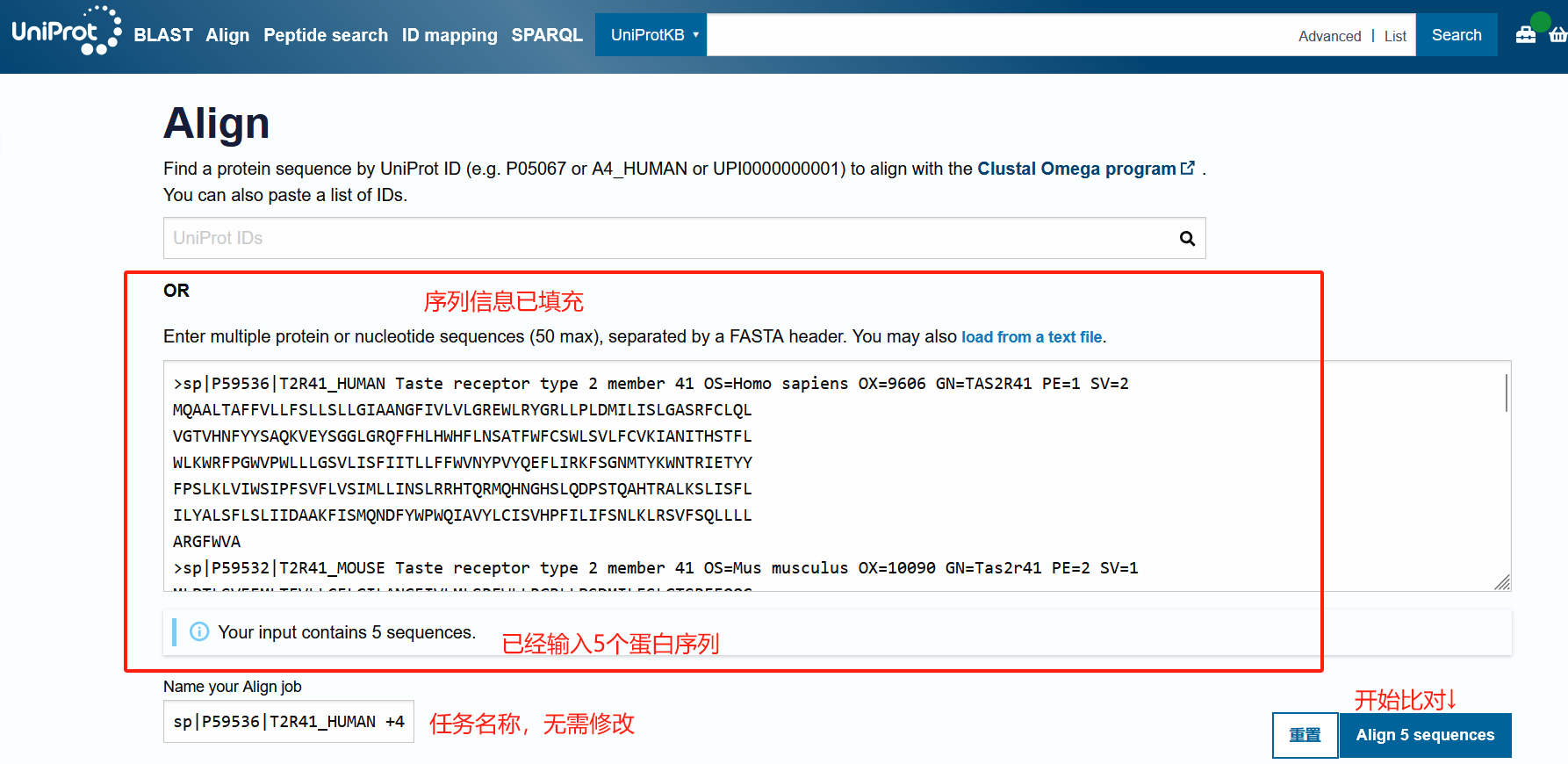
进入比对界面后,Uniprot会自动填入你选择的5条蛋白序列。你可以检查是否全部正确,同时可以在序列名称前的>号后自定义名称,方便后续结果的展示更加直观。
确认无误后,点击 Align 5 sequences,进入比对任务提交。
步骤四:查看比对结果
任务完成后,会显示Completed,点击页面中的蓝色链接进入结果页面。 
进入比对界面后,可以看到比对结果,比对结果页面非常直观,可以看到这几种物种间蛋白序列的保守性。某些氨基酸位点在所有物种中完全一致,这提示它们可能具有重要的功能作用。
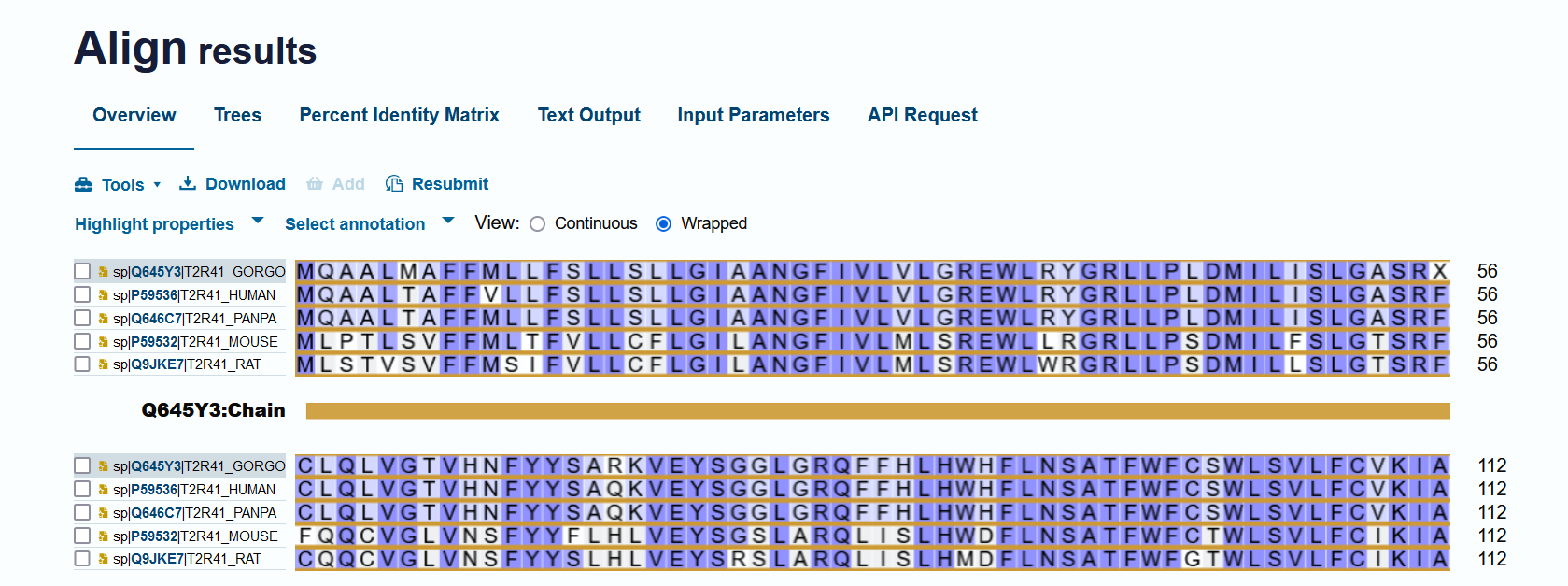
步骤五:探索进化树和其他信息
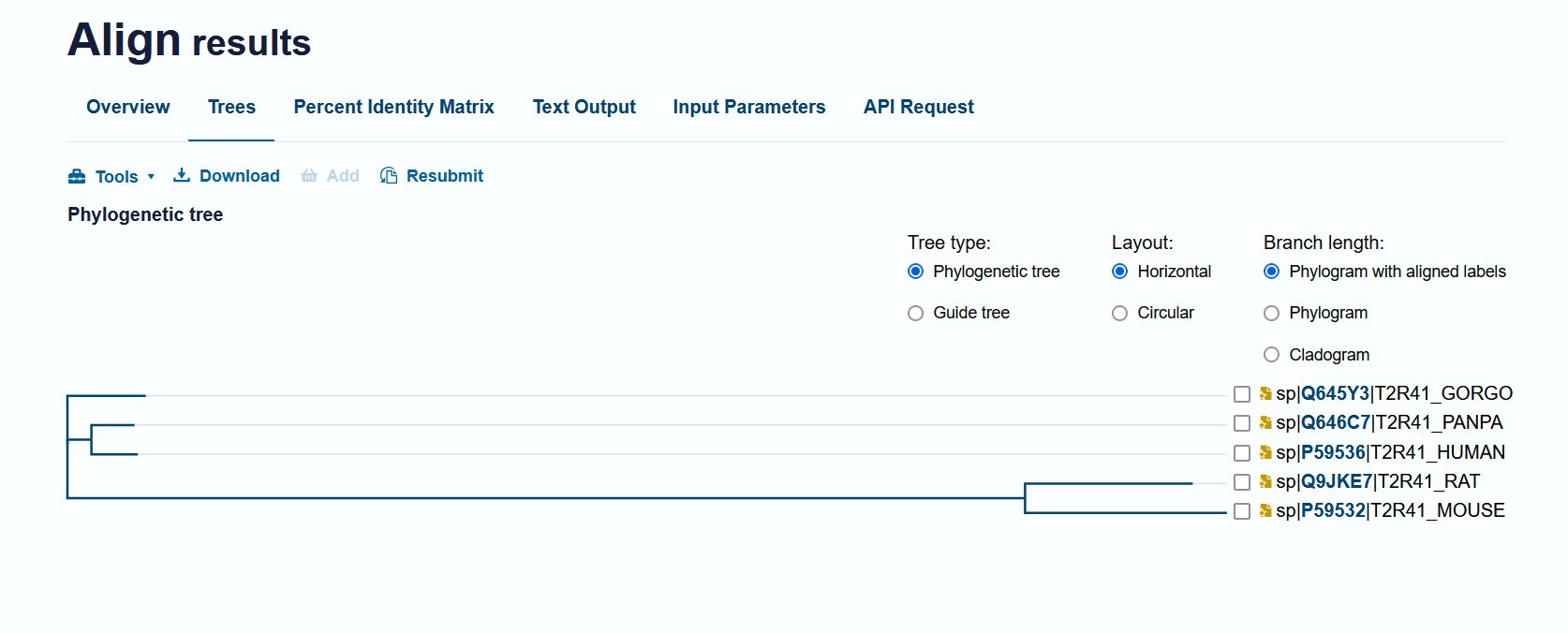
除了序列比对结果外,Uniprot还提供了进化树、Percent Identity Matrix、运行参数等结果和信息:
-
进化树:直观展示蛋白在不同物种间的进化关系。
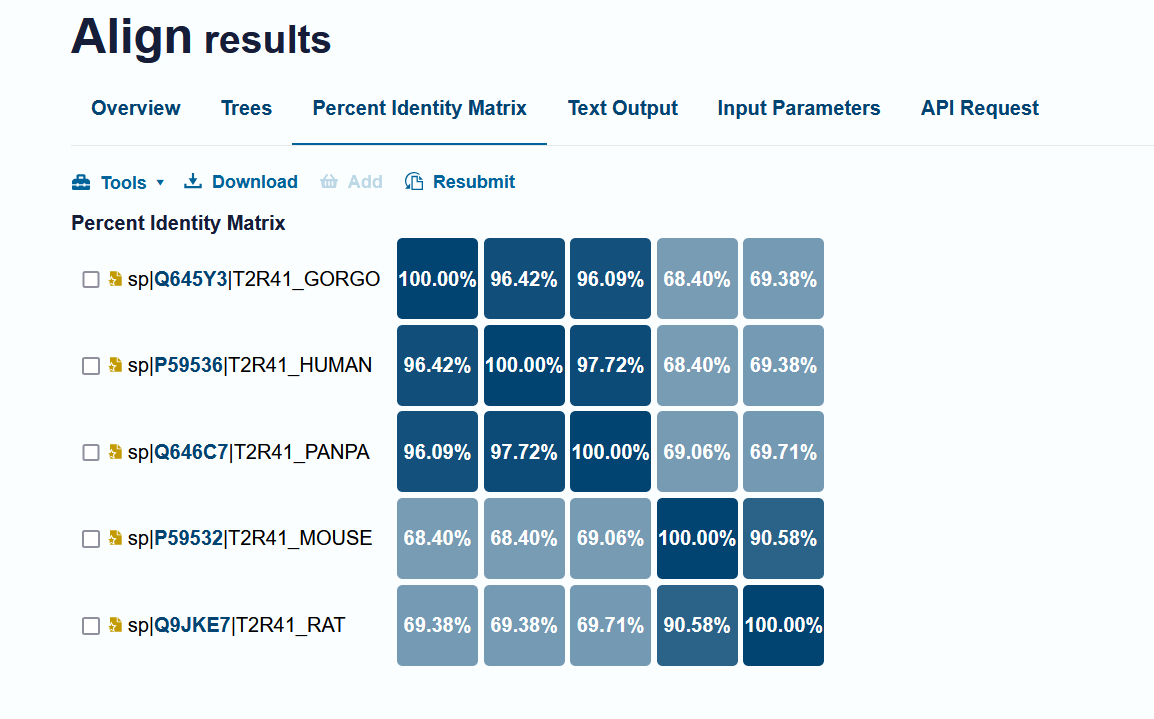
2.Percent Identity Matrix:显示物种间蛋白序列的相似性百分比,便于快速比较。
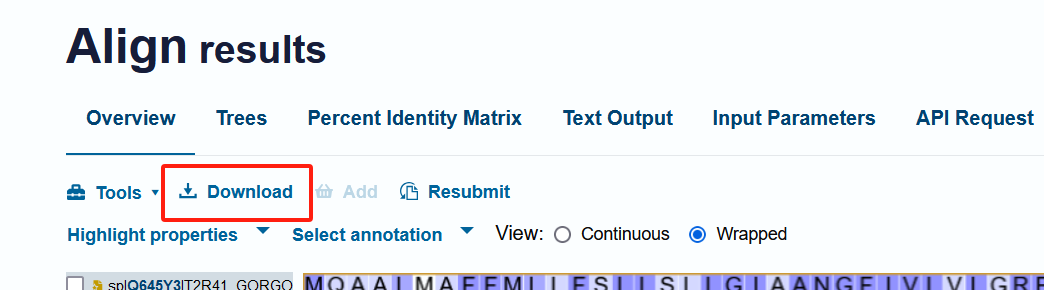
步骤六:下载比对结果
Uniprot支持将比对结果下载到本地,方便保存和后续分析。点击 Download 按钮,会弹出多种下载选项。
下载选项提供了多样的下载选项,可以下载FASTA比对结果序列。
小结
通过Uniprot网站,我们可以快速完成多物种蛋白序列比对,不仅能直观了解目标蛋白的保守性,还能获得进化树和相关分析信息。这种方法操作简便,特别适合希望快速获取比对结果的科研人员。
如果你对比对结果的美化和高质量呈现感兴趣,请持续关注我们。在下一期,我们将深入讲解如何使用专业工具调整序列比对结果,使其符合SCI文章发表的标准,敬请期待!
如果你觉得这篇文章对你的研究有帮助,欢迎点赞、分享,并在评论区留言你的研究心得!
- 本文固定链接: https://maimengkong.com/kyjc/1837.html
- 转载请注明: : 萌小白 2024年11月15日 于 卖萌控的博客 发表
- 百度已收录
