各位医学方的朋友,大家好,我是Flyman,今天给大家分享一篇WGCNA的文章,WGCNA不可否认在样本数目较多的情况下,应用越来越多,特别是基于WGCNA进行biomarker筛选,这篇文章是17年发表在Experimental Eye Research((IF:3.152)杂志上。
今天,我们主要从数据集的搜集与前处理、网络构建、模块识别、功能富集以及hub基因挑选5个方面来解读这篇文章。
01
数据集的搜集、前处理
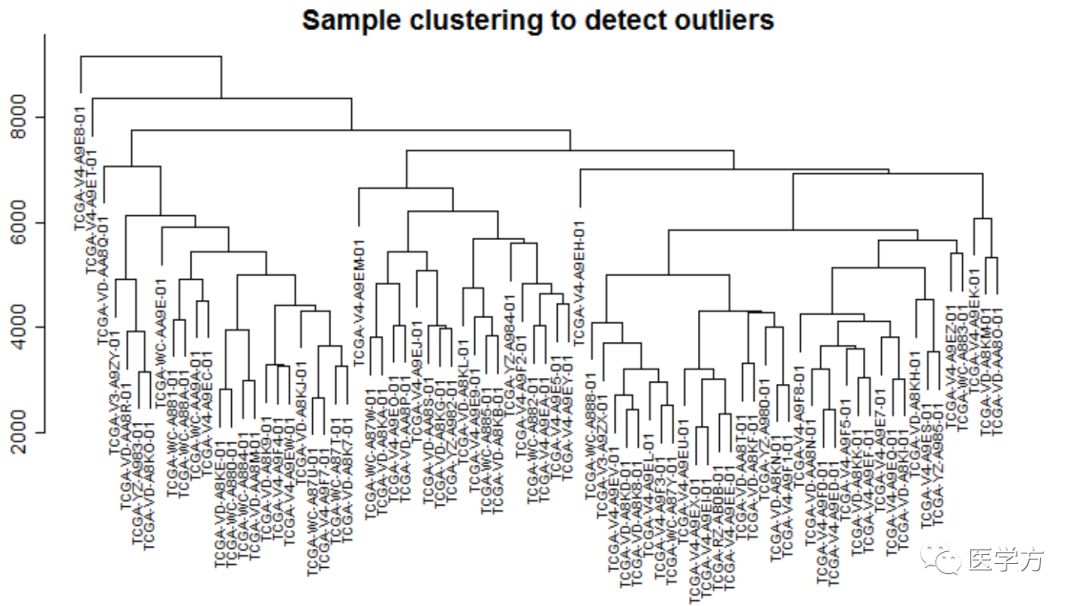
这篇文章是讲述的是葡萄膜黑素瘤,数据源作者是从TCGA官网数据库上下载获得的,对于多个探针对应一个基因时,作者取的是平均表达量,接着作者使用flastClust工具进行样本聚类分析。
02
加权基因共表达网络的构建
作者并没有筛选差异表达,而是对所有的基因进行网络构建,基于TCGA数据库,作者首先需要筛选一个合适的power值,作者从pwer1-20之间选择了最合适的一个power=6,并设置了最小的module的基因数目为40,一般情况下默认为30,所以大家可以根据自己的结果好坏进行设置,只需要文章中写明即可。

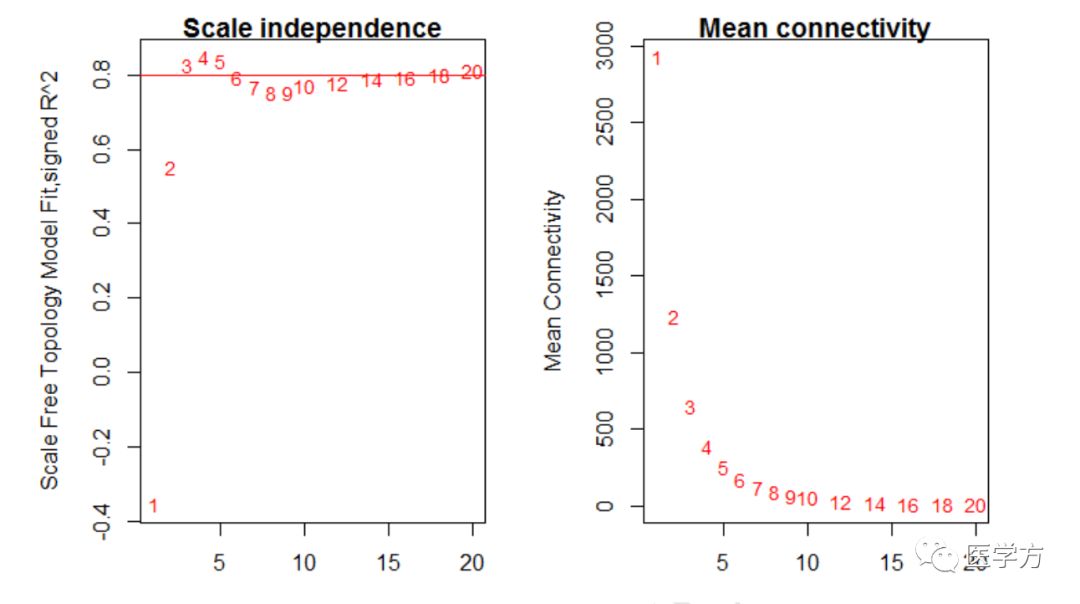
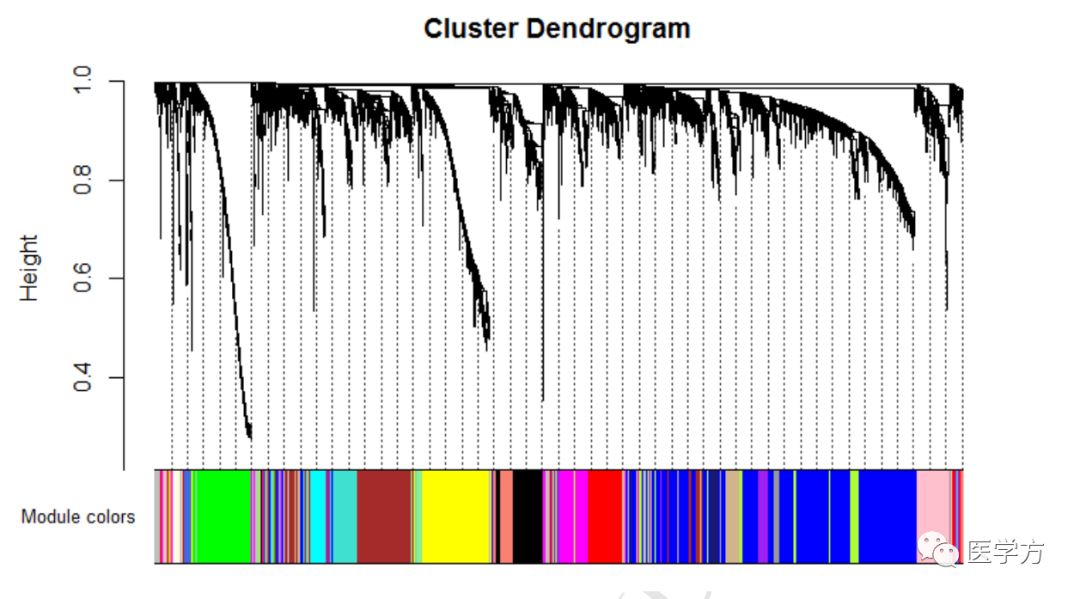
接着作者选择了合适的β值为6,进行下一步研究分析,一共识别出21个模块
0 3
识别临床特征显著相关的模块及功能分析
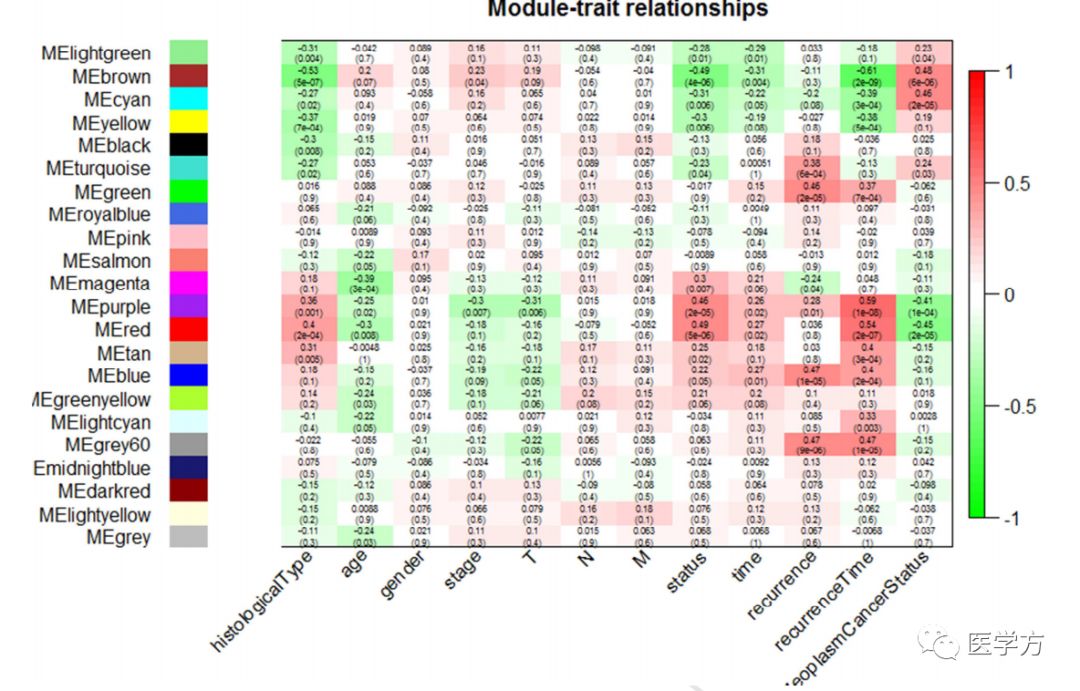
作者选择了年龄,性别,stage,TNM分期,生存状态,生存时间等临床因素用于模块的构建和识别,作者重点关注了生存状态,癌症复发与否,复发时间因素,发现存在四个模块与之关联性最高分别是红色,紫色,绿色以及品红色模块关联度最高,同时绘制了这四个模块的散点图,结果如下:
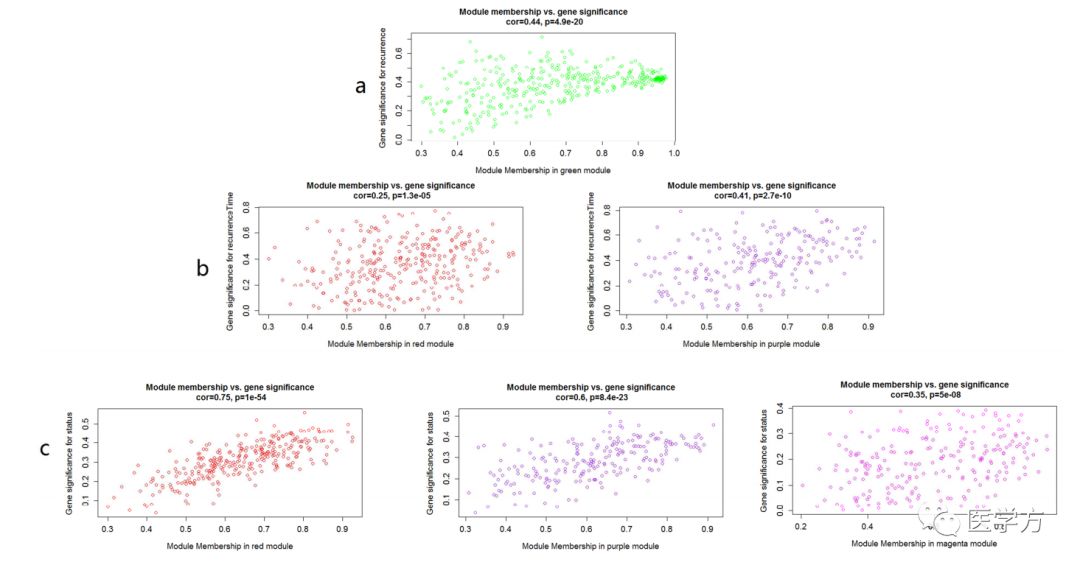
0 4
功能富集分析
作者接着对这四个模块进行了富集分析,发现绿色模块参与到cell projection,neurological system process等,品红模块主要参与到基因合成组装的过程,紫色模块主要参与到tissue homeostasis,红色模块主要参与到metastasis of cell等,如下:
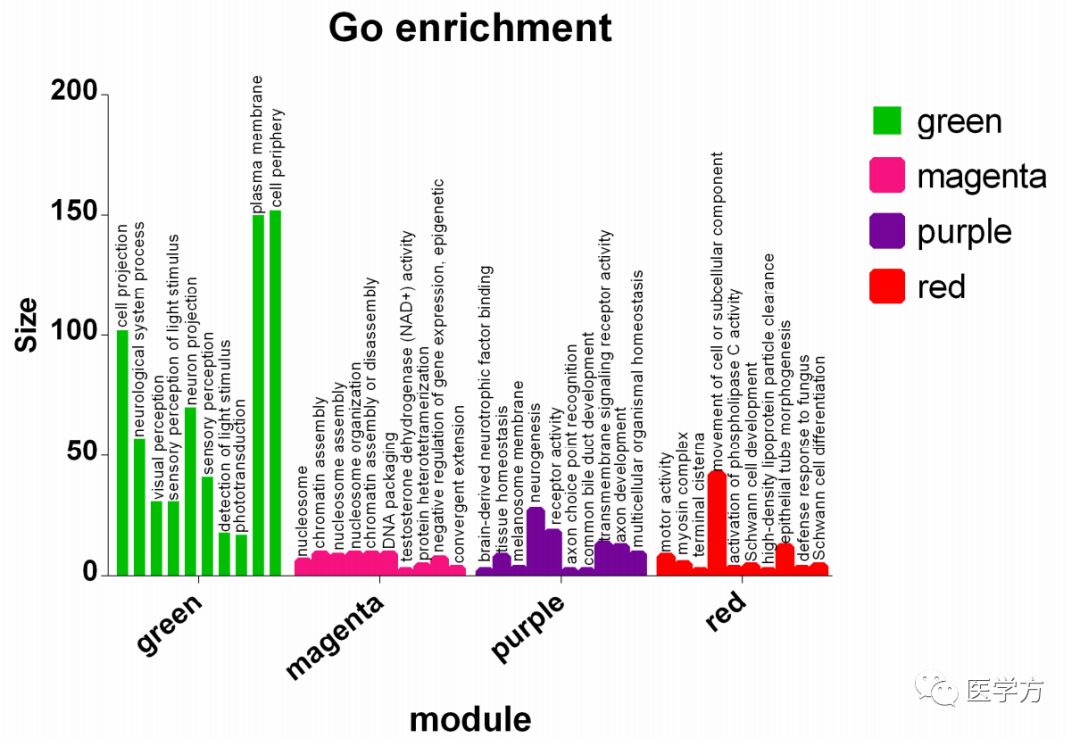
0 5
模块可视化及hub gene筛选
作者借助Cytoscape软件进行网络可视化,作者不是采用传统的MM和GS来筛选hub gene 而是采用内部连通性的概念来筛选,通过计算每一个基因的连通性的综合,按着值从高到低排序,连通性越高的基因则为hub gene来进行筛选:
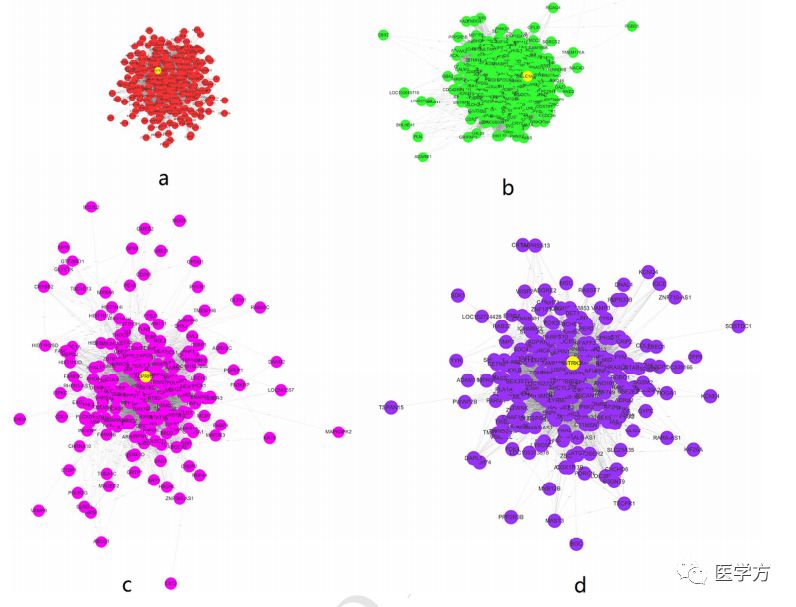

至此整篇文章就结束了,最后我们总结一下这篇文章的流程如下:
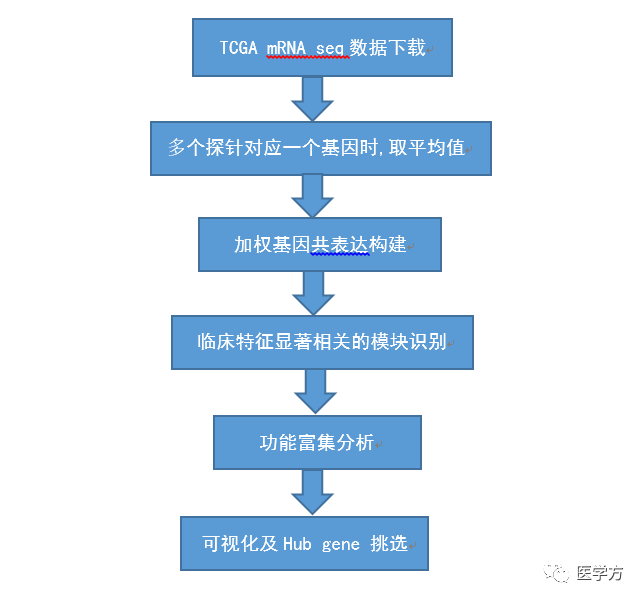
这篇文章思路非常清晰,大家是不是看的有点小激动?那就赶快去试一下吧!另外,我们医学方也推出了关于WGCNA的课程,欢迎大家关注学习。
- 本文固定链接: https://maimengkong.com/zu/925.html
- 转载请注明: : 萌小白 2022年5月18日 于 卖萌控的博客 发表
- 百度已收录
