
5月10日,《Plos One》杂志(最新影响因子: 3.057)在线发表了派森诺与上海交通大学合作的简化基因组+重测序项目的一篇文章。下面,小编就来分享一下该文章的内容。
研究背景
多趾,即趾头数目过多,是脊椎动物中常见的一种遗传性肢体异常。鸡常见趾型为四趾,多趾畸形一般为额外多一个拇趾在一边或两边脚趾。
研究目的
前人利用混池测序性状定位(BSA)、连锁分析和全基因组关联分析(GWAS)等方法,已经定位到控制该性状的区域位于2号染色体上。然而,多趾性状的详细的遗传模式和控制该性状的具体的基因信息还不是特别清楚。本研究旨在鉴定控制鸡多趾性状的基因,并揭示多趾性状的遗传模式。
研究方法
首先,利用简化基因组测序的方法对多趾进行GWAS性状定位。然后,结合重测序的方法进一步缩小候选基因范围。
研究结果
1. 北京油鸡多趾性状的系统性分类
鸡多趾性状总体可以分为两类,即额外趾的复制和多趾节。Warren进一步规范了分类标准,将鸡的多趾分为5类(A、B、C、D、E型)。本研究发现,北京油鸡多趾类型丰富,主要是B和D型。而且除了Warren描述的所有类型,还发现了新的类型。

北京油鸡中多趾的主要类型
2. 多趾性状的遗传定位
北京油鸡是中国优良的肉蛋兼用型地方品种,约25~31%具有多趾现象。因此本研究采用北京油鸡作为研究对象,构建了北京油鸡和山岐杂鸡(正常趾)的杂交群体,挑选了11个家系中的112个个体,利用简化基因组的方法做基因分型。一共得到约70万个tag,每个tag的平均测序深度为11X,得到~7.6万个 SNP。进一步过滤后,~4.7万个SNP用于全基因组关联分析。
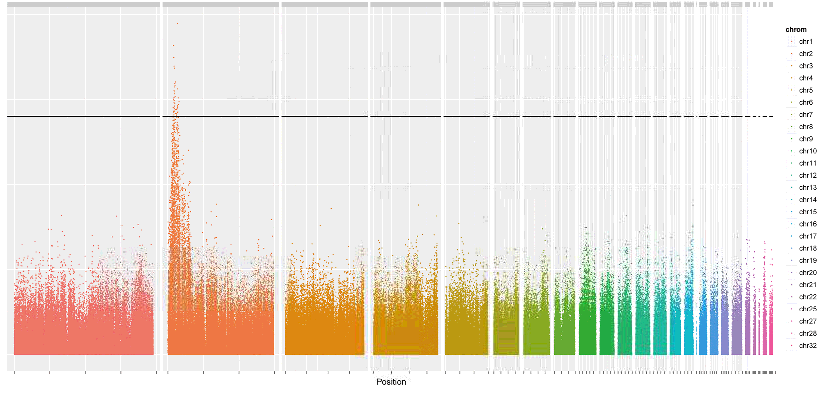
全基因组关联分析的manhattan plot
全基因组关联分析定位到2号染色体一个区段,其候选基因如下表。
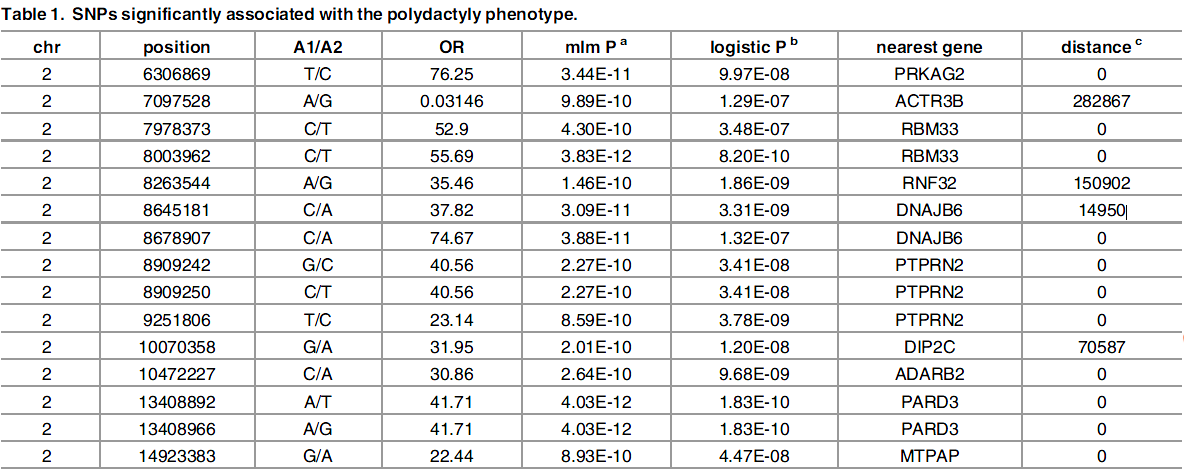
全基因关联分析定位到的与多趾相关联的SNP及其候选基因
3. 重测序鉴定多趾与正常个体之间的受选择区域
对3个多趾个体和3个正常趾个体进行20x重测序,鉴定到9.9百万个SNP。作者进一步对这些SNP分类,寻找在两个群体间是相反纯合基因型的位点,这些位点可能与多趾性状相关。一共鉴定到6079个这样的SNP位点。利用这些SNP数据,以100 Kb为窗口长度,步长为10 Kb的滑动窗口方法,计算群体分化指数Fst,寻找全基因组范围内两群体间的受选择区域。结果发现the limb region 1 homolog (LMBR1)同时位于GWAS定位和受选择区域内,更为值得关注。其中,位于该基因5号内含子中的rs80659072在这两个表型不同的群体中是相反的纯合基因型。
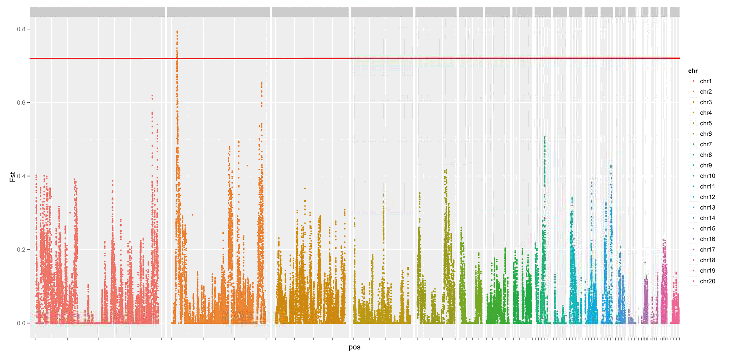
多趾与正常趾群体间分化指数Fst分布图
4. 最后,对于在以上重测序得到的在多趾和正常趾群体中具有相反纯合基因型的SNP位点,作者挑选了一些有意义的位点,在另外的136个个体中利用Sanger测序进行了验证。结果显示rs80659072在这136个个体中的多趾与正常趾群体中基因型有显著差别。这提示该SNP位点可能与多趾性状强烈相关,这与本研究GWAS和重测序的结果一致。

总结:
本研究主要利用简化基因组+重测序的方法对鸡的多趾性状进行了遗传定位,提示LMBR1可能就是控制鸡多趾性状的基因。
参考文献:
He C, et al. Genetic pattern and gene localization of polydactyly in Beijing fatty chicken. Plos One, 2017, 12(5):e0176113
- 本文固定链接: https://maimengkong.com/zu/807.html
- 转载请注明: : 萌小白 2021年11月21日 于 卖萌控的博客 发表
- 百度已收录
