必备软件
Aspera 插件 下载网址 : https://www.ncbi.nlm.nih.gov/Traces/sra_sub/sub.cgi
“
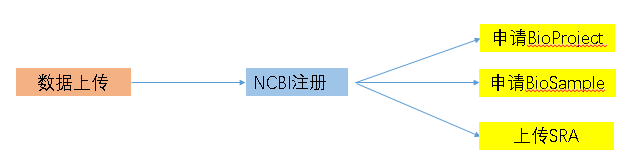
每个过程对应的网址:
NCBI注册 网址:https://www.ncbi.nlm.nih.gov/account/
申请BioProject 网址:https://submit.ncbi.nlm.nih.gov/subs/bioproject/
申请BioSample 网址:https://submit.ncbi.nlm.nih.gov/subs/biosample/
上传SRA 网址:https://www.ncbi.nlm.nih.gov/Traces/sra_sub/sub.cgi
数据整体上传的流程图如下:
1
注册NCBI
打开 NCBI,点击右上角的“Log in”,如下图:

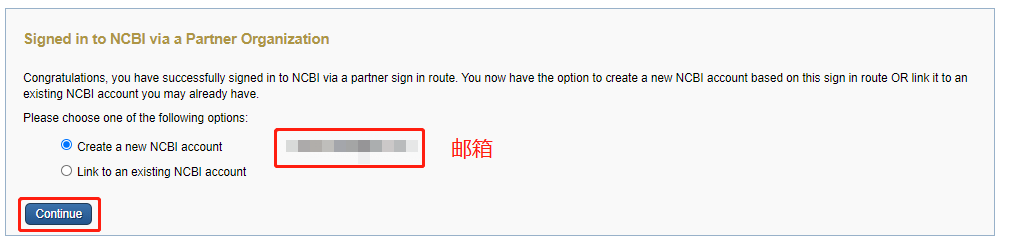
很多同学现在打开NCBI想注册/登录的时候,会发现如下提示:强制性使用第三方进行注册/登录。

继续往下看就能找到解决办法,NCBI支持用各种第三方账户:

根据经验,ORCID账号和Microsoft注册起来都很方便,只需要个人邮箱并按要求填写信息后,点击“Continue”完成注册。
2
创建Bio-Project
01
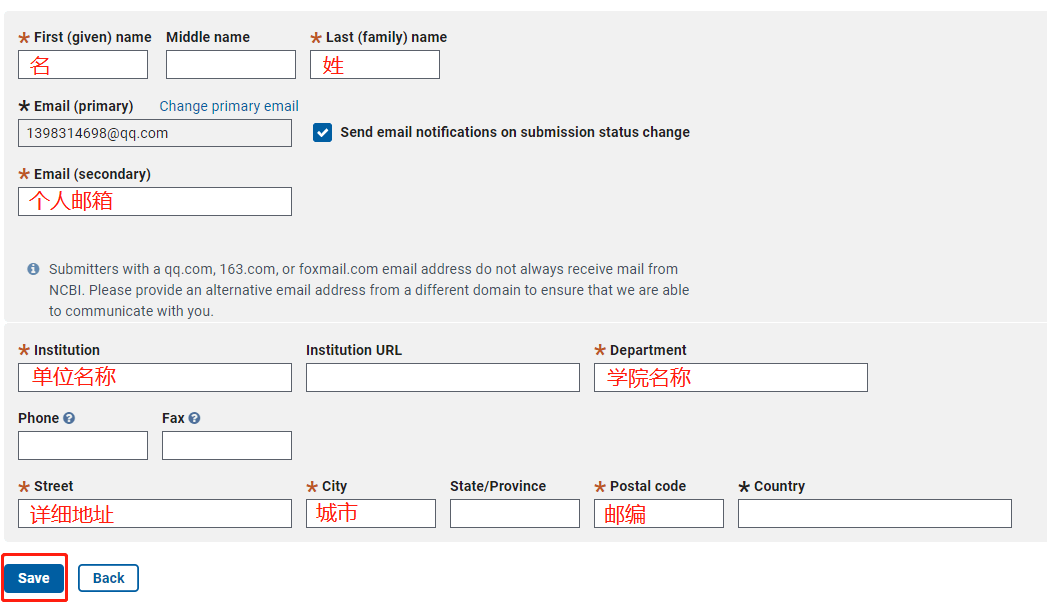
输入BioProject网址,先进行个人信息完善,以便成功申请到 Bio-Project。点击“My profile”完善个人信息后并点击“Save”,即可完成填写。

02
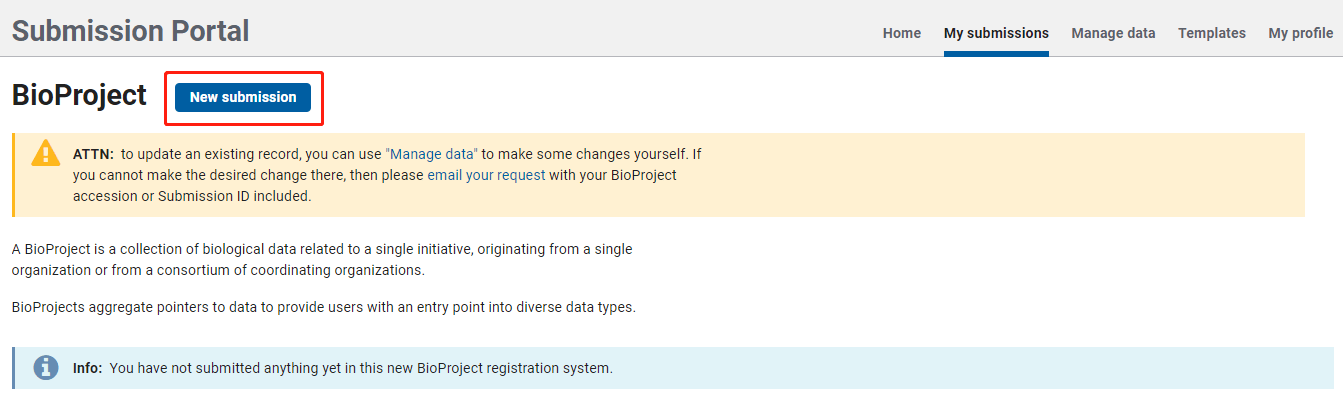
点击 BioProject,再点击 New submission,进入BioProject 的信息填写页面,按照步骤进行填写。
A
Submitter界面直接点击continue;
B
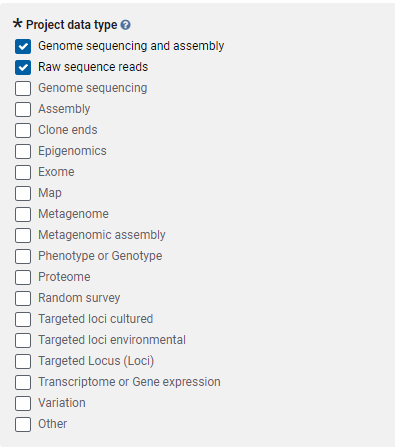
Project Type 界面,选择 Raw sequence reads,有组装数据的也选择 Genome sequencing and assembly;Sample scope 有 5 类选择:单一样本,多个样本,多物种,环境样本,合成样本等,可按照实际点击,其中普通转录组选择:Multiisolate;微生物多样性或宏基因选择Environment;

C
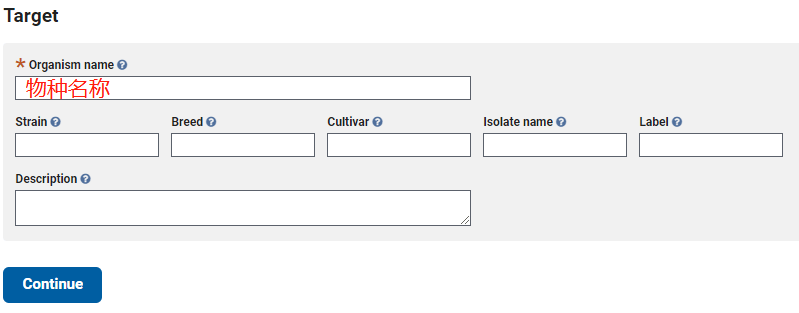
Target 界面,只需填写研究物种拉丁文名即可;
D
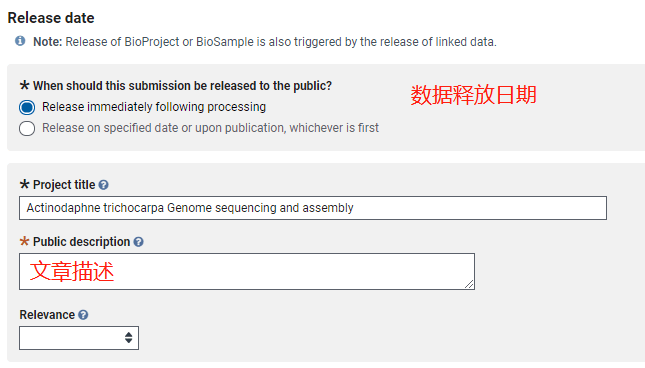
General Info 界面,选择数据释放日期(可以立马释放,也可指定日期),Public description 部分对自己的研究简要描述即可;
E
BIOSAMPLE、PUBLICATIONS 界面可直接跳过,最后点击 Submit,等待 NCBI 审核通过,即可完成 Bio-Project 的创建。
3
创建Bio-Sample
01
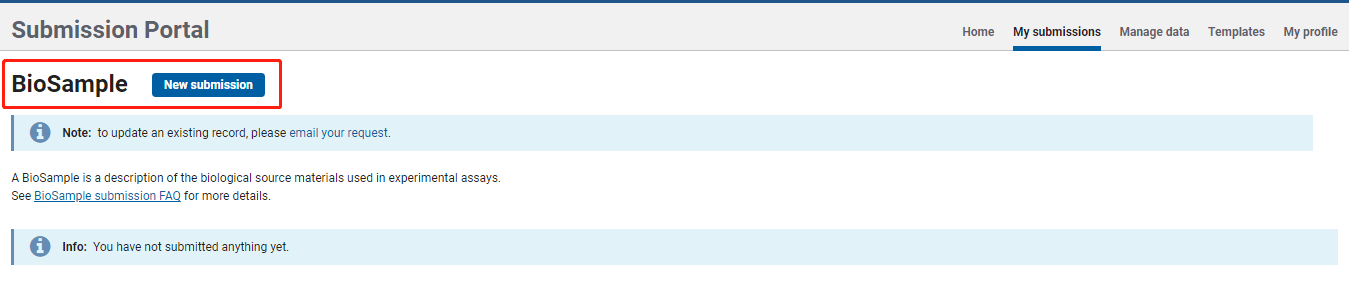
输入BioSample网址,点击 New submission,即出现 BioSample 界面,如下:
02
Submitter界面直接点击Continue;
03
General Information 界面,选择数据释放日期和样本个数,点击 Continue;

04
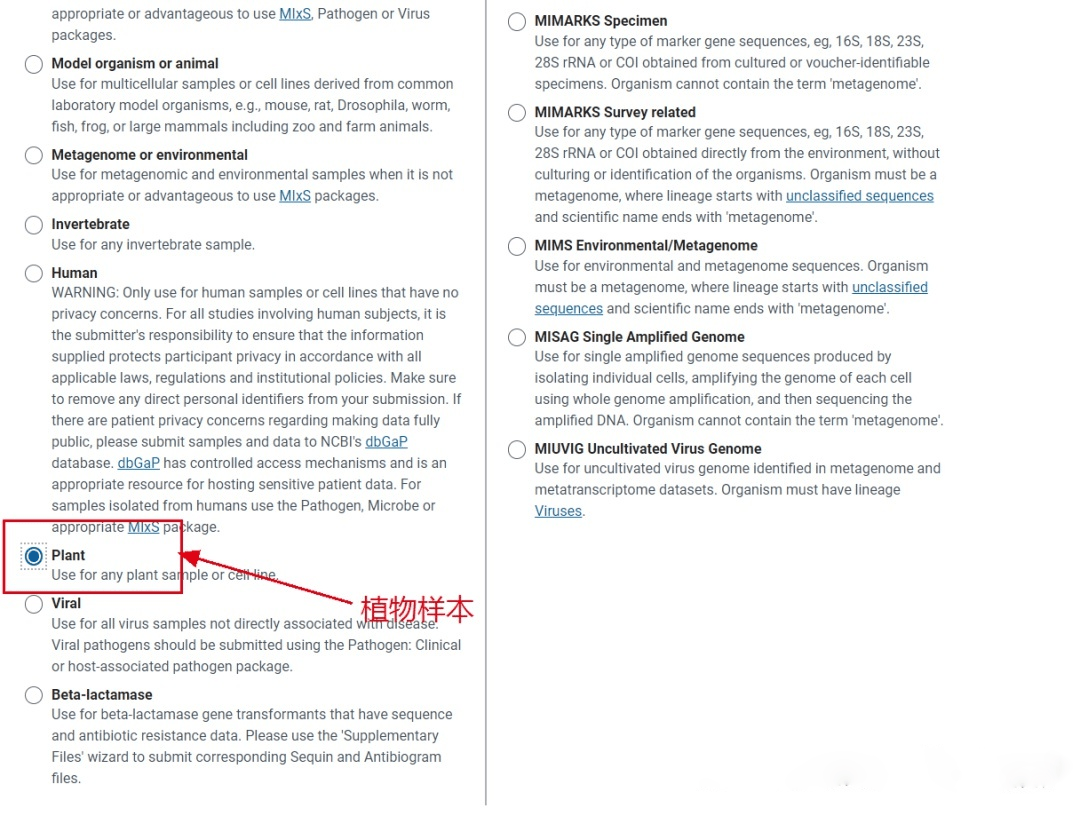
Sample Type 界面,有 10 种类型描述样本,例如病原体、微生物、模式生物、无脊椎动物、人、植物样本等,这里我们选择“Plant Sample” ,点击 Continue;
05
Attributes界面,可以上传表格,也可以自行填写表格,
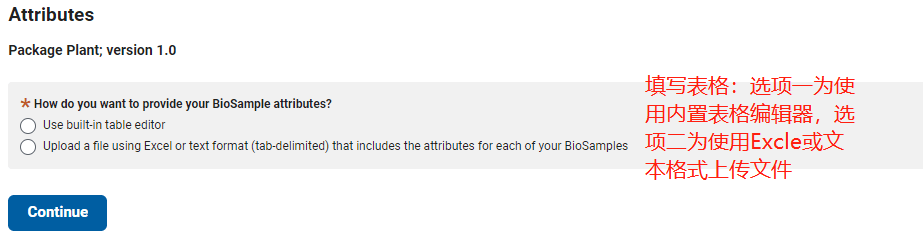
这里我们推荐首次上传原始数据的用户选择自行填写表格;
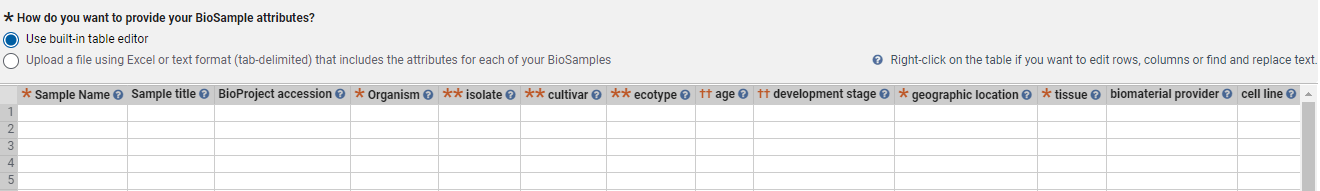
其中,必填字段用*星号标记;标有**、††或†的字段为选填项,但至少填写一个。
如上传表格数据,以下载Excel表格为例进行操作:

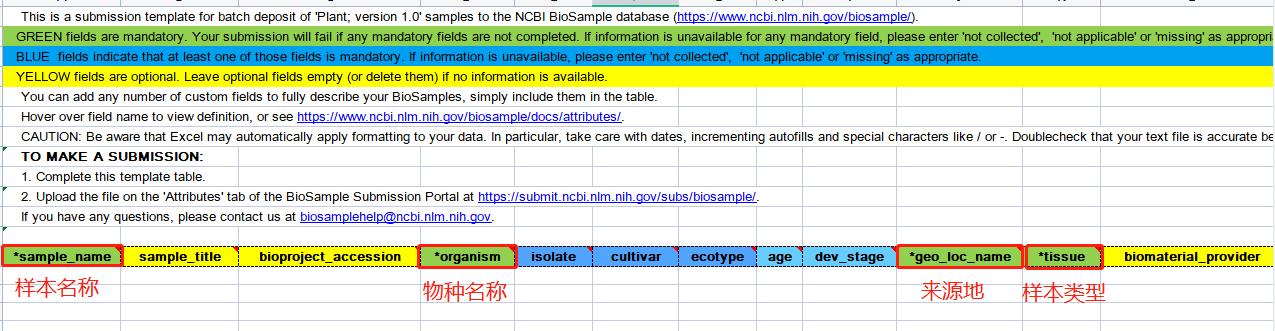
这里需要注意一下表头的颜色,其中绿色为必填项,蓝色为选填项,黄色为非填项。如果绿色的任何必填字段的信息不可用,可输入“not collected”、“not applicable”或“missing”替代。蓝色虽为选填项,但至少填写一个,同样出现何必填字段的信息不可用,请输入“not collected”、“not applicable”或“missing”替代。黄色可以不予填写。每一个单元格填写是有格式要求的,可以点击需要填写的列名查看具体格式!
06
完成后,确认信息无误,点击Submit,即完成BioSample的创建。
4
数据上传SRA
SRA简介:SRA(Sequence Read Archieve)数据库是NCBI搭建的存放原始测序数据的平台。SRA能兼容不同测序平台的数据,比如Sanger测序、Illumina测序、PacBio测序等,所以无论是基因组、重测序、转录组、微生物扩增子,还是宏基因组等组学分析,都可以将原始数据上传SRA。
01
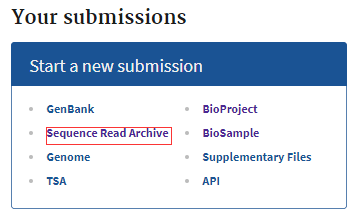
点击My submissions页面中的“Sequence Read Archive”,之后点击“New submissions”,即可进入到SRA提交界面。
02
A
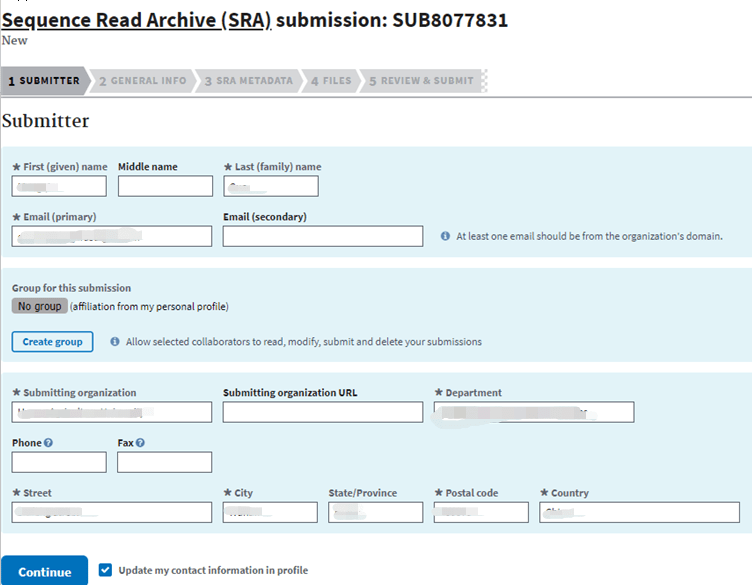
Submitter 页面点击Continue;
B
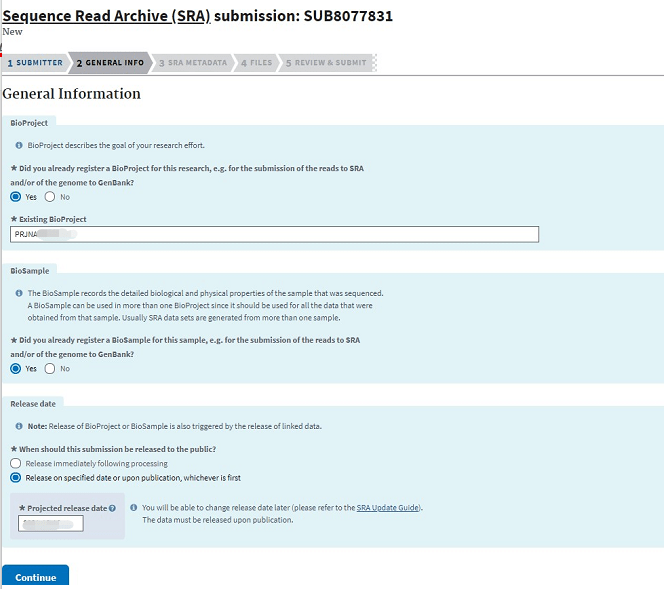
进入General Information页面,填入Existing BioProject号码,然后选择数据释放日期,需与前两步数据释放日期一致,之后点击Continue;
C
进入SRA metadata界页面,填写对应表格信息,这里我们推荐直接在网页版上填写表格,需要注意的地方如下:
BioSample accession:注册好的Biosample编号直接填写;
Library ID:它是唯一的,我们可以使用物种拉丁文名命名。
Library strategy可以选择WGS,Library source可以选择Genomic,Library selection可以选择size fractionation,Library layout可以选择paired,Platform可以选择illumina,instrument model可以选择Illumina NovaSeq 6000;
Filetype可以选择fastq;后面的Filename 可依次填写M_sinensis_A_illumina_R1.fastq.gz、M_sinensis_A_illumina_R2.fastq.gz等,填写完毕后点击Continue。
D
Files界面:数据上传的选择方式,若单个文件数据量小于10G,样本数小于300个,我们推荐Aspera方式上传(如下图);
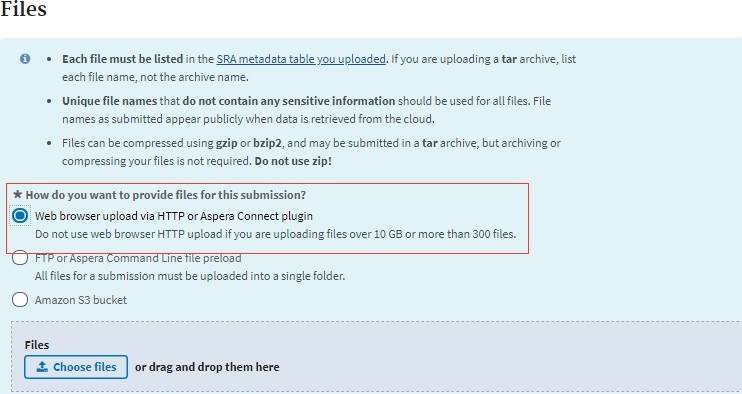
然后我们点击choose files,选择相应的桌面文件,如下:
注意事项:
(1)所有样本的原始数据需放在同一目录下;
(2)原始数据为 fastq 格式的压缩文件;
(3)若为双端测序,需要提供双端的原始数据,即 R1 端和 R2 端。
接着会显示上传界面,网页版显示如下:
Aspera软件界面显示如下:
03
待所有数据上传成功后,我们就可以进入确认界面,点击submit,即SRA数据上传完成,我们只需等待NCBI审核通过,就可获得对应样本的SRA编号。
- 本文固定链接: https://maimengkong.com/zu/1782.html
- 转载请注明: : 萌小白 2024年8月21日 于 卖萌控的博客 发表
- 百度已收录
