今天小编要和大家分享的是去年12月发表在OncoImmunology(IF:5.869)杂志上关于免疫聚焦多组学数据分析前列腺癌的文章,更多生信人原创文章见:生信人。https://biosxr.gaptools.cn/#/
Immune-focused multi-omics analysis of prostate cancer: leukocyte Ig-Like receptors are associated with disease progression
前列腺癌免疫聚焦多组学分析:白细胞 lg样受体与疾病进展相关
一.研究背景
近年来,免疫检查点抑制剂的发展使肿瘤免疫治疗发生了巨大的变化。免疫检查点是一系列受体/配体,可以抑制或激活免疫细胞的功能。然而,到目前为止,前列腺癌(PCa)免疫疗法对晚期癌症疗效仍然是有限的。因此,今天分享的文章整合多组学分析研究了LILR家族基因,特别是LILRB1作为PCa免疫治疗靶点的潜力。
二.数据与方法
1.患者及数据集:作者从TCGA下载了498个样本的NGS RNA-seq,miRNA,甲基化,CNA,SNP数据,及从GDC下载了相关的临床数据。作者也从GEO下载了106例患者的RNA-Seq及临床数据。此外,作者也收集了温哥华前列腺癌中心(VPCC)中85例患者的RNA-Seq及临床数据作为验证集。
2.组学数据的处理:
1)RNA-Seq 数据:作者完全分析RNA-Seq数据,以避免数据处理中的变异性。同时使用共同的分析 pipeline确保转录组数据集综合分析的准确性。原始FASTQ文件使用FastQC定量Trimmomatic修饰。
2)miRNA 数据:作者对来自TCGA的level 3 NGS miRNA数据进行标准化,将标准化计数为零或NA的miRNA从miRNA数据集中删除。
3)甲基化数据:使用level 3的甲基化数据且选择β值,使用ß值评估甲基化水平,ß值表示甲基化和未甲基化等位基因之间的比率强度。
4)CNA数据:作者从TCGA下载level 3 DNA拷贝数数据,并使用Birdsuite进行处理。对数据进行清洗、标准化、分段和对数变换。
5)SNV 数据:作者使用VCFtools处理来自TCGA的level 3 SNV数据。
6)免疫基因集:分析所选择的免疫基因集由812个基因组成。这组基因来源于与免疫系统相互作用的肿瘤细胞anti-genome 的meta分析。
3.生存分析:作者使用cox风险比模型、K-M分析来进行生存分析。
4.免疫组化分析:作者使用免疫组化分析LILRB1表达。
三.主要内容与结果:BCR相关的特征
首先,作者为了识别与生化复发(BCR)相关的免疫基因相关特征,使用R包mixOmics 的sPLS-DA模型分析每种组学数据。最终,作者识别出51个mRNA, 44个miRNA, 36个甲基化位点,32个CNA和6个SNV。从图1中可以观察到mRNA, miRNA,及甲基化预测BCR发生的平衡错误率(BER)良好, 然而CNA 及SNV没有很好的预测BCR。接下来,作者将所有这些特征合并到一个数据集中,并执行一个通用的sPLS-DA分析,结果发现能够很好的预测BCR(图2)。
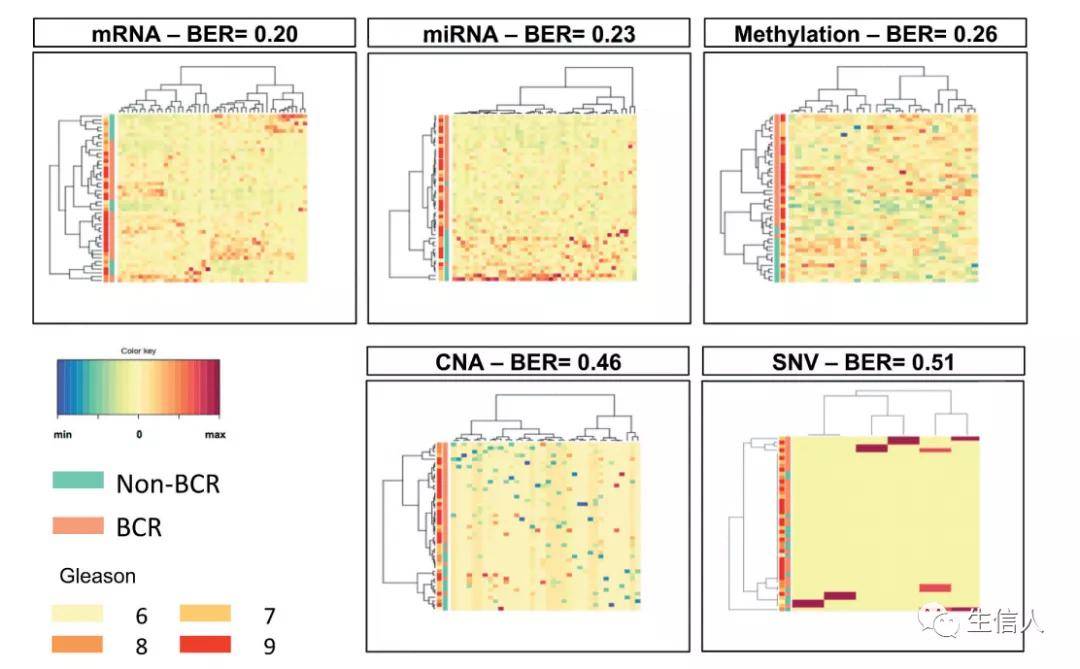
图1 来自TCGA 的单个组学数据集的sPLS-DA结果
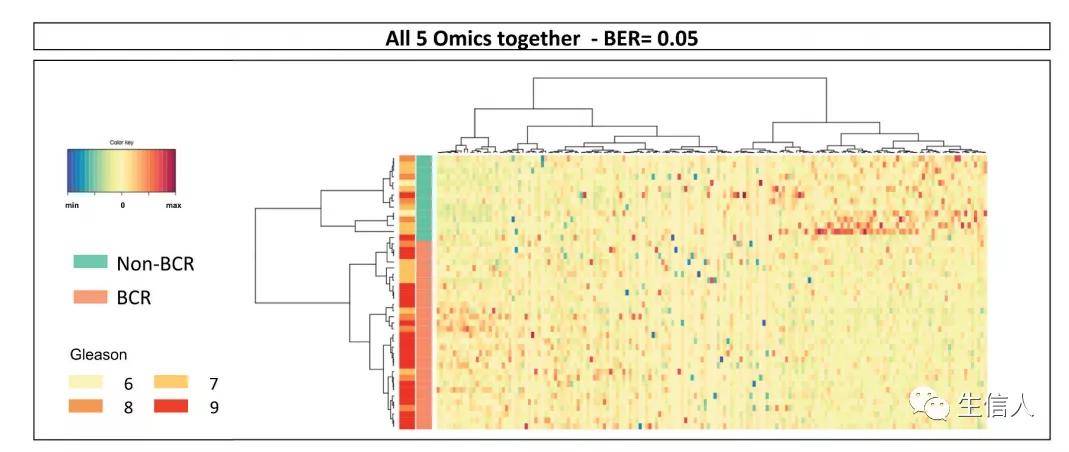
图2 TCGA 组学数据集的sPLS-DA结果
接下来作者分析识别了mRNA的特征,发现一些特征与抗原加工与呈递有关,这其中包括HLA分子、杀伤细胞免疫球蛋白样受体、及LILR家族基因。由于LILR免疫调节因子家族,显示出作为癌症免疫治疗靶点的潜力,因此,作者进一步分析了30个LILR基因相关特征与BCR之间的关联。如图3所示,可以观察到在sPLS-DA分析中,仅30个LILR基因相关特征就可以预测BCR,表明这个家族的受体在PCa的进展中发挥了作用。
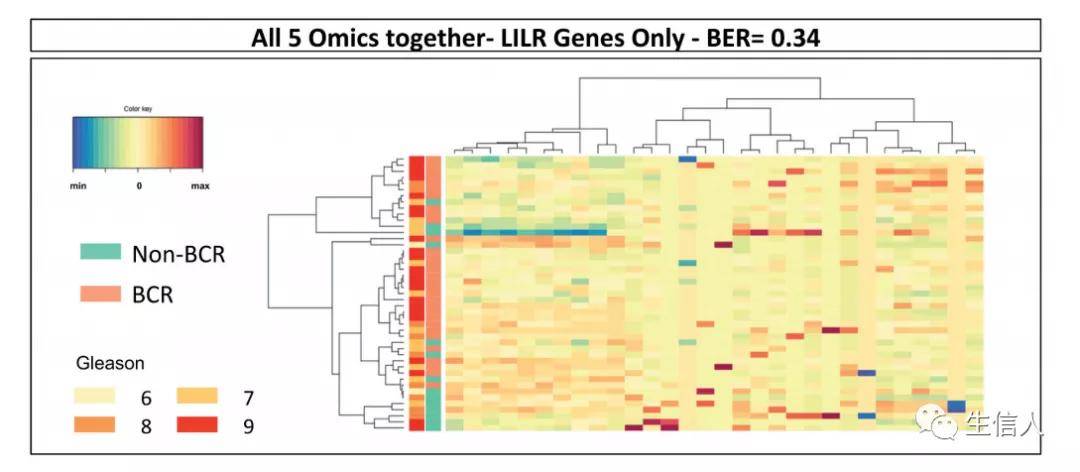
图3 TCGA 结合组学数据中LILR基因相关特征的sPLS-DA结果
接下来作者为了进一步验证LILR基因和BCR的相关性。作者整合171例肿瘤RNA-seq数据集分析使用Multivariate INTegrative (MINT)方法评估LILR基因与BCR的相关性。图4展示了来自LILR基因家族的5个基因(LILRB1、LILRB2、LILRB3、LILRB5和LILRA3)与BCR相关,证实了LILR基因与BCR之间存在关联。

图4 MINT分析结果
这一部分,作者为了进一步刻画LILR基因和BCR的关联,作者进行了K-M分析。图5a可以看到,正如MINT分析所揭示的,LILRB1是一个与BCR密切相关的基因,因为LILRB1 mRNA的高水平与较短的BCR生存相关。此外,也可以观察到高水平的LILRB2 mRNA也与较短的BCR生存相关,但这种关联不那么显著(图5b)。LILRB5 mRNA的高水平与较短的BCR生存几乎显著相关(图5d),而LILRB3 mRNA单独表达与BCR生存无关(图5c)。生信人进一步研究发现,将这4个基因的mRNA水平加起来,与BCR生存的相关性并不比单独的LILRB1 mRNA更好(图5e),说明LILRB1是这种相关性的主要驱动因素。此外,将LILRB1从组合中去除会极大地影响该关联显著性,这也支持了LILRB1的重要性。相反,高水平的LILRA3 mRNA与较好的BCR生存显著相关(图5e)。
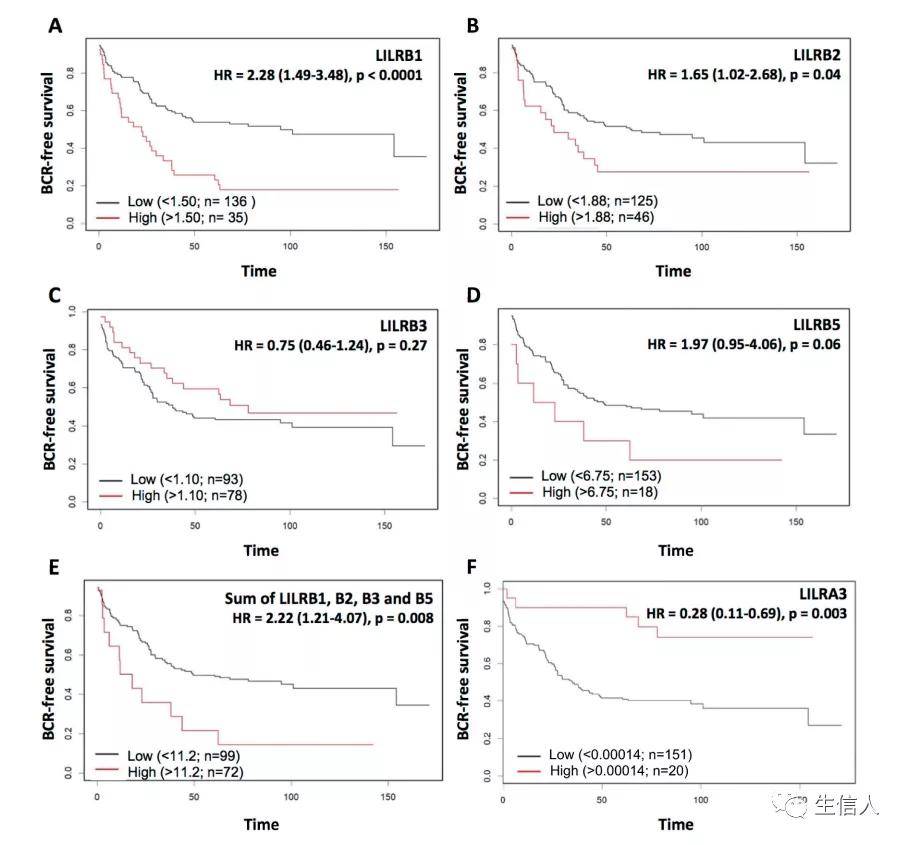
图5 K-M分析LILR家族基因与BCR生存相关性
接下来,由于TCGA-GSE54460-VPC联合队列由高风险肿瘤组成,作者下一步试图确定这些基因与BCR的相关性是否在中危PCa样本队列中保持不变。因此,作者使用由144名男性组成的亚队列进行了新的Kaplan-Meier分析。图6(b-d)显示,高水平的LILRB2、LILRB3和LILRB5 mRNA水平与较短的BCR生存显著相关。接下来,作者整合LILRB1、LILRB2、LILRB3和LILRB5 mRNA水平发现其总和与BCR相关(图6e)。
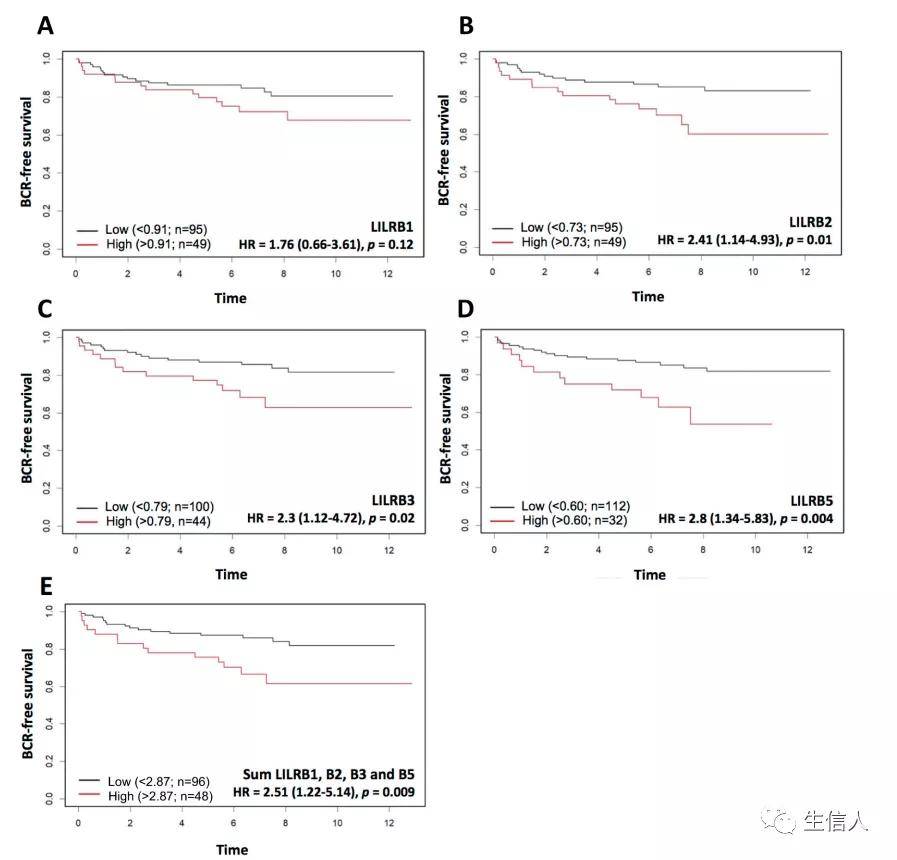
图6 CPC-GENE RNA-seq 数据中分析LILR家族基因与BCR生存相关性
由于在中等风险队列中,LILRB1 mRNA水平与BCR之间没有统计学意义上的显著关联,作者推测LILRB1基因的表达可能与grade有关。而Spearman相关分析也显示,在TCGA-GSE54460-VPCC队列中,LILRB1 mRNA水平与grade相关,而在联合队列中,LILRB2、LILRB3、LILRB5、LILRA3 mRNA水平与grade无关。为了进一步评估LILRB1基因与BCR的关系,作者通过免疫组化方法分析了20例高危前列腺肿瘤中LILRB1蛋白的表达。结果在分散于肿瘤腺体间的免疫细胞中发现了LILRB1蛋白。肿瘤细胞或基质细胞均未表达该蛋白。K-M分析也发现 LILRB1+细胞浸润肿瘤临床结局更差(图7b)。
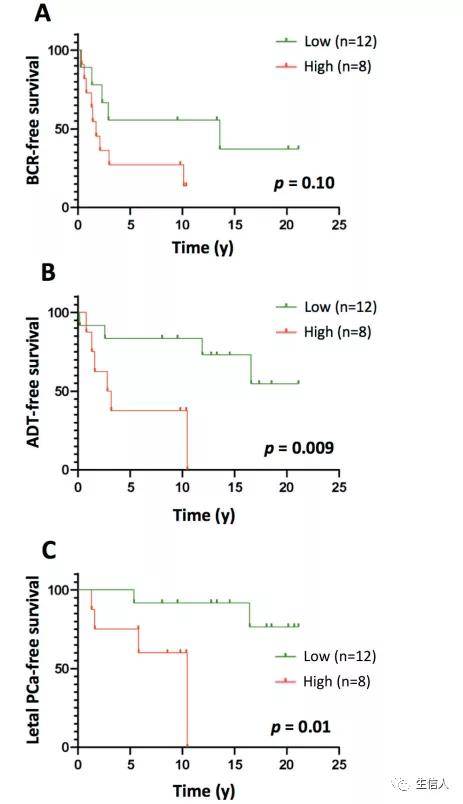
图7 BCR生存分析
到这里这篇文章的主要内容就介绍完了,文章使用PCa数据集进行了多组学分析,结合稀疏偏最小二乘判别分析、生存分析等生物信息方法识别了一系列与BCR密切相关的免疫特征。并对这些特征进行进一步分析最终确定一些候选分子靶点,这些分子靶点可以用于PCa的免疫治疗干预,而文章的亮点应该是整合了多组学数据进行分析,小伙伴们可以参考哦。生信人。https://biosxr.gaptools.cn/#- 本文固定链接: https://maimengkong.com/zu/1666.html
- 转载请注明: : 萌小白 2024年2月4日 于 卖萌控的博客 发表
- 百度已收录
