这段时间许多童鞋在后台问,TRIzol裂解的细胞能做翻译组测序(Ribo-seq)吗?今天统一回复下。
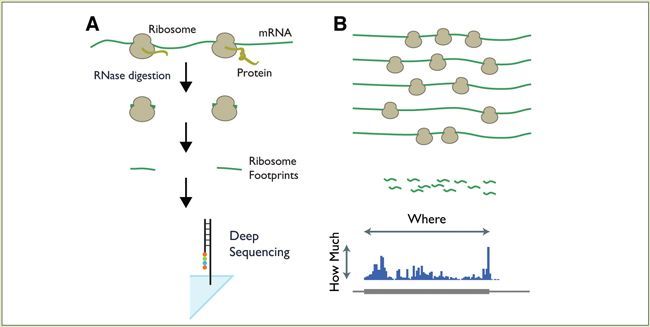
图 (A)核糖体保护mRNA足迹不被消化;
(B)将足迹与基因组比对,可以显示出哪些mRNA正在翻译以及翻译水平高低
上图是Ribo-seq简化的实验流程图。Ribo-seq实验是这样做的:快速收获细胞并裂解,保持体内核糖体结合mRNA的位置。然后用RNase I对细胞裂解液进行消化处理(Nuclease Footprinting),此时未被核糖体包裹的RNA序列会被消化掉,只留下核糖体结合的28 nt左右的RNA片段。接着通过超速离心从消化的裂解物中沉淀出核糖体,然后通过变性胶电泳从核糖体沉淀中分离出RNA(Ribosome Footprints)。将接头连接到RNA片段上并进行后续的文库构建和测序。最后对测序得到的数据进行分析。
核糖体结合的RNA片段是从哪步分离出来的?是从沉淀下来的核糖体里分离出来的。Ribo-Seq实验是从细胞或组织开始的,而不是从总RNA开始的。而裂解细胞或组织的试剂也不是TRIzol,而是用专门配制的裂解液。为获取体内核糖体结合mRNA的真实情况,通常在裂解细胞前,会使用Cycloheximide(放线菌酮)来抑制翻译延伸。我们已准备好了翻译组实验专用的裂解液配方,需要的童鞋可以留言索取。
专用裂解液裂解的细胞还能做转录组测序吗?不能!
想将转录组和翻译组数据一同分析的童鞋,请另外多准备一份样本做转录组测序,如果是细胞,这份样本用TRIzol裂解处理是ok的。
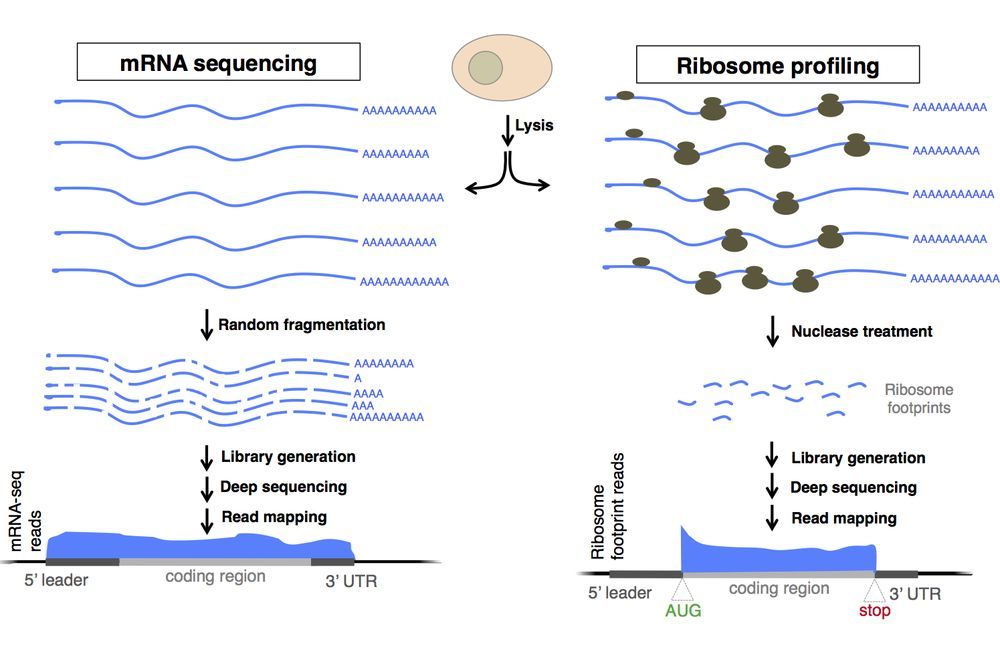
图 转录组与翻译组整合分析
参考文献
1. N.T. Ingolia, et al. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments, Nat. Protoc. 7 (2012) 1534–1550.
2. N.T. Ingolia, et al. Tranome-wide measurement of translation by ribosome profiling, Methods 126 (2017) 112–129.- 本文固定链接: https://maimengkong.com/zu/1593.html
- 转载请注明: : 萌小白 2023年7月28日 于 卖萌控的博客 发表
- 百度已收录
