近年来,全基因组关联分析(Genome wide associationstudy,GWAS)在筛查和鉴定动植物重要经济性状的主效基因方面得到了广泛应用。GWAS关联精度高,研究周期短,已成为功能基因挖掘的重要手段。
我们在看文献的时候一般会看到全基因组关联分析线性模型计算出的曼哈顿图和QQ图。那在关联分析之前,如何评估群体结构和亲缘关系?你知道么?
图1 曼哈顿图和QQ图[1]
今天我们就从GWAS入手,分享其相关的研究方法,帮助大家深入了解应该怎么进行GWAS。
1
系统发育树的构建
我们进行系统发育树的构建是为了揭示群体中每个个体的聚类关系,它可以用来描述样本之间的分类和演化关系,是反映群体结构最经典、直观、有效的方法。目前发育树主要的构建方法有距离法(如NJ)、最大简约法(MP)、最大似然法(ML)和贝叶斯推断法(BI)。
但是ML、BI法主要用于序列差异较大材料的进化树构建,构建过程需要的时间较久;MP法则易出现长枝吸引(LBA)现象,会使原本不是姐妹群的分类元错误的聚集在一起,干扰发育树的构建。而在GWAS模型中,个体间关系相对较近,序列差异小,因此采用NJ法即可满足要求。
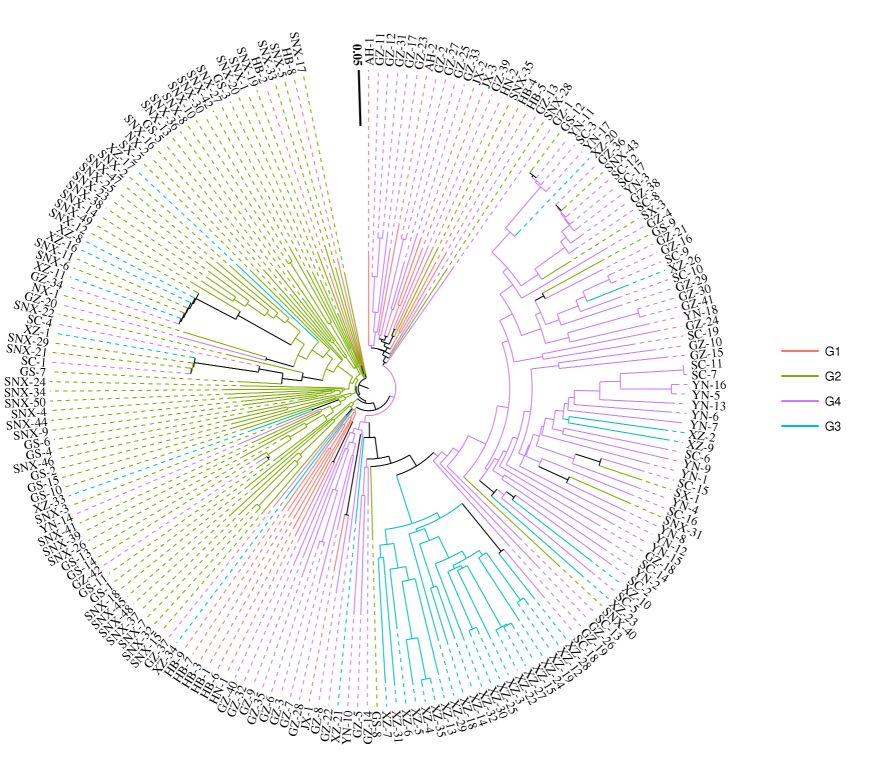
图2 系统发育树
2
群体结构分析
群体结构分析是为了显示每个个体的来源以及个体的组成信息。一般利用Structure软件进行群体结构推断,该软件是使用贝叶斯推断的方法,对每个K值模拟的结果都会对应产生最大似然值(likelihood)。
structure软件中每个最大似然值是取了自然对数后输出的(ln likelihood)。lnlikelihood越大、重复间的ln likelihood变化越小,说明K值越接近于真实情况。简单来说,就是likelihood越大,越趋近于平缓处对应的最小K值,则是我们要选择的最优K值。
但是Structure软件有一个很大的缺点就是运行效率太慢,因此出现了很多类似的代替软件,其中Admixture软件是比较常用的。该软件与Structure软件分析原理相同。但Admixture是根据交叉验证错误率来确定最优分数群,交叉验证错误率最小的K值对应最优的分群数。
最优K值对应的样本遗传成分矩阵,可以作为GWAS的固定效应协变量矩阵(Q矩阵),用以控制群体结构造成的假阳性。
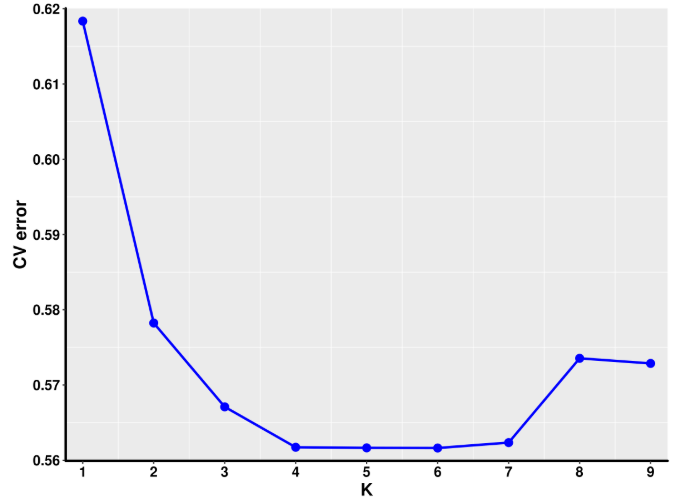
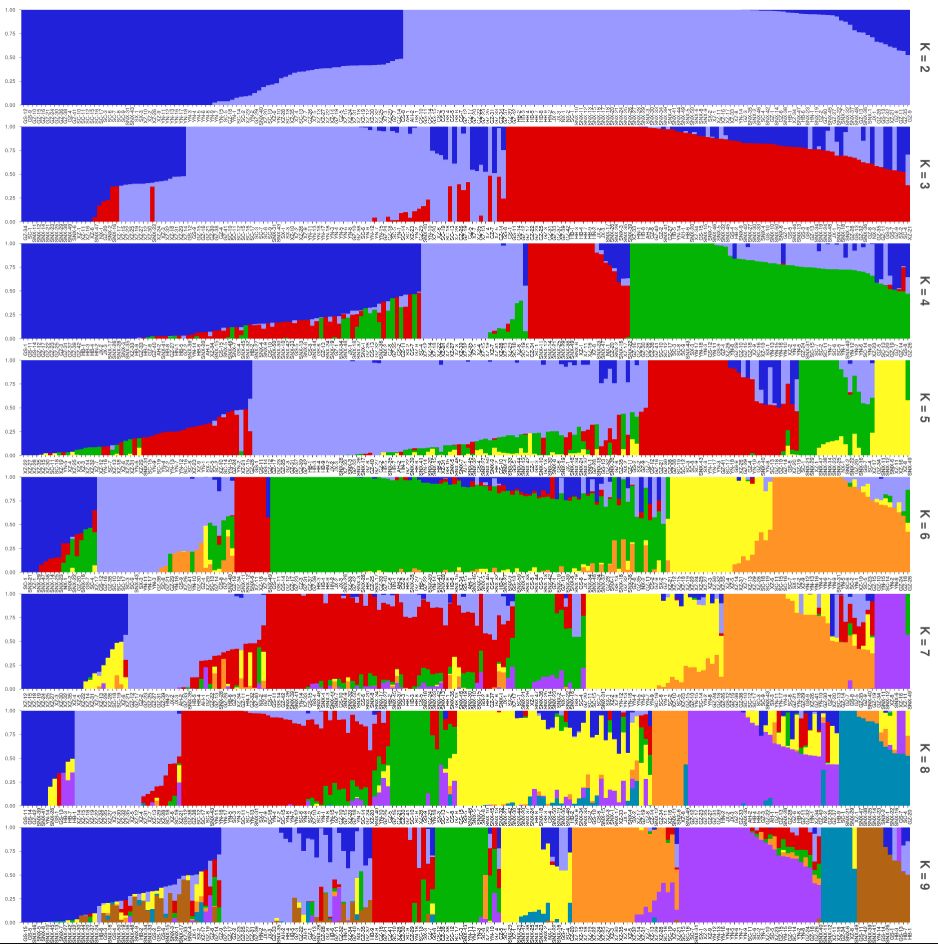
图3 Admixture群体结构分析
3
PCA分析
在群体遗传学中,PCA分析可以把成千上万的标记进行综合,抽取主要信息,用2-3个维度把群体中样本的聚类关系弄清楚。PCA分析与群体结构分析可以相互验证,使用时选择其一即可。
当群体结构的最优分群数较低,从进化树和PCA结果看材料分化程度又比较高时,优先选用PCA分析中各个材料在前几个主成分中的得分矩阵作为协方差矩阵参与关联分析,用于模型中控制群体结构造成的假阳性。

图4 样本聚类图
4
亲缘关系的评估
亲缘关系是指在非家系群体或系谱不明确的群体中两特定材料之间的遗传相似度与任意材料之间的遗传相似度的相对值。材料间不平衡的血缘关系是导致标记出现非连锁相关的另一个重要原因,小家系的存在会使关联分析结果出现假阳性。为了避免这种情况的产生,往往会把亲缘关系矩阵作为随机效应协变量矩阵(K矩阵)加入GWAS模型。
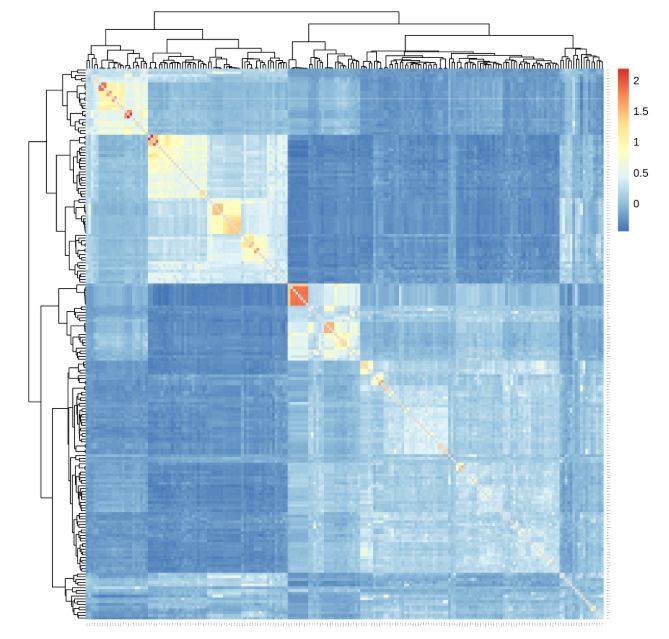
图5 亲缘关系矩阵
那么现在,你了解GWAS了么?
基迪奥一直秉持优秀的服务质量和定制化的个性分析,全面更新了群体进化与全基因组关联分析流程,让您的GWAS更丰富、更简单。欢迎各位老师与我们联系!
参考文献
[1]Huang X, Zhao Y, Wei X, et al.
Genome-wide association study of flowering time and grain yield traits
in a worldwide collection of rice germplasm[J]. Nature Genetics, 2012,
44(1):32.
转自:基迪奥
- 本文固定链接: https://maimengkong.com/zu/1456.html
- 转载请注明: : 萌小白 2023年4月17日 于 卖萌控的博客 发表
- 百度已收录
