在特定的细胞或组织中,发挥作用的转录因子在调控网络中的地位也是不同的。来自胚胎干细胞和其他细胞模型的研究表明,一小组细胞类型特异性或谱系特异性转录因子(TFs)形成了相互连接的自动调节环,以控制特定细胞类型的转录程序。将某种细胞或者组织中的核心转录因子及其调控回路称之为core transcription regulatory circuitry, 简称CRC。核心TF及其相互连接的自动调节环对于维持细胞身份和细胞状态至关重要,它代表了核心转录调节通路(CRC)。
a database of Core transcriptional Regulatory Circuitries,简称dbCoRC。dbCoRC在2018 NAR中发布,是第一个全面的交互式CRC数据库,也是核心启动因子数据库。它基于H3K27ac ChIP-Seq数据的计算分析,包含来自188个人类和50个鼠类样本的CRC模型。与CRC模型配合使用时,该数据库提供以下可下载的信息:1)单个样品的超级增强子(SE),增强子和H3K27ac景观; 2)CRC内SE区中每个核心转录因子(TF)的假定结合位点。此外,dbCoRC结合了CRC TF的一般描述以及它们在正常或癌细胞/组织中的表达数据。因此,dbCoRC可作为研究生理(非疾病)和疾病状态下的转录网络和调控回路的宝贵资源。
dbCoRC
http://dbcorc.cam-su.org/

一起来看看dbCoRC是如何使用的吧!
dbCoRC编译188个人类和50个鼠细胞系/组织样本的CRC模型。在188个人类样本中,其中79个是癌细胞/组织,其中109个是非肿瘤细胞/组织。鼠数据集包含1个恶性细胞系和49个正常细胞/组织。详细的样品信息可在“浏览”页面中找到 ,样品和CRC信息的总体摘要显示在“ 统计信息”页面中。
1
搜索
快速搜索与精确搜索
在dbCoRC的主页上,我们可以输入TF基因ID或符号来快速搜索相应的感兴趣基因。已实施模糊搜索功能,以帮助识别包含相同字符的潜在相关基因。快速搜索结果将被定向到“ SEARCH”页面。

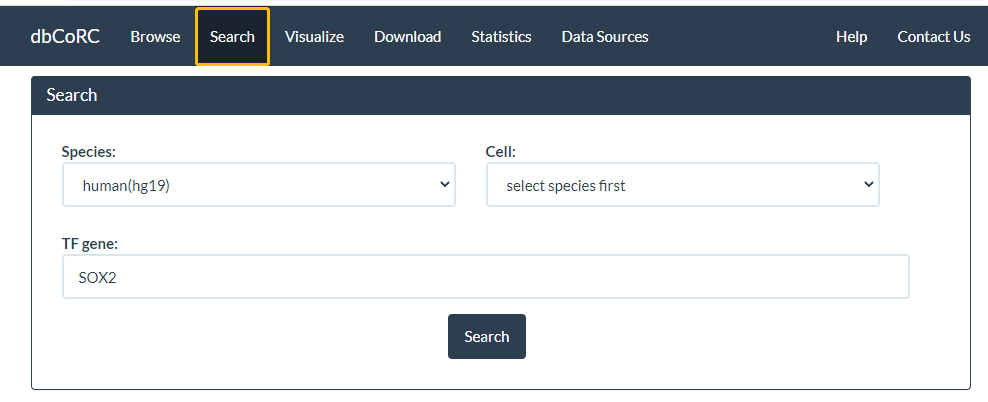
在“搜索”模板中,可以精确搜索各种样本中潜在的核心TF。我们可以选择“基因组”或“细胞/组织类型”以限制搜索范围,然后在“基因”框中输入感兴趣的单个或多个TF(以逗号分隔)。搜索结果将作为数据表显示。
本次选择CAMSAP1基因作为示例,搜索结果如下:
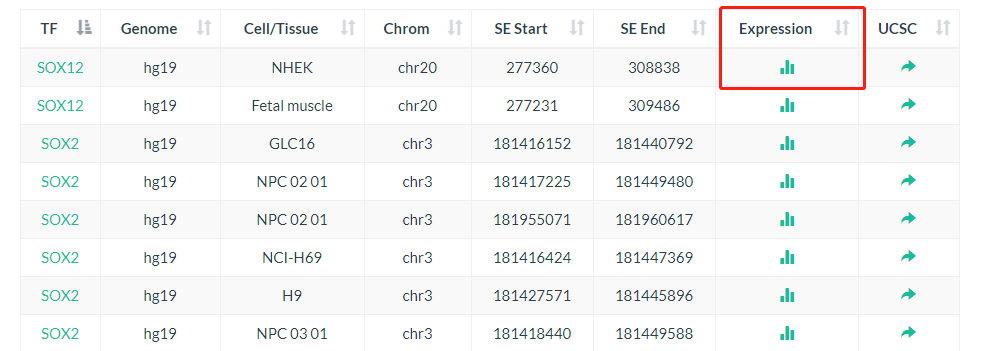
展示的是核心转录因子对应的超级增强子区域,我们可以通过单击EXPRESSION找到各个核心TF的表达模式,展示内容分为4个部分,具体如下:
Part1--转录因子描述,介绍其名称与组织类型、数据源等内容:

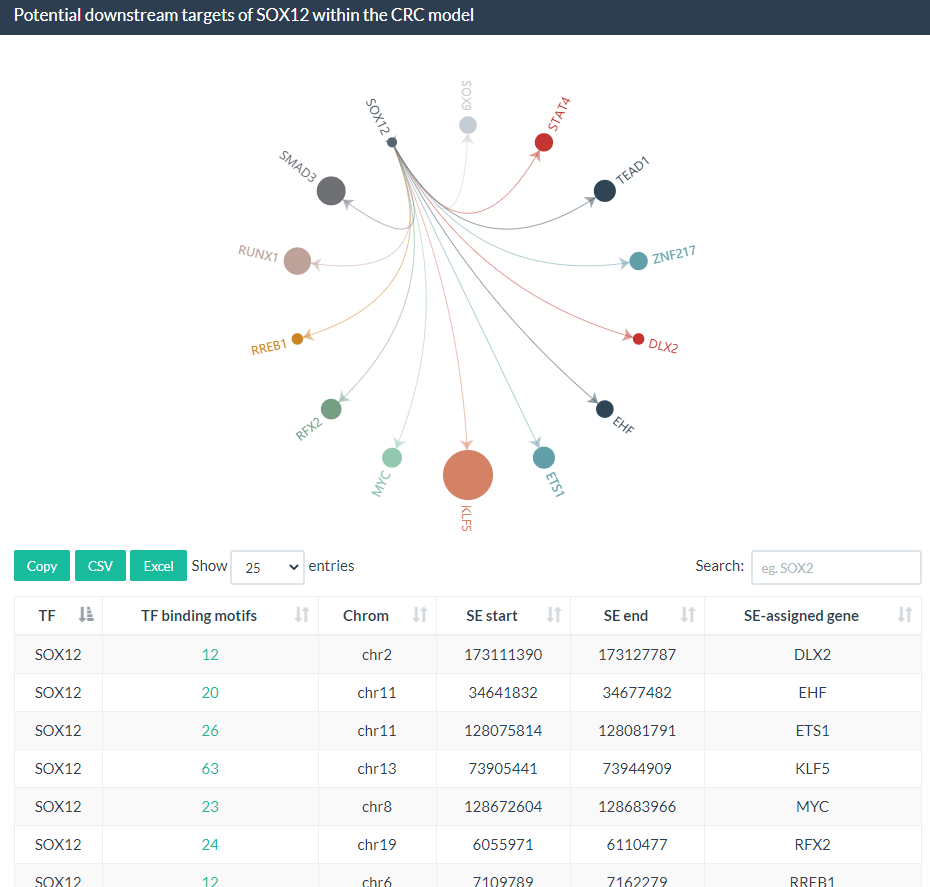
Par2--CRC模型中SOX12的潜在下游目标,点击转录因子的ID, 可以查看该转因子在其他转录因子SE区域的结合位点:
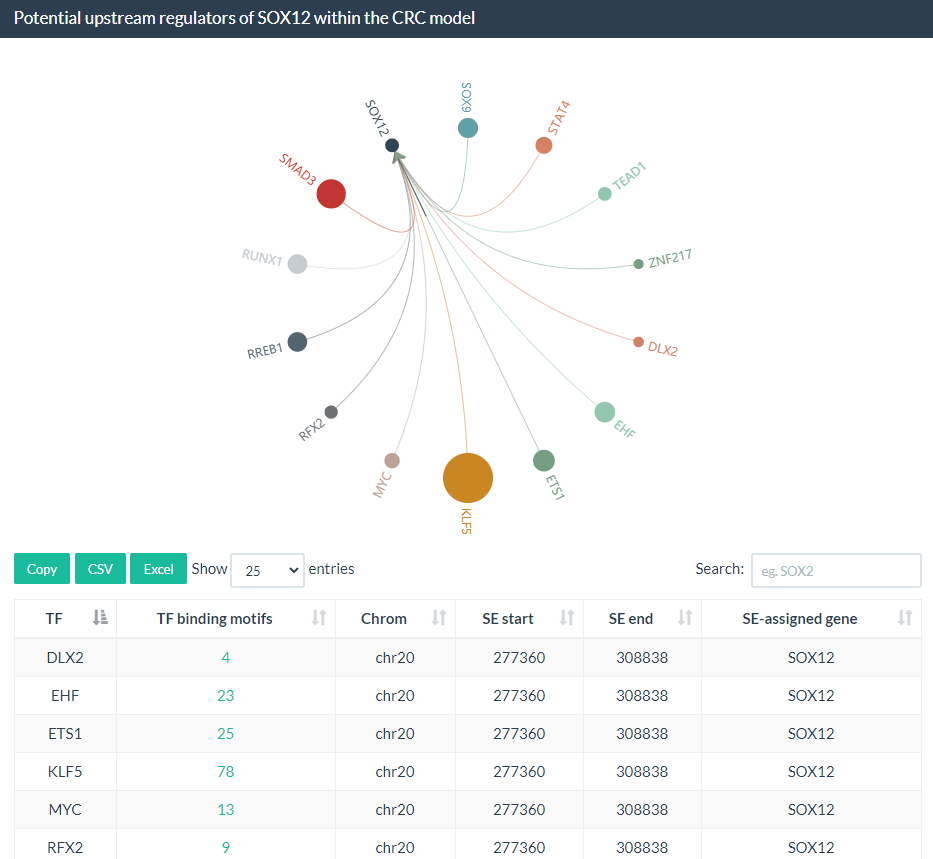
Part3--CRC模型内SOX12的潜在上游调节剂:
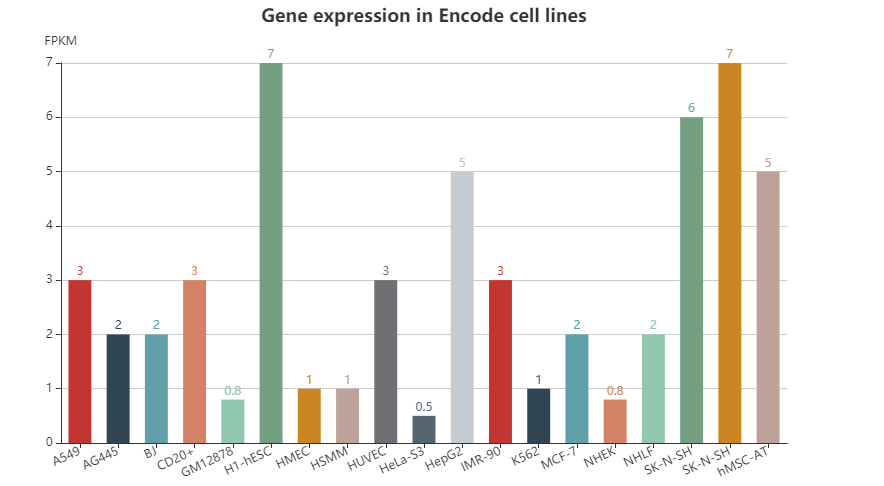
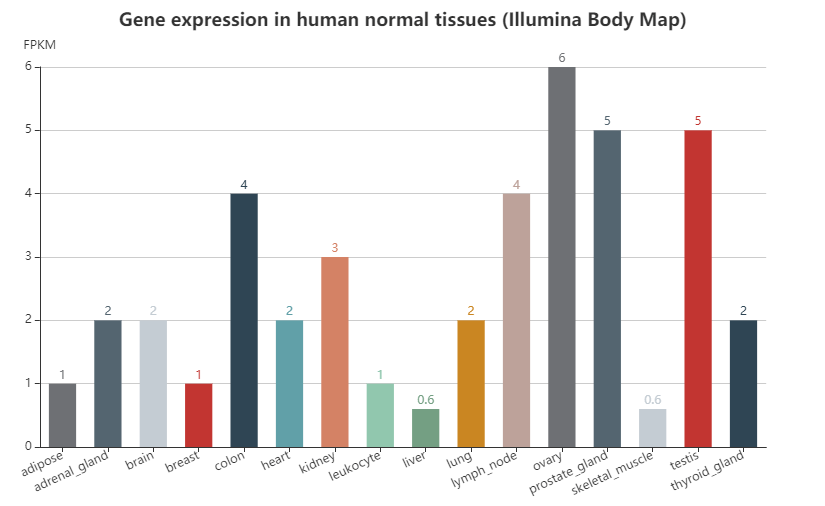
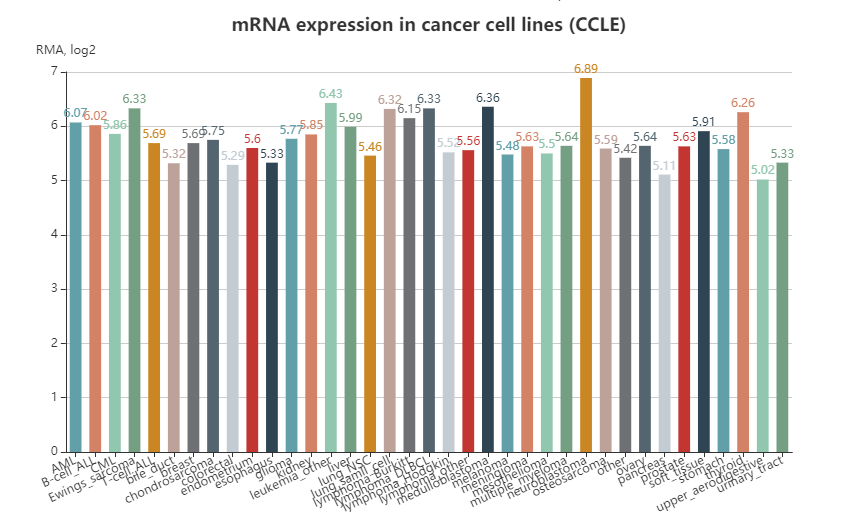
Part4--Expression,正常或癌细胞组织中的基因表达:

上面这些内容是针对表达的内容,我们还可以单击USCS基因组浏览器进行查阅。
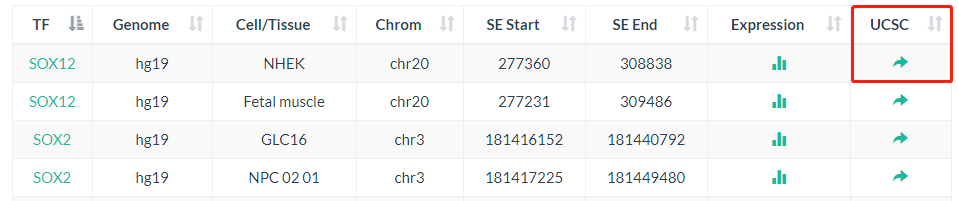
点击后我们可以在USCS基因组浏览器中可视化特定命中SE区域内其他核心TF的推定结合位点:

2
浏览
BROWSE&Visualiza
在“BROWSE”页面中,可以轻松单击或输入搜索词以过滤数据库中包含的样本。浏览页面的左侧面板被组织用于基于物种,生物样品类型,组织类型和细胞类型进行样品过滤,如下:

左上方的“搜索”框还可用于查找感兴趣的样本。通过单击样本名称,对应样本的CRC模型将显示为互连回路的交互图像和核心TF列表。可以通过单击找到各个核心TF的表达模式。可以在USCS基因组浏览器中查看特定核心TF SE区域内核心TF的推定结合位点,和上面搜索得到的结果页面的内容一致。比如任意点击的6A:
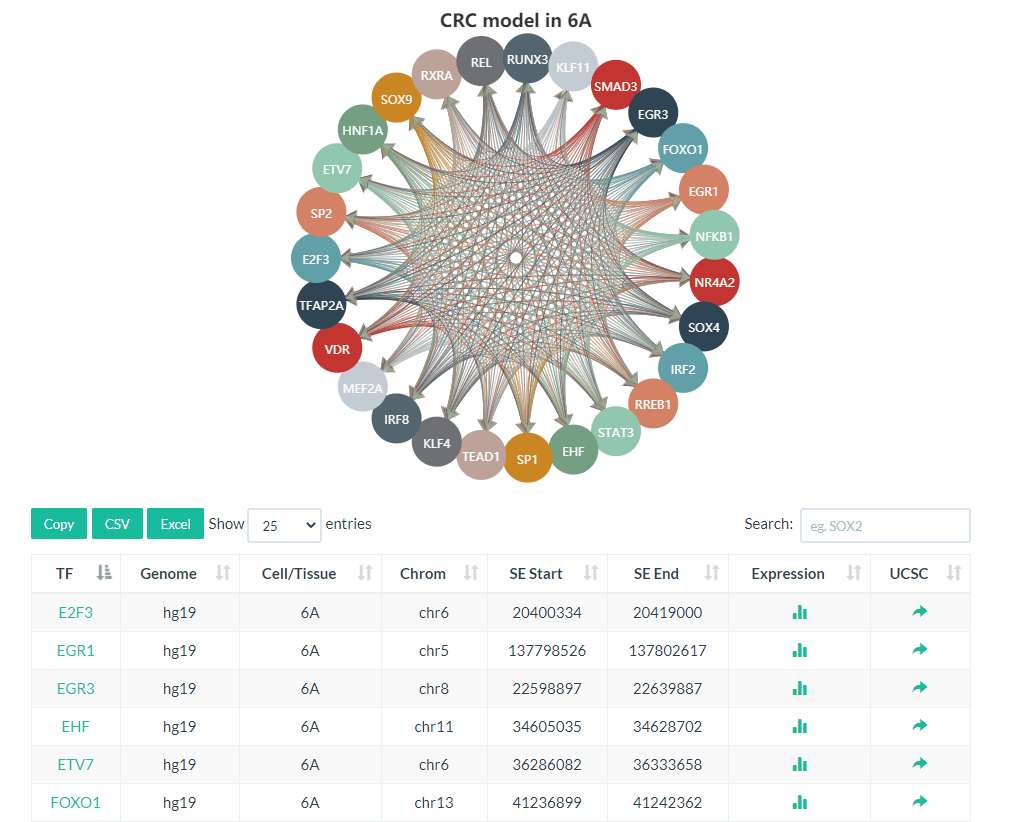
在Visualiza模块也是类似的,但这里只有物种和细胞的筛选:

如果进一步单击感兴趣的核心TF将打开一个新选项卡,其中显示样本信息,TF基因的一般说明,CRC模型内的下游靶标和上游调节剂,以及正常或癌细胞/组织中的基因表达。
3
DOWNLOAD
下载核心转录因子数据
当我们设想一些转录核心因子在生物学研究中的几种潜在应用,这些研究与但不限于超级增强子谱系指定过程中的主TF开关以及疾病特异性转录网络有关时,可以下载这些数据进行使用。
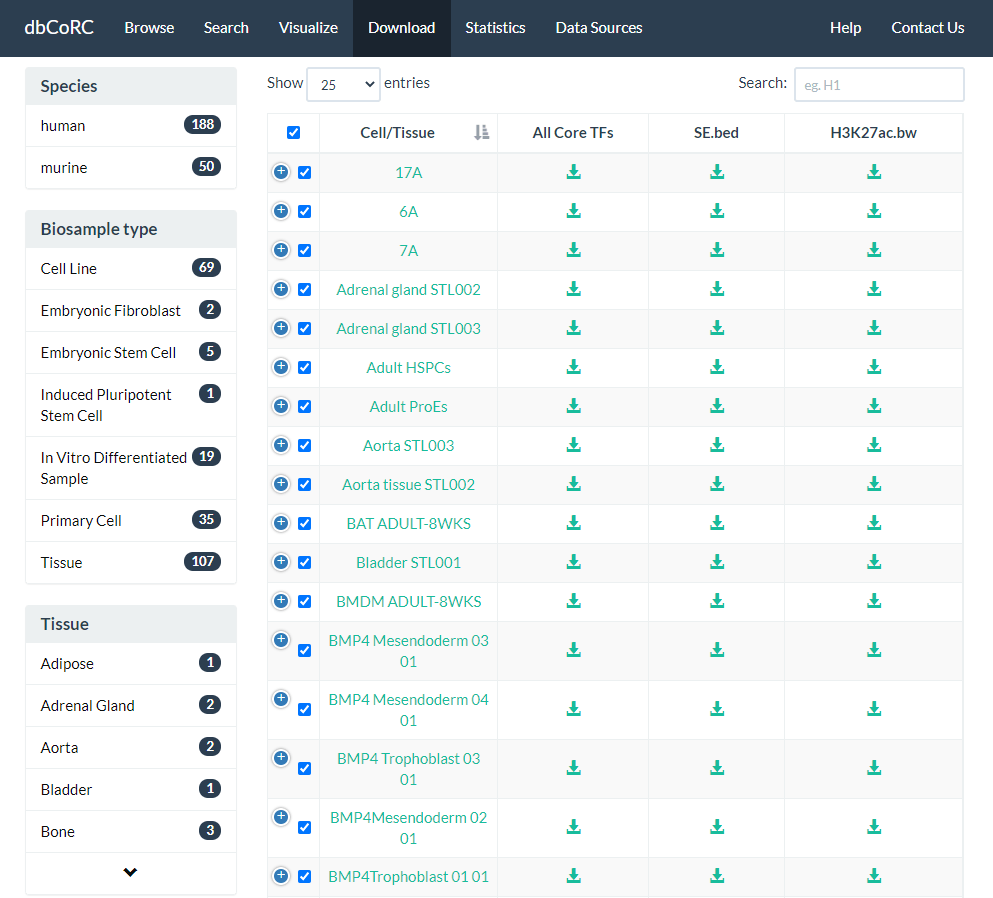
在“DOWNLOAD”页面中,数据库提供每个样本的所有可下载文件,包括核心TF结合位点(.bed),SE注释(.bed),已处理的H3K27ac ChIP-seq信号(.bw)和峰注释(.xlsx)的Zip文件,方便轻松搜索和下载这些数据用于内部和/或深入的生物信息学分析。可以将这些数据与其他bw文件(例如,NANOG,SOX2 ChIP-Seq数据)一起导入IGV,以探索整个SE区域的TF绑定功能。比如dbCoRC默认设置下显示每个样本的CRC模型。我们可以比较同一组织来源的非肿瘤样本和肿瘤样本之间核心TF的差异状态。
总的来说,通过dbCoRC我们能够迅速找到基因对应的核心转录因子,查看相应的核心转录因子构成的调控网络,超级增强子区域以及在其他转录因子SE区域的结合位点、不同表达量信息。这也是dbCoRC的特色,侧重于分析超级增强子区域关联的转录因子间的调控关系。本期内容就到这里,希望大家使用能够愉快使用dbCoRC~
References
Saint-Andre, V., Federation, A.J., Lin, C.Y., Abraham, B.J., Reddy, J., Lee, T.I., Bradner, J.E. and Young, R.A. (2016) Models of human core transcriptional regulatory circuitries. Genome Res, 26, 385-396.
- 本文固定链接: https://maimengkong.com/zu/1387.html
- 转载请注明: : 萌小白 2023年3月2日 于 卖萌控的博客 发表
- 百度已收录
