图中横坐标为基因位置,纵坐标为reads的覆盖深度,它可以反映目的蛋白分布情况。input由于没有富集,其reads通常分布较低,而IP对目的蛋白及其结合序列的富集作用则会使reads覆盖深度提高,产生相应的结合峰。TSS上下游通常是组蛋白和转录因子主要结合、调控的区域,所以也是大量出现结合峰的区域,那么我们从这一张图,就能判断ChIP测序是否成功,钱花得值不值。
更有意思的是,我们在多个H3K4me3的ChIP测序项目中都观察到了相似Peak分布图:TSS附近出现非常明显的结合峰,长度约2kb。
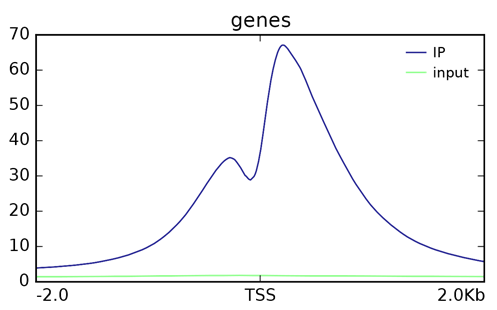
(项目KC2018-CXX,动物组蛋白H3K4me3)
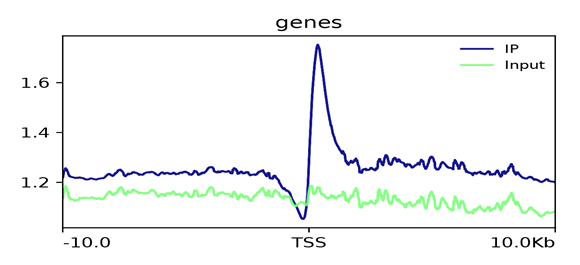
(项目KC2018-CXX,动物组蛋白H3K4me3)
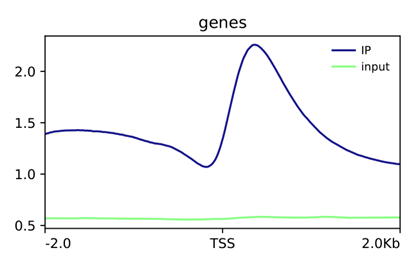
(项目KC2018-CXX,植物组蛋白H3K4me3)
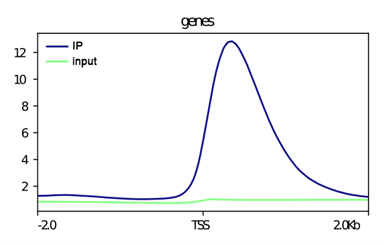
(项目KC2018-CXX,植物组蛋白H3K4me3)
这与文献中报道的H3K4me3在基因上的分布特征相吻合,集中分布在TSS富集。
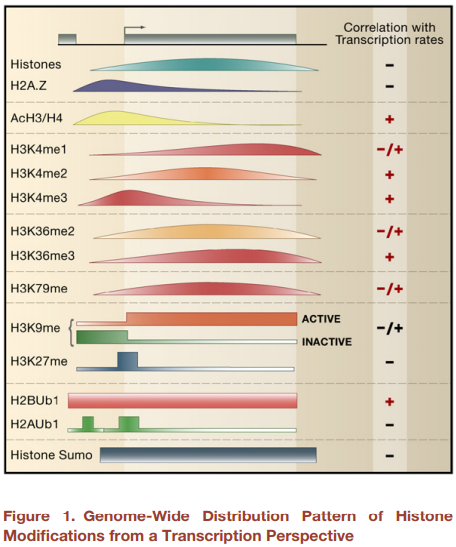
(Li B , Carey M , Workman J L . The Role of Chromatin during Transcription[J]. Cell, 2007, 128(4):0-719.)
而在转录因子的ChIP测序项目,我们发现,Peak在TSS上下游的分布则不太相同。
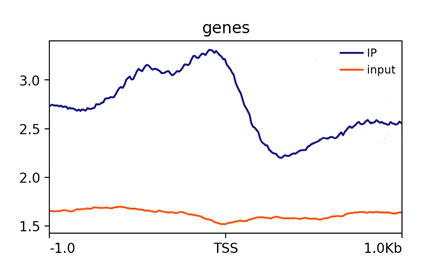
(项目KC2017-BXX,动物某转录因子ChIP测序,peak主要位于TSS上游)
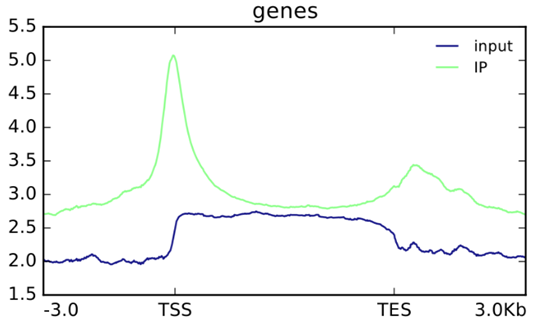
(项目KC2018-CXX,动物某转录因子ChIP测序,peak主要位于TSS附近和TES下游)
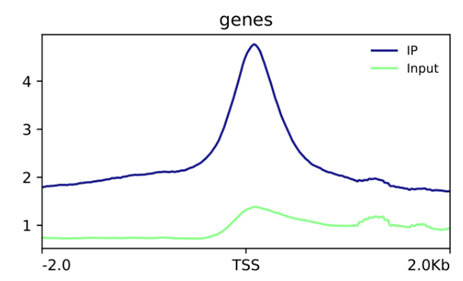
(项目KC2018-CXX,植物某转录因子ChIP测序,peak主要位于TSS上下游)
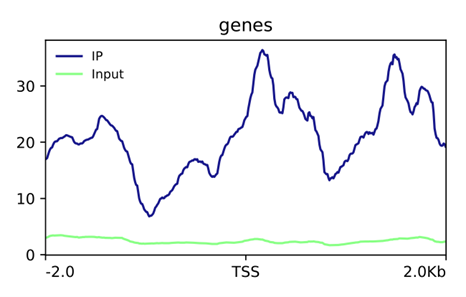
(项目KC2018-CXX,动物某转录因子ChIP测序,TSS及上下游较大范围内有不规则的Peak)
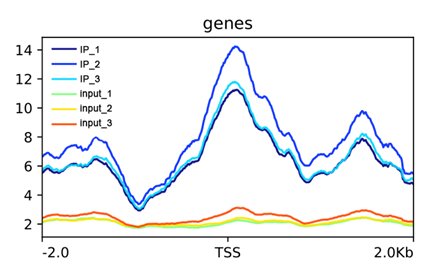
(项目KC2018-CXX,动物某转录因子ChIP测序,3个生物学重复,TSS及上下游较大范围内有不规则的Peak)
细细想来,这正是转录因子的结合特性所至,在转录起始位点上游有启动子、增强子等调控元件,这些区域正是转录因子结合、发挥作用的位置,而在转录终止位点之后也有诸如沉默子、绝缘子这样的调控元件,也是某些转录因子结合的位置,不同的转录因子结合、调控不同的序列、元件,那么我们看到的reads分别自然有所差别了。这不也正是我们想要探索、阐明的转录因子特性及调控方式吗?
- 本文固定链接: https://maimengkong.com/zu/1340.html
- 转载请注明: : 萌小白 2023年1月14日 于 卖萌控的博客 发表
- 百度已收录
