ATAC-Seq简介
ATAC-seq(Assay for Transposase-Accessible Chromatin with high throughput sequencing) 是2013年由斯坦福大学William J. Greenleaf和Howard Y. Chang试验室开发的用于研讨染色质可及性(通常也了解为染色质的敞开性)的办法, 原理是经过转座酶Tn5简单结合在敞开染色质的特性,然后对Tn5酶捕获到的DNA序列进行测序。
真核生物的核DNA并不是裸露的,而是与组蛋白结合构成染色体的根本结构单位核小体,核小体再经逐渐的紧缩折叠终究构成染色体高级结构(如人的DNA链完整展开约2m长,经过这样的折叠就变成了纳米级至微米级的染色质结构而能够储存在小小的细胞核)。而DNA的仿制转录是需求将DNA的严密结构翻开,然后允许一些调控因子结合(转录因子或其他调控因子)。这部分翻开的染色质,就叫敞开染色质,翻开的染色质允许其他调控因子结合的特性称为染色质的可及性(chromatin
accessibility)。因此,以为染色质的可及性与转录调控密切相关。
敞开染色质的研讨办法有ATAC-seq以及传统的DNase-Seq及FAIRE-seq等,ATAC-Seq由于所需细胞量少,试验简单,能够在全基因组范围内检测染色质的敞开状况,现在已经成为研讨染色质敞开性的首选技能办法。
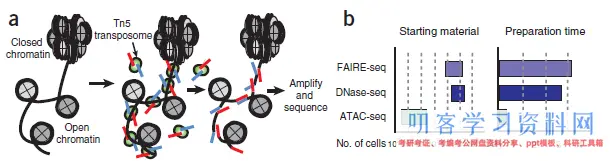
Nat Methods, 2013. doi: 10.1038/nmeth.2688. Epub 2013 Oct.
ATAC-Seq与ChIP-Seq的异同
ATAC-Seq与ChIP-Seq的不同的是ATAC-Seq是全基因组范围内检测染色质的敞开程度,能够得到全基因组范围内的蛋白质或许结合的位点信息,一般用于不知道特定的转录因子,用此办法与其他办法结合筛查感兴趣的特定调控因子;但是ChIP-Seq是明确知道感兴趣的转录因子是什么,依据感兴趣的转录因子设计抗体去做ChIP试验拉DNA,验证感兴趣的转录因子是否与DNA存在彼此作用。ATAC-Seq、ChIP-Seq、Dnase-Seq、MNase-Seq、FAIRE-Seq全体的剖析思路共同,找到富集区域,对富集区域进行功用剖析。
- ChIP-Seq是揭示特定转录因子或蛋白复合物的结合区域,实践是研讨DNA和蛋白质的彼此作用,利用抗体将蛋白质和DNA一同富集,并对富集到的DNA进行测序。
- DNase-Seq、ATAC-Seq、FAIRE-Seq都是用来研讨敞开染色质区域。DNase-Seq是用的DNase I内切酶识别敞开染色质区域,而ATAC-seq是用的Tn5转座酶,随后进行富集和扩增;FAIRE-Seq是先进行超声裂解,然后用酚-氯仿富集。
- MNase-Seq是用来判定核小体区域。
翻译部分
下面这一部分是对HBC课程中ChIP-Seq
Introduction这一节的介绍,主要包含的ChIP-Seq的试验设计和剖析办法总体思路。原文链接:https://github.com/hbctraining/In-depth-NGS-Data-Analysis-Course/blob/master/sessionV/lessons/01_Intro_chipseq_and_setup.md。
ChIP-Seq Introduction
学习方针
-
了解ChIP-Seq的试验设计
ChIP-Seq简介
ChIP试验(Chromatin immunoprecipitation)即染色质免疫沉淀,依据DNA与蛋白质彼此作用的原理,别离富集与感兴趣的蛋白彼此作用的DNA。ChIP-Seq即对别离得到的DNA扩增测序,然后经过剖析得到DNA的富集区域也称为peaks,同时能够判定过表达的序列motif以及进行功用注释剖析。
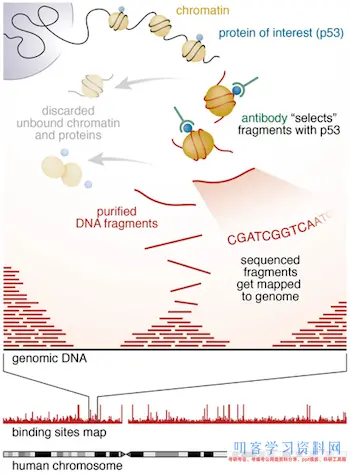
下面这一部分将会介绍ChIP-Seq数据剖析的整个流程,从试验设计到发生原始的测序reads,以及到最后的功用富集剖析和motif查找。
试验设计和文库构建
文库构建包含以下5过程:
- 蛋白质与DNA的交联
- 超声打断DNA链
- 加附有抗体的磁珠用于免疫沉淀
- 解交联,纯化DNA
- DNA片段巨细挑选和PCR扩增
富集到的DNA片段只要一部分是真实的信号(感兴趣的蛋白结合的DNA区域),这个份额取决于number of active binding sites, the number of starting genomes, and the efficiency of the IP.
ChIP-Seq富集序列存在以下特点:
- 敞开染色质区域比严密区域更易打断;
- 重复序列会呈现似乎被富集的现象
- 序列在整个基因组上不均匀分布
因此,ChIP-Seq需求有适宜的对照组,对照样本需求满意以下其中一个条件:
- 没有IP(input DAN)
- 没有抗体 (“mock IP”)
- 没有特定的抗体 (IgG “mock IP”)
示例数据介绍
所用到的示例数据是来自于人类胚胎干细胞系(h1-ESC)中Nanog和Pou5f1(Oct4)两个转录因子的结合图谱。这两个转录因子的功用涉及干细胞的多能性,该研讨的方针之一是探求这两个转录因子在转录调控中独自和彼此的调控作用。
两组重复,每组重复包含3个试验样本信息,共6个样本,数据剖析中只用到了12号染色体的信息。
Nanog IP
Pou5f1 IP
Control input DNA
剖析流程
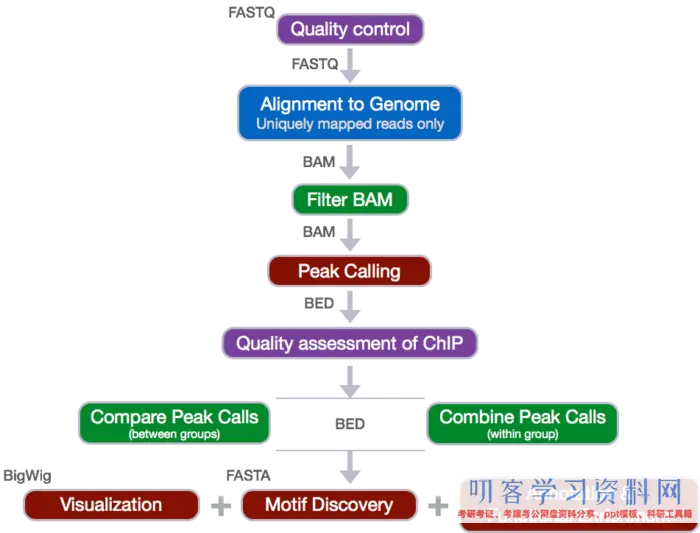
下面这幅图给出了整个剖析流程,和每一步需求的数据格式,后面会展开介绍。
剖析环境装备
这个课程提供了示例数据和剖析代码,能够参考这里衔接他们的服务器,我没有衔接成功,不知道是不是翻开方式不对,我们能够尝试下,假如衔接成功,这一部分便是装备服务器的环境,准备数据;假如也衔接不上能够用自己的数据或者下载公共数据。
- 本文固定链接: https://maimengkong.com/zu/1324.html
- 转载请注明: : 萌小白 2023年1月2日 于 卖萌控的博客 发表
- 百度已收录
