1. 四种基因分型技术简要比较
对于GWAS 研究,关键就是需要对目标群体的每个个体开展全基因组水平的基因型检测。4种测序策略所能检测的区域有所区别,如下图示意图所示。
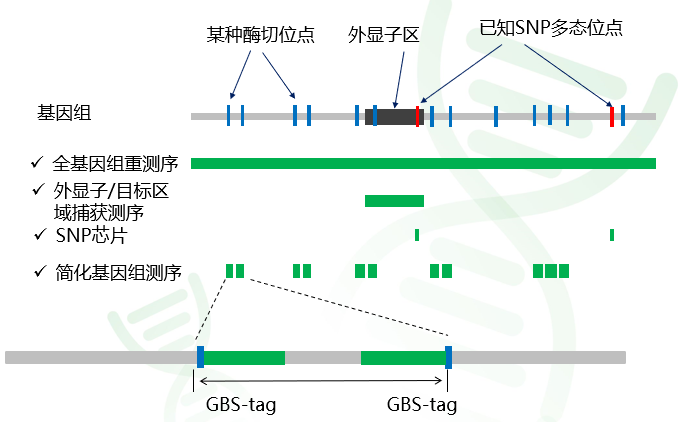
图1 四种基因分型策略检测区域的示意图,图中绿色长方块代表对应的测序技术所能检测的区域
(1 )全基因组重测序
理论上可以检测基因组范围内所有位置的突变信息,常见检测的突变类似包括:SNP 、短插入缺失(short indel)和CNV。其他结构性变异,例如大的插入、倒装、异位等突变,二代重测序检测能力则比较弱,需要依赖于三代测序才能较好检测。
(2 )外显子/目标区域捕获测序
该技术主要利用目标物种的DNA 序列设计核酸探针,然后与所要检测的样本DNA进行杂交,来钓取富集与探针可以结合基因组DNA,然后进行测序。该技术目前的应用策略主要有3种类型:
(a )外显子捕获测序
如果基于外显子区域设计探针,则捕获区域为外显子区,对应就是外显子捕获测序。检测区域大概是在40Mb~70Mb 的范围(外显子以及上下游区域)。
(b )特定区段捕获测序
如果基于基因组特定区段的序列,则捕获区域为对应区段。比如,你只关注1 号染色体上4Mb~5Mb区间的序列,那么就可以用这个区域的序列定制探针,进行捕获测序。
(c )对SNP标记上下游100bp左右的范围进行捕获测序
这种应用思路和下面要提到的SNP 芯片非常相似,但不同的地方是这里是把已知存在SNP的区间抓取出来,只对这个区域进行测序,从而对SNP标记进行基因型检测。如果你抓取10k个SNP区间,就相当于可以对10K个SNP位点进行基因分型。由于成本考虑,应用该策略的基因分型产品,一般会将SNP数量控制在50k以内。
以上3 种策略里,都是只对自己关注的区域进行捕获测序,从而降低了测序量和成本。a和c都可以用于全基因组关联分析。其中,外显子捕获测序在人类疾病研究中应用非常普遍。而c策略,在动植物育种研究中应用也很常见,因为该策略可以较好满足位点定制调整的需求。
(3 )SNP芯片
该技术基于已知的SNP 位点设计探针,然后让探针与检测样本的基因组DNA进行杂交。SNP探针在与SNP位点序列杂交后,遇到不同的基因型会产生不同的颜色(比如,A基因型是绿色,T基因型是红色),从而不需要测序就实现基因分型。
该技术主要的供应商是illumina(illumina实际上做芯片起家的,测序仪业务是后来收购的)和affymatix。SNP芯片适合量产,不适合定制(定制起订量高,且成本高昂)。因此SNP芯片比较适合做成熟固定的商业化芯片,例如针对人类、牛、羊、水稻等物种。商业化芯片的SNP密度一般在10k~1M范围。
(4 )简化基因组测序
该技术要利用限制性内切酶对样本DNA 进行酶切和富集,然后对酶切位点附近的DNA片段进行测序。该类技术会细分出不同的建库策略,对应不同名称(比如GBS、RDA),但整体上大同小异。该类技术可以通过调整内切酶的种类,对富集区域进行调整,一般可以对基因组2~10%酶切位点附件的区域进行测序,从而检测这些区域的SNP信息,检测到的SNP数量从10k~100k范围。
简化基因组测序的优势就是成本比较低,且甚至不需要参考基因组也可以进行分析,所以对非模式物种的大群体研究非常适合。对应的缺点主要是由于采用酶切的策略,所以检测稳定性不如以上的3 种技术,检测出的SNP位点有较高的缺失率。且只能检测酶切位点附近的SNP,而无法定制选择特定区域的SNP。
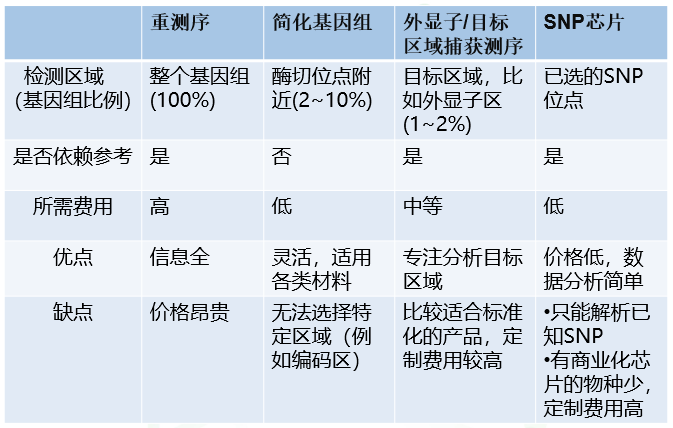
表1 四种基因型分析技术的比较
下面这里我们将介绍和比较4 种全基因组高通量基因分型策略在GWAS应用中的特性进一步进行详细解析和比较。
2. 简化基因组测序
简化基因组测序由于其无需定制、低成本,在非模式生物研究中非常受欢迎。不过简化基因组策略,还是比较适合用于遗传图谱构建或群体遗传分析,不是十分适合用于GWAS 研究。主要原因是:
1. 标记密度有限,且标记分布均一性无法保证
简化基因组策略在自然群体可以获得几万个标记。如果是1 个1G基因组大小的物种,标记间的平均间距大概是几十K的水平。而且,由于酶切位点在基因组上的位置分布并不是均匀分布的,这会导致某些缺乏酶切位点的区间标记密度更低。标记越稀疏,那么标记与潜在QTL的连锁不平衡值(r 2)越小。所以,用简化基因组策略进行GWAS 研究,QTL被漏检的概率很大。
2. 没有参考基因组的情况
简化基因组测序,是唯一一种不需要参考基因组也可进行基因分型的策略。但没有参考的简化基因组测序,对GWAS 研究却没有太大价值。即使我们分析得到了与性状关联的标记,也不知道与标记连锁的潜在基因都有哪些(因为没有参考基因组),而找到候选基因是GWAS研究的重要目的。
所以,简化基因组测序更适合进行遗传图谱构建、群体遗传分析、全基因组选择育种等这些不那么关注找到具体基因的研究方向。
3.SNP芯片
SNP 是芯片都模式物种来说,是性价比较高的大群体基因分型策略。在人类、牛等这些模式物种,高密度SNP芯片的密度基本上都到了800k~1M的水平,且标记都是精心挑选的好的,基本在基因组上均一分布。这就保证了任何1个候选QTL与最近的SNP的连锁不平衡值(r 2)可以大于0.8 ,从而降低GWAS分析QTL漏检的可能性。所以在人类GWAS研究中,高密度芯片应用的风生水起。
但如果使用中低密度的芯片(SNP密度在1~50k)开展GWAS研究,则有较大QTL漏检的可能性。另外,由于SNP芯片都是基于已知的SNP位点进行设计,所以不能用于发现新的突变。在得到关联标记后,还是需要进行测序去发现与关联标记连锁的潜在功能突变。
4.外显子/目标区域捕获测序
外显子捕获测序在人类疾病研究中是一项常用的策略。对于外显子区研究来说,外显子捕获测序和重测序几乎没有差别(除了少了外显子区不易设计探针而无法捕获)。所以在人类研究中,既关注新生突变(这意味着需要测序)又有较大样本量的情况下(例如,大样本肿瘤研究),外显子测序是一种高性价比的选择。但外显子测序也有其不足:
①实际上外显子捕获测序只能覆盖全外显子的85%~95% ,还是有些区域漏检;
②一般没有mtDNA 等这些特殊区域的覆盖;
③CNV 和SV检测能力不足;
④无法检测基因区外的信息,比如非编码RNA 、启动子区。而大部分影响复杂疾病的QTL,实际上位于基因区之外的调控元件中,而这些区域外显子测序无法覆盖。
另外一种捕获测序策略,如我上文刚刚提到的“ 对SNP标记上下游100bp左右的范围进行捕获测序“,则重要受限于成本,一般检测的SNP数量在50k以内。对于GWAS研究来说,还是有标记密度偏低的问题。”
5.全基因组重测序
全基因组重测序是全基因组水平最全面检测突变的技术,毫无疑问是开展GWAS 研究最好的技术。除了唯一的缺点的贵,其他方面都是优点。但由于GWAS研究一般仅用中等深度的重测序(10X左右)就足以开展研究,在重测序成本不断降低的情况下,这个成本也慢慢可以接受了。所以,现在国际上大样本人群的研究也逐渐使用全基因组重测序替代外显子测序或SNP芯片。
重测序最大的优点是检测的突变信息全。
(1 )检测的变异种类全,除了我们通常关注的SNP、indel之外,还可以开展CNV的检测。利用CNV开展GWAS研究,也是一种很有趣的选择;
(2)检测的标记密度大。目前GWAS研究主要还是基于SNP和indel的标记。对于SNP和indel检测,重测序可以做到接近饱和密度的检测。如同我们上文提到过的,足够密度的标记,可以保证标记与潜在QTL之间保持强连锁(连锁不平衡r 2≥0.8 ),从而提高对QTL的检出能力。
这里要解释一点,虽然QTL 的存在几乎必然是对应某种功能性变异的存在,但除了SNP,大的结构变异,例如CNV、插入、倒位都是可能的功能突变。由于二代重测序只是对SNP和short indel有较强的检测能力,其他类型的变异则检出能力较弱。所以,基于重测序的GWAS也只能保证与足够高的标记把强关联的标记找到,但也无法保证找到的最强关联标记,就是决定QTL的功能标记本身。
比如在西红柿果色GWAS 研究论文中[1],利用重测序作者定位到1个与果色强关联的SNP(下图)。但这个SNP并非功能突变,相关的功能突变还是通过对部分材料开展一代测序后才发现。分别是基因启动子区一段603bp的插入/缺失,以及基因编码区的两个单碱基突变。
至于重测序为什么没有检测到这些功能突变呢?前者属于结构变异的范畴,重测序的检出能力相对较弱。后者两个SNP可能是在群体中频率较低,也降低了被检测出的概率。其他在玉米[2]、水稻[3]等研究中,也是重测序GWAS仅仅定位了关联SNP,实际的功能突变也是需要配合一代测序和功能验证才可以确定。
以上的案例并不是说要贬低重测序的价值。首选,重测序还是有很大概率可以直接检测到功能突变。其次,哪怕没有直接检测到功能突变,还是基本可以保证总是有与功能突变连锁足够强的SNP 标记,来保证来自功能突变的QTL信号可以被检测到。
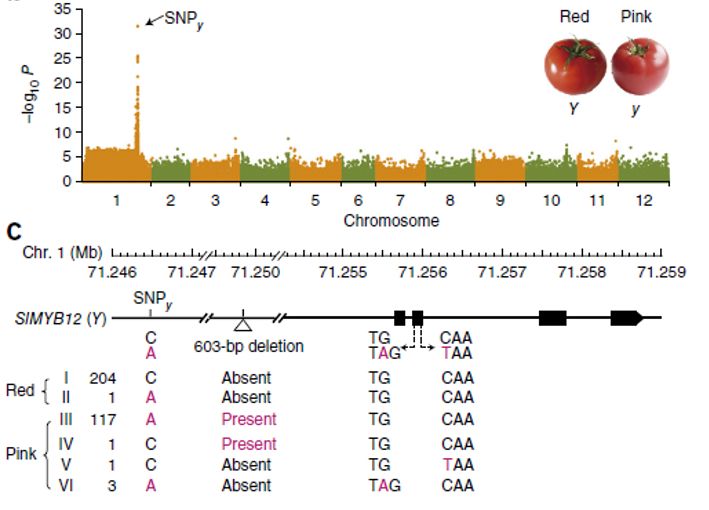
图2 GWAS 定位关联标记以及利用一定测序挖掘功能突变
- 本文固定链接: https://maimengkong.com/learn/855.html
- 转载请注明: : 萌小白 2022年3月26日 于 卖萌控的博客 发表
- 百度已收录
