单核苷酸多态性(single nucleotide polymorphism,SNP)主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。它是人类可遗传变异中最常见的一种,占所有已知多态性的90%以上。SNP研究是人类基因组计划走向应用的重要步骤,这主要是因为SNP将提供一个强有力的工具,用于高危群体的发现、疾病相关基因的鉴定、药物的设计和测试以及生物学的基础研究等。
经过近年检测技术的发展,科研人员可以通过NGS或SNP芯片筛选与疾病关联基因或区段,进而针对这个特定区段或基因上的SNP进行更为仔细研究,也有科研人员会通过已发表文章查询到与其研究相关的基因,再通过对该基因的DNA序列变化分析遗传机制,但如何查询特定区段或基因上的有用SNP位点呢?是很多刚接触科研的师弟师妹们的苦恼,小忆秋在此分享一下个人经验,供参考哈~
本人查询关注基因的SNP信息,一般会通过NCBI数据库进行,首先打开NCBI数据库链接:http://www.ncbi.nlm.nih.gov/,在信息栏选择gene选项,在搜索栏中输入需要检索的基因name或ID,假如我们查询human的APC基因(不知道该基因的ID号),
输入后点击search,会出现如下界面:
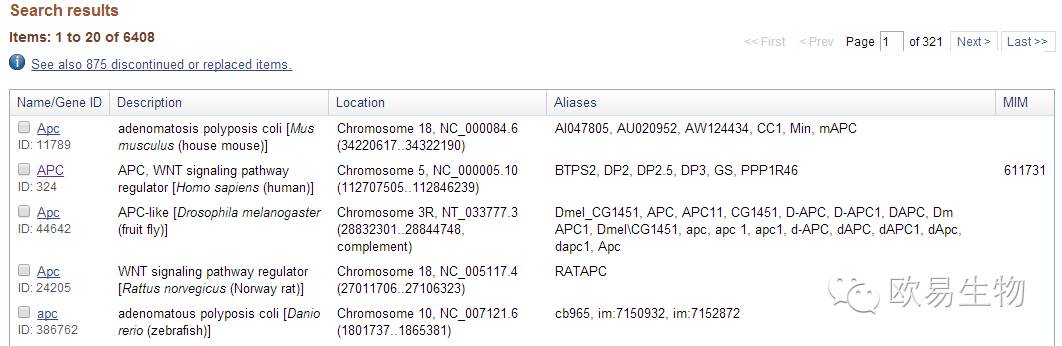
结果中会包含不同物种的APC基因信息,我们选择human的一项点开,即可获得该基因的相关信息,如下图。如果我们知道要查询基因ID号,直接在搜索栏中输入ID,例如human的APC基因,gene ID为324,点击search,会直接出现一样的搜索结果。

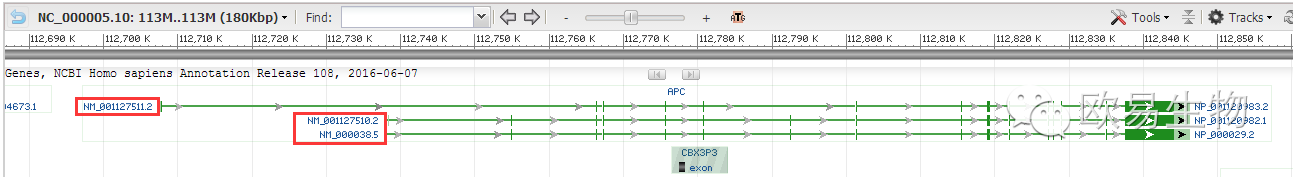
将以上页面下拉,可查询该基因的相关信息,其中有一项,如下图显示,可看出该基因有3个转录本(分别为NM_001127511.2、NM_001127510.2和NM_000038.5),如果需要对转录本层面进行SNP查询,可在随后操作进行区分。
继续浏览页面右侧信息,可以发现SNP: GeneView选项,

点开,即可获得该基因SNP的所有信息(见下图)。其中红色框中描述了该搜索结果显示SNP的参考基因组版本以及如何搜索其他版本基因组信息介绍;粉色框中包含的即为上面所述的不同转录本信息,共有6条记录,其中前3条记录为后3条记录的未更新链接,后3条记录可以与我们刚才看到的3条转录本号一一对应;深绿色框中限定显示SNP的条件,一般我们常选择cSNP(编码区的SNP位点)。至于为什么选择cSNP呢,文章最后有介绍哦~

如果您没有特别关注的转录本,那可以选择NM_001127511.2,这个转录本最长,对应的DNA序列最长,可以含有更多SNP信息,具体操作是点击 NM_001127511.2最右侧的View snp on GeneModel即可, 如下图,想看哪条转录本信息,就点击哪条即可。

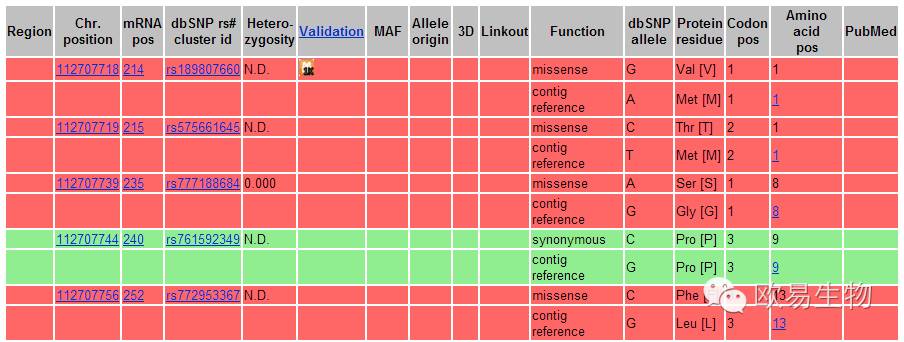
这样输出的结果即为需要查看转录本的SNP信息(如下图)。其中红色底纹的是错义突变,绿色底纹的为同义突变。如果想要看每一个SNP的具体信息,可以点击rs号查看。每一个rs号都会有两行记录,其中Chr. Position是SNP位点所在染色体位置;mRNA pos是SNP位点所在mRNA序列上的位置;dbSNP rs# cluster id是SNP的ID号;Function是突变类型或SNP所在区域(是非编码区还是编码区,是同义突变还是错义突变);db SNP allele是碱基变化情况,其中第一行是突变后的碱基,第二行是参考碱基;Protein residue是氨基酸变化情况,其中第一行是突变后的氨基酸,第二行是参考序列氨基酸;Codon pos是指突变的碱基位于组成氨基酸的3个密码子中的哪一个位置,Amino acid pos是突变的氨基酸位于该蛋白氨基酸序列的位置。
按照上面的办法,挑选错义突变的SNP位点即可,如果还是觉得位点比较多,可以根据MAF值进行筛选。
Ps:为什么选择错义突变的cSNP研究呢?
cSNP是位于编码区内的SNP,比较少。从对生物遗传性状的影响上来看,cSNP又可分为2种:一种是同义cSNP(synonymous cSNP),即SNP所致的编码序列的改变并不影响其所翻译蛋白质的氨基酸序列,突变碱基与未突变碱基的含义相同;另一种是非同义或错义cSNP(non-synonymous cSNP或missense cSNP),指碱基序列的改变可使以其为蓝本翻译的蛋白质序列发生改变,从而影响蛋白质功能,这种改变常是导致生物性状改变的直接原因。因此错义突变的cSNP位点在遗传性疾病研究中却具有重要意义,也更受关注。- 本文固定链接: https://maimengkong.com/kyjc/1833.html
- 转载请注明: : 萌小白 2024年11月2日 于 卖萌控的博客 发表
- 百度已收录
