“葡萄美酒夜光杯,欲饮琵琶马上催。”葡萄自古以来就是非常重要的水果,无论是直接食用、酿酒还是制成葡萄干,都深受世界各地人们的喜爱,同时葡萄(包括晶莹剔透的葡萄美酒)也经常成为文学家、艺术家笔下描绘的对象。时至今日,人们对葡萄的青睐有增无减,于是培育出更加美味的葡萄也就成了当务之急。对于那些重要的果实性状,若能将它们在基因组上进行定位,育种工作将会事半功倍。对于自然群体的性状定位而言,GWAS(Genome-wide association study,全基因组关联分析)是最直接、快捷的方法,而GBS简化基因组测序能够更进一步为性状定位提供便利。河南科技大学的研究者利用GBS-GWAS方法对葡萄的多种性状对应的QTL在基因组上进行了定位,未来的葡萄会不会风味更佳?我们可以一起期待一下。

深受人们喜爱的葡萄
1.材料与方法
研究者收集了179个品系的葡萄,并对这些葡萄品系进行了精细的表型鉴定。鉴定的表型包括果实发育周期(BDP)、果穗大小(CS)、果穗密度(CD)、果实重量(BW)、果肉质地(BFT)、果实颜色(BC)、果实形状(BS)、 果实风味(BF)八种性状。
不同品系的葡萄取嫩叶提取DNA后使用ApeKI酶进行单酶切GBS测序,测序数据比对参考基因组和变异识别使用SOAP软件完成。得到变异信息后,使用ADMIXTURE对群体结构进行分析,并使用PHYLIP构建系统发育树。
进行GWAS分析之前,为了保证分析的精度,对SNP进行过滤,去除缺失比例超过0.05以及最小等位基因频率(MAF)小于0.05的SNP。研究者在比较了多种软件后,最终选定了R包GEMMA作为GWAS分析软件,使用标准线型混合模型进行分析,并使用GEC软件进行阈值计算和数据校正。
2.结果与分析
2.1 GBS测序与群体遗传结构
GBS测序共产生了43.71 GB数据,得到的SNP经过质量过滤后,共有32311个高质量SNP用于后续分析。这些SNP标记覆盖所有19条染色体,标记间的平均距离为14.26 kb。
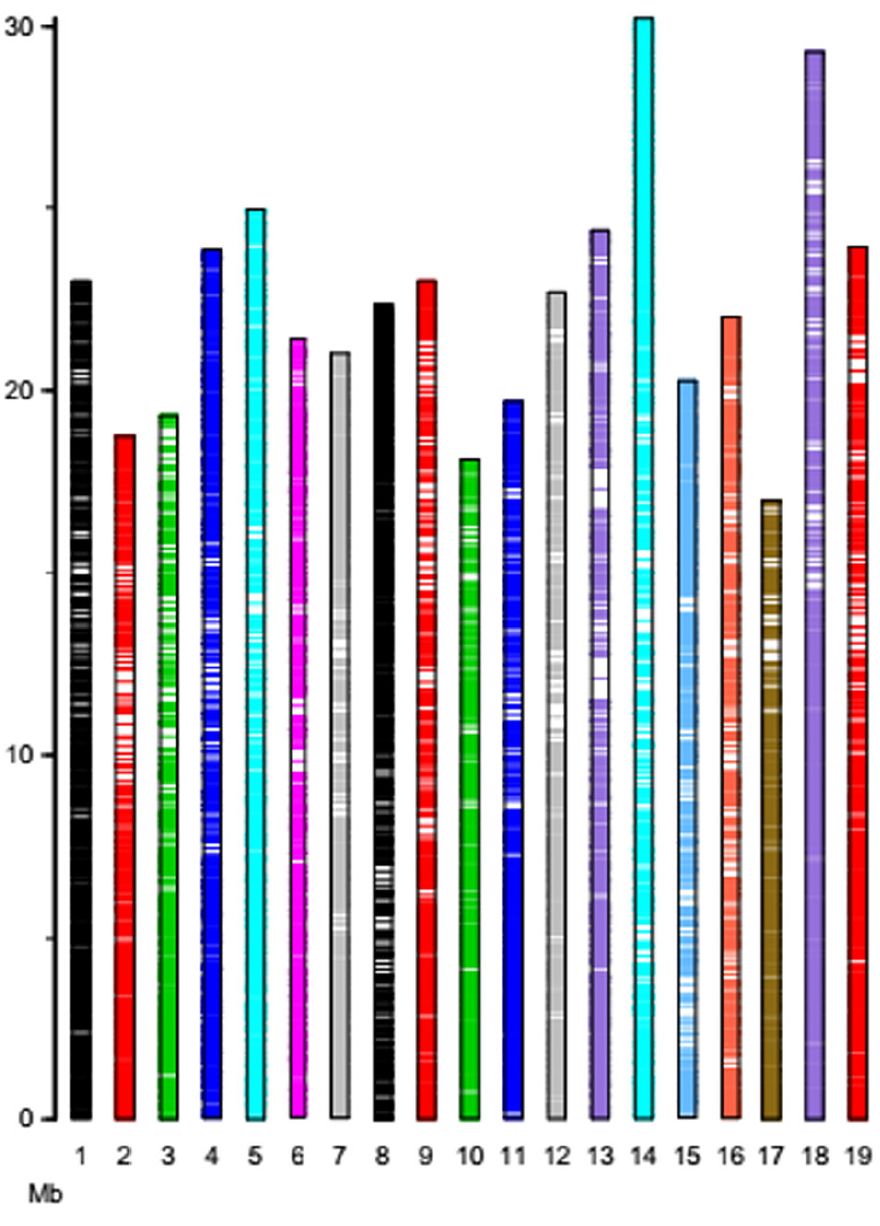
SNP标记在基因组上的分布
使用ADMIXTURE软件使用不同K值对群体结构进行分析,交叉验证误差(cross validation error)分析显示K=8为最优结果。群体结构显示所选择的植物材料没有存在明显的家系分化,适宜进行后续的GWAS分析。系统发育树结果显示所选择的群体可以分为8个亚群,验证了群体结构分析中K=8为最优结果的结论,选择K=8的Q矩阵进入后续的关联分析。同样,PCA结果也验证了所选的材料没有出现明显的群体结构分化,适宜进行GWAS分析。
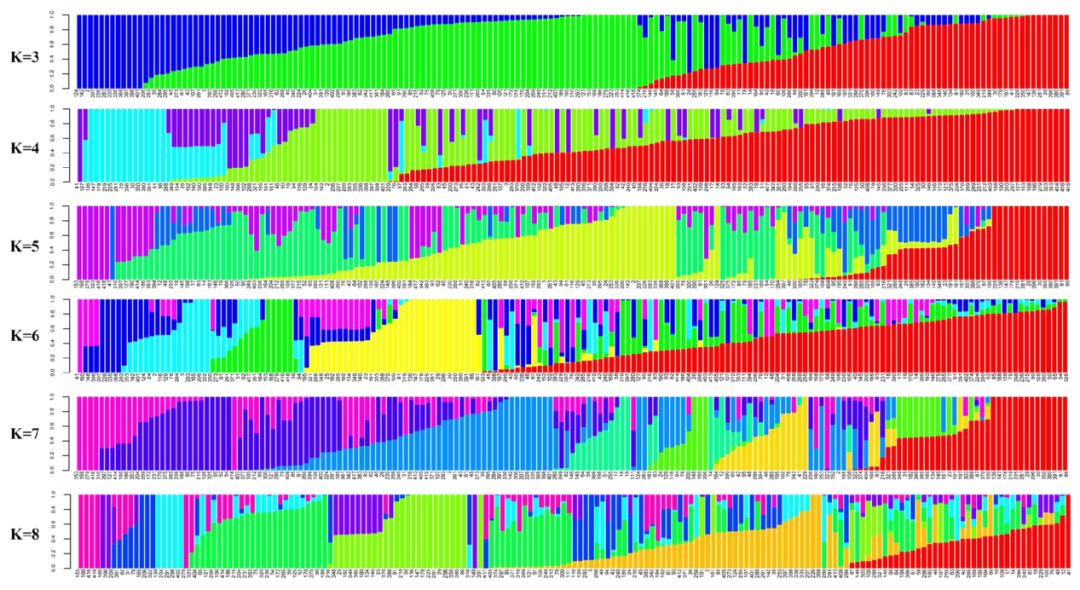
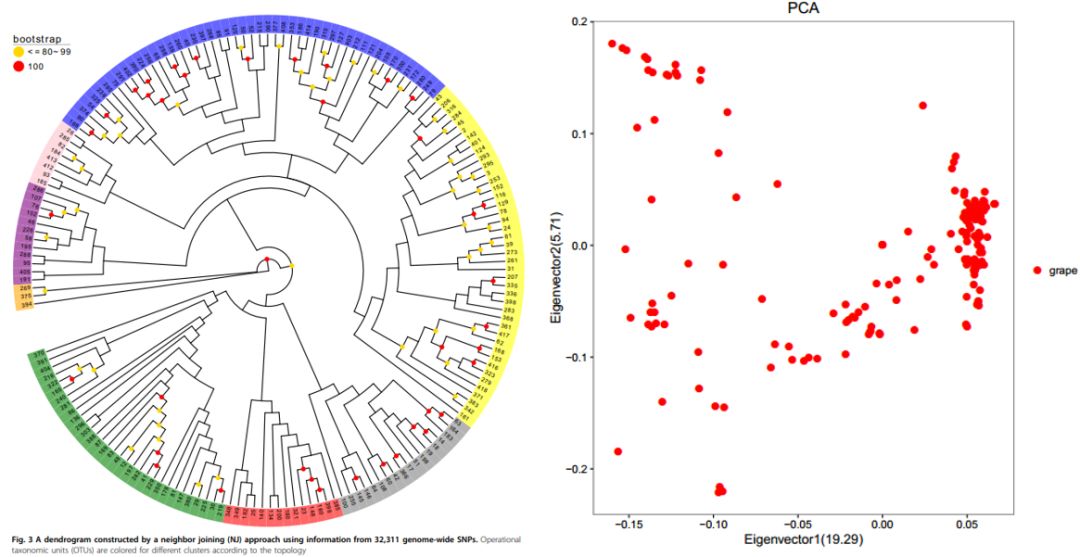
所选材料的ADMIXTURE、系统发育树与PCA群体结构分析结果
2.2 GWAS分析
将SNP标记与性状进行关联后,在所有8种性状中有6种性状发现了显著关联的SNP。与果实颜色、果实发育周期、果穗大小、果实重量、果肉质地以及果实风味相关的SNP标记数量均在1-5个之间,其中与果实重量相关的4个SNP标记分别分布在3条不同的染色体上,而与果实颜色相关的5个SNP标记分布在同一条染色体上。对这些标记周围约1Mb区域内的基因进行分析,从功能角度搜索可能与性状相关的候选基因。
分析找到了一些曾经在其他研究中得到验证的基因,部分SNP标记与之前发现的位点所在的位置存在一定的差异,而另外一些SNP标记则位于之前没有报道过的区域。葡萄的果实颜色曾经被研究认为受到染色体2上MYB转录因子家族控制,而本次研究找到的SNP标记附近找到了基因mybA3,属于MYB转录因子家族,但在之前的研究中未提及mybA3这个特定基因的作用。与果实重量相关的SNP标记发现了一个在18号染色体上曾报道过的位点,以及在17和19号染色体上的两个新位点。
利用GWAS分析,研究者找出了与葡萄的多个性状相关的SNP标记以及对应的候选基因,这项研究增进了对葡萄这一重要作物遗传层面的了解,为未来的育种工作提供了新的思路。
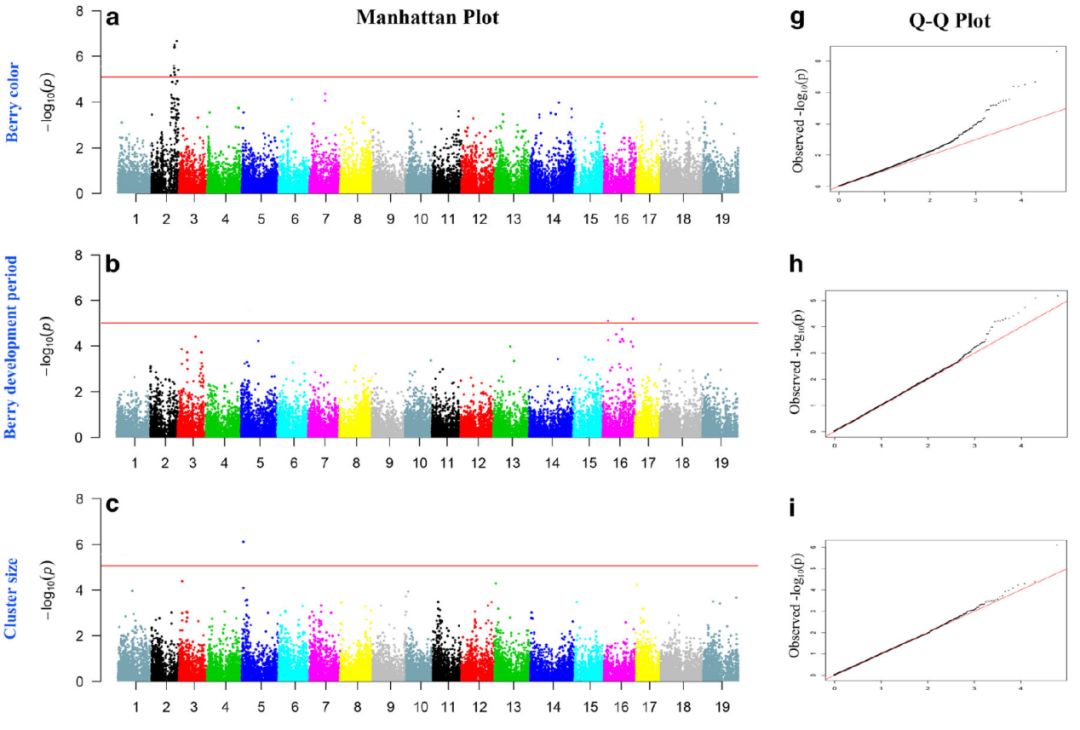
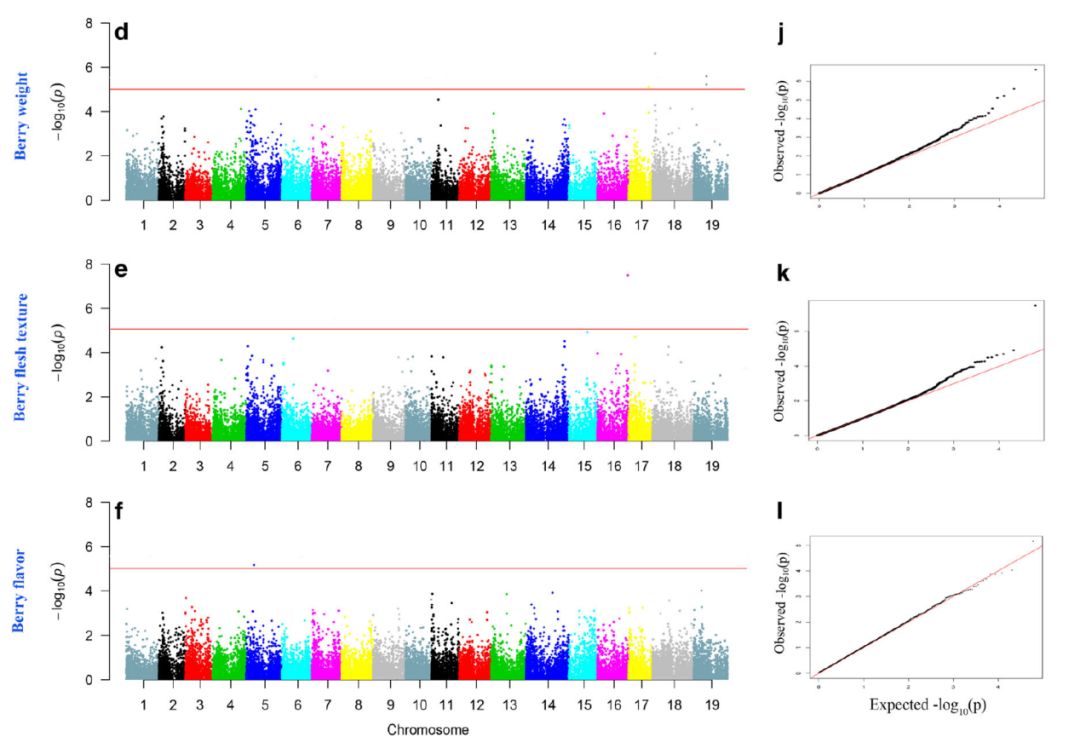
六种性状的GWAS关联分析结果
靠谱er点评
GWAS是对自然群体进行性状定位的重要工具,但需要对每个个体进行测序,测序量大、需要的预算高是GWAS得到更广泛应用所面对的障碍。而GBS简化基因组能很好地解决这一问题,本研究中GBS测序得到了高质量的SNP标记,使得GWAS分析对各个性状都筛选到了对应的显著关联的标记。同时,材料的选取对于GWAS分析的成功进行也必不可少,本文不仅选取了179个不同基因型的品系,并且群体结构分析表明所选取的材料中并不存在明显的聚类,即没有显著的群体结构分化,适宜进行GWAS分析。最后,GWAS的分析方法选择也很重要,本文使用了PLINK、TASSEL、GEMMA、EMMAX四款软件利用不同的数学模型进行GWAS分析,从各个软件的Q-Q plot来看,GEMMA能够最好地匹配本研究的数据。本文最终选择使用R包GEMMA作为分析软件使用混合线性模型进行分析,得到了较为理想的结果。
但是利用GBS测序也会有一些弱点,比如:1. 标记平均密度为14.26 Kb,而分析之后LD衰减值值为16.6 Kb,也就是标记密度不够导致不能捕获到所有的LD block,靠谱er推测这点可能是作者GWAS分析后没有进行LD block进行候选基因筛选的原因;2. 另外,进行关联分析时候部分性状虽然有关联到SNP标记,但是SNP支持度偏少,导致无法准确判断是真实关联还是假阳性SNP。而重测序则可以完美解决这几个问题,事实上,本文合作单位农科院郑州果树所也继续收集资源,利用重测序进行葡萄进化和GWAS研究,成果在本文online两个月后发表在Nature communication上(Liang Z, Duan S, Sheng J, et al. Whole-genome resequencing of 472 Vitis accessions for grapevine diversity and demographic history analyses[J]. Nature communications, 2019, 10.),感兴趣读者可继续深究对比两文的差异。
参考文献
Guo D L, Zhao H L, Li Q, et al. Genome-wide association study of berry-related traits in grape [Vitis vinifera L.] based on genotyping-by-sequencing markers[J]. Horticulture research, 2019, 6(1): 11.- 本文固定链接: https://maimengkong.com/kyjc/1494.html
- 转载请注明: : 萌小白 2023年4月29日 于 卖萌控的博客 发表
- 百度已收录
