分化时间,当前宏观进化分析的一个热点,以某一特定类群的化石记录作为参照点,通过基因序列间的分歧程度以及分子钟来估计速率恒定分支间的分歧时间,同时计算系统发育树上其它节点的发生时间,从而推断相关类群的起源时间和不同类群的分歧时间。在这里有一个重要概念,分子钟:一个特定的生物大分子(蛋白质或DNA)在所有的演化谱系中具有恒定的演化速率。其中,演化谱系指不同类群。
目前进行分化时间分析的软件还是比较多的,比如beast2、paml软件包中的mcmctree、mega等软件。
今天呢,就给大家介绍一下如何利用beast2进行分化时间的分析。
beast2软件:
下载地址:http://www.beast2.org/
测试数据:软件提供的数据集Primates.nex
具体操作步骤:
1、软件安装(已经编译,下载即可运行)
2、相关软件包安装(解决其它生物学问题)
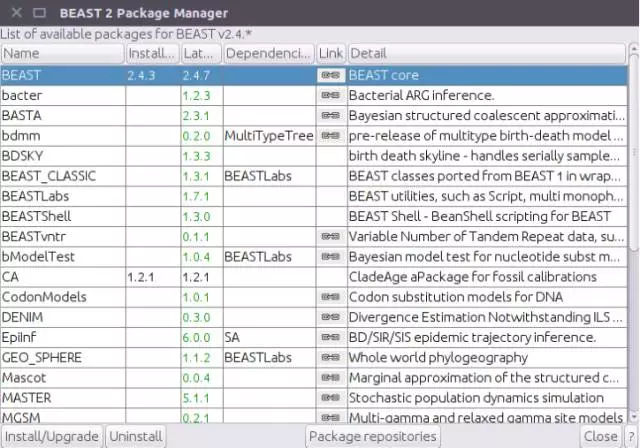
File—Manage Packages—跳出对话框
根据自己的需求去安装
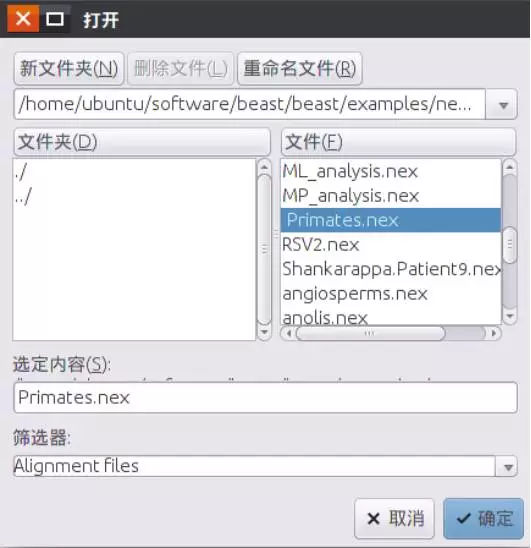
3、序列导入
File—Import Alignment—跳出对话框
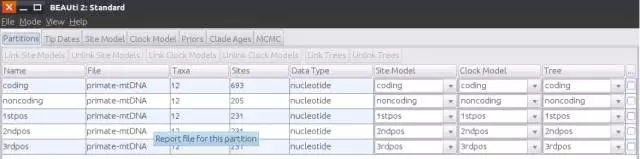
4、Link/Unlink partition models
一般在分析中,导入的是比对好的基因序列信息,但有些情况下,也会存在partitions情况,如果联合分析,就需要Link,如果不需要unlink。
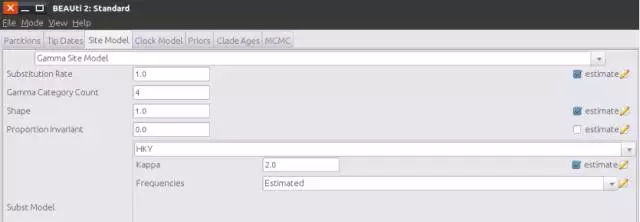
5、替代模型设定
针对核苷酸替代模型,可供选择的有GTR、HKY、JC69、TN93,这里用HKY替代模型,Gamma Category Count(碱基替代速率变化等级)一般可设置4-8,在这里选择4。其它参数如Substitution Rate(替代率)、Shape(GAMMA分布的形状参数)、Frequencies(碱基频率)在分析中均估计。
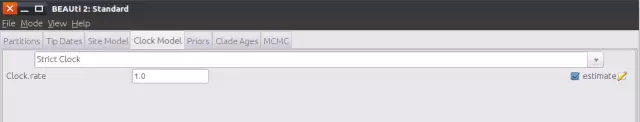
6、分子钟
点击Clock Model,有Strict Clock(严格分子钟)、宽松分子钟(Relaxed Clock Exponential 和Relaxed Clock Log Normal )等。在这里选择Strict Clock,不考虑模型中分支之间的速率变化差异。
7、先验信息设定
点击Priors tree prior:Yule Model 其它默认即可。
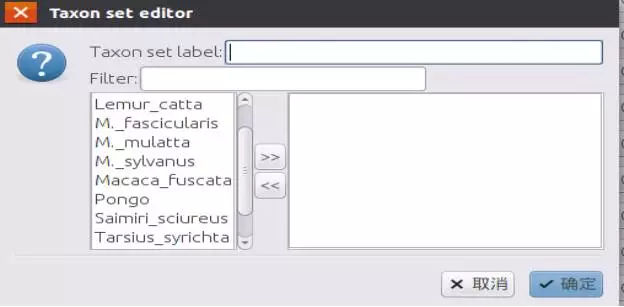
分化时间设定:
在Priors这一栏最下方有一个"+" 按钮,点击确定后,出现下图:
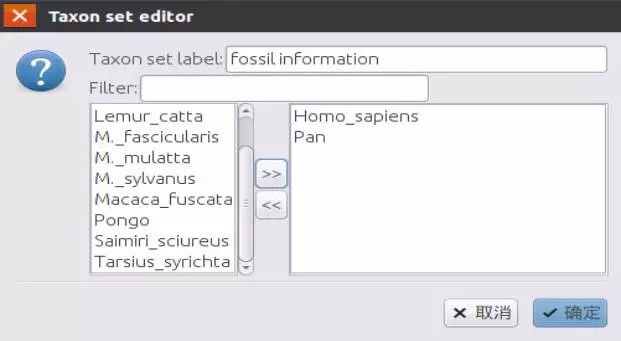
在Taxon set label自定义一个名字:比如fossil information。然后选择需要标定分歧时间的两个物种,移到右边边框。
现在需要在校正节点上设置先验分布信息,首先点击monophyletic在下拉菜单([none])中选择Normal
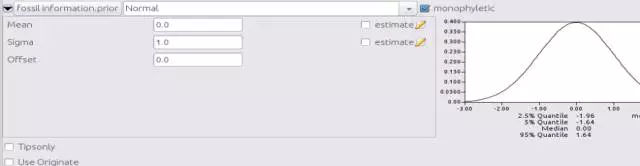
随后设置分歧时间,Homo sapiens 和 Pan的分歧在5-7My,因次设定正态分布中心点是6My,标准偏差0.5My。如图:

备注:可以设定多个分歧时间,重复该步骤即可。
8、MCMC选项设定
点击MCMC
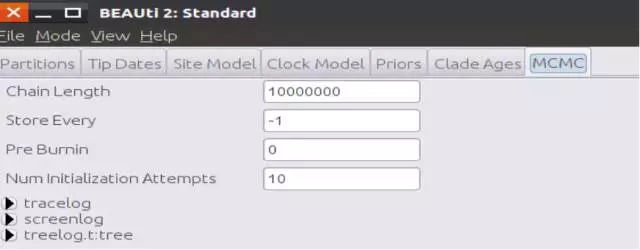
运行链长:10000000(默认1千万步,根据自己实际情况自行调整)
tracelog:链长运行1000次抽样一次
screenlog:运行1000次打印到屏幕一次
treelog.t:tree:运行1000次打印一个拓扑结构树
在第8步运行中,由于设置的链长是10000000次,所以在tracelog、treelog.t:tree等输出的结果文件中,会有10000000/1000=10000个抽样值等信息。具体输出结果多少依据自己的设定信息。
备注:根据设定的运行链长(假设1千万次)
抽样频率太小的话,输出无用信息会很多,且增加运行时间;
抽样频率太大的话,输出有用信息太少,影响后续分析。
在这里设定的是1000次,相对总的运行链长,比较合适。
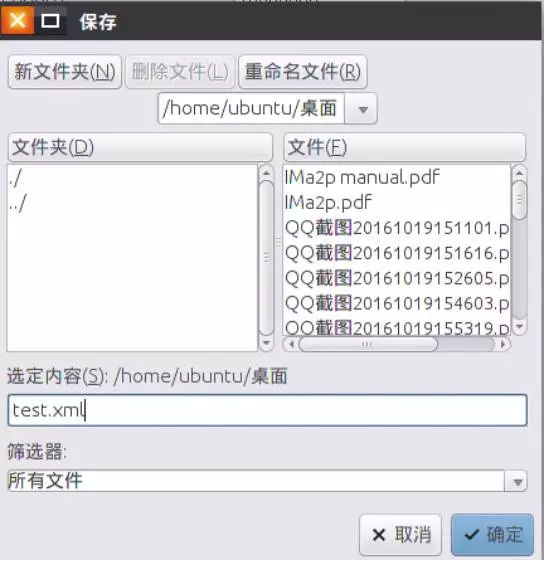
9、xml文件保存
点击File—Save As,定义一个输出文件名字,结果生成一个XML文件
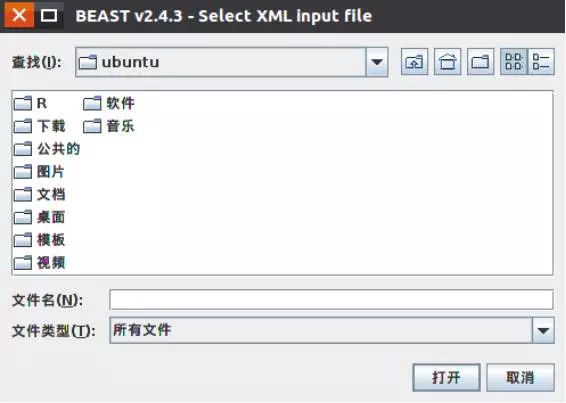
10、如果是linux系统,运行./beast,产生一个如下对话框,导入制作好的XML文件即可运行 。
命令行模式:nohup ./beast -threads 线程数 test.xml &
其它系统文件导入类似:
11、结果评估
结果主要生成3个文件:日志文件(*.logs)、树文件(*.trees)、xml状态文件(*.state)。
日志文件可用trace(软件)查看,评判结果是否收敛。
Trace软件下载地址:
http://tree.bio.ed.ac.uk/software/tracer/
主要查看:1、ESS ;2、Trace
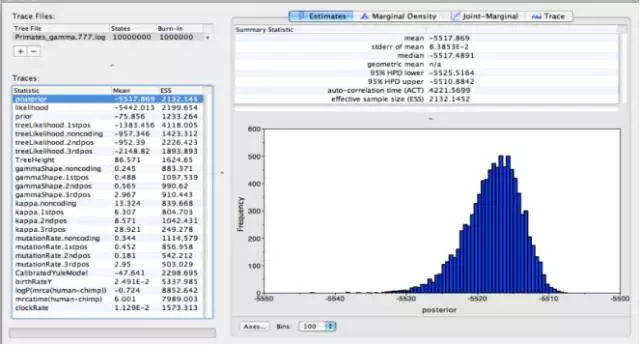
12、分化时间
利用beast软件包中的程序包treeannotator进行评估。
点击treeannotator,出现对话框:
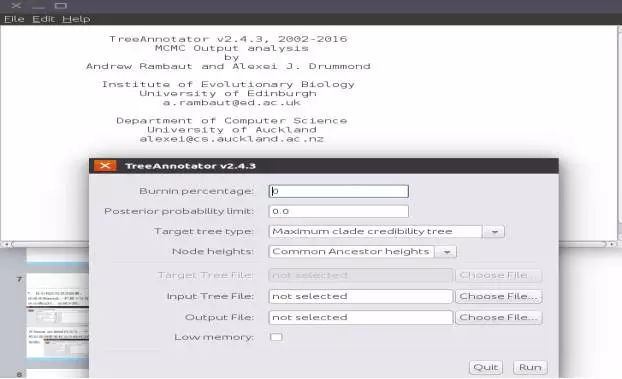
在这里设定Burnin percentage:50;Posterior probability limit:0.0;Target tree type:Maximum clade credibility tree ;Node heights:Mean heights
Input Tree File:主程序运行生成的树文件(*.trees)
Output File:输出文件名字
设置完成后,点击Run即可,最后会得到一个主要含有分化时间的树文件(NEXUS格式)。
13、查看
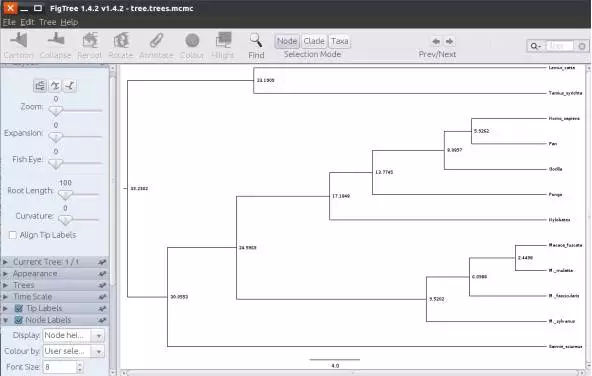
上一步生成的树文件可用软件figtree查看。
Figtree软件下载地址:
http://tree.bio.ed.ac.uk/software/figtree/
如图:前面设定Homo sapiens和pan分歧时间在5-7My,结果运行后时间是5.9262百万年。
- 本文固定链接: https://maimengkong.com/kyjc/1460.html
- 转载请注明: : 萌小白 2023年4月19日 于 卖萌控的博客 发表
- 百度已收录
