作者:易基因
总体来说,DNA甲基化一般遵循三个步骤进行数据挖掘。
首先,进行整体全基因组甲基化变化的分析,包括平均甲基化水平变化、甲基化水平分布变化、降维分析、聚类分析、相关性分析等。
其次,进行甲基化差异水平分析,筛选具体差异基因,包括DMC/DMR/DMG鉴定、DMC/DMR在基因组元件上的分布、DMC/DMR的TF结合分析、时序甲基化数据的分析策略、DMG的功能分析等。
最后,将甲基化组学&转录组学关联分析,包括Meta genes整体关联、DMG-DEG对应关联、网络关联等。
一、甲基化图谱分析
(1)平均甲基化水平的比较
平均甲基化水平能反应样本整体的甲基化水平。
但是平均水平差异不大并不能说明样本间甲基化图谱没有差异。
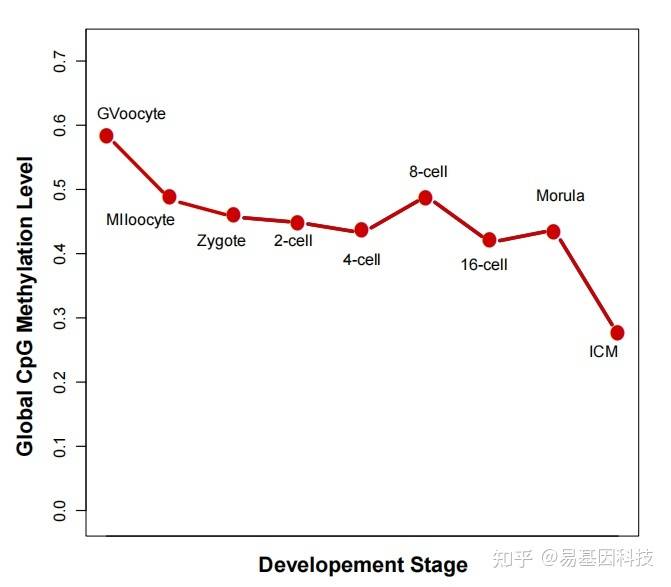
胚胎发育
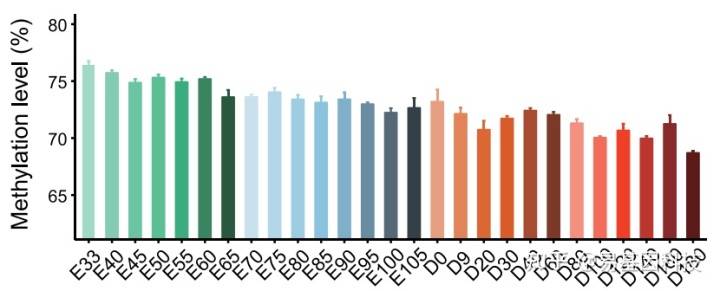
果实成熟
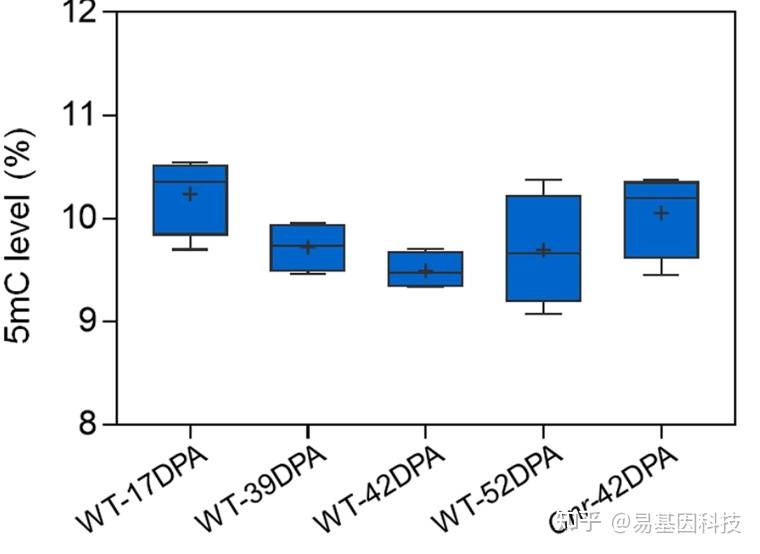
肌肉发育
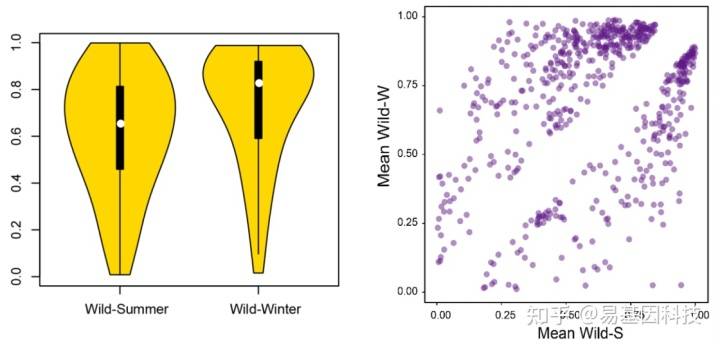
(2)CG/CHG/CHH甲基化水平分布
不同物种中,甲基化修饰可能倾向于发生在不同类型的C位点上,该分析有助于反应甲基化发生位点类型的偏好性。
甲基化水平分布的组间比较,能够更进一步了解组间甲基化水平的变化。
不同基因组元件(CGI相关元件、重复序列元件、基因元件等)的甲基化水平分布规律不同。特别是在不同物种中,基因元件的甲基化水平可能有一定的特点。
比较特定元件甲基化水平的组间差异也能发现潜在的功能差异。
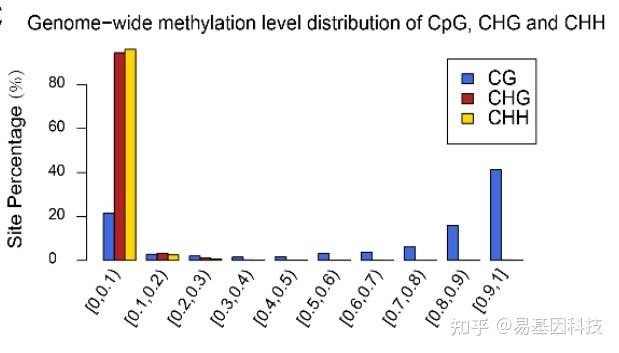
单样本三类甲基化水平分布
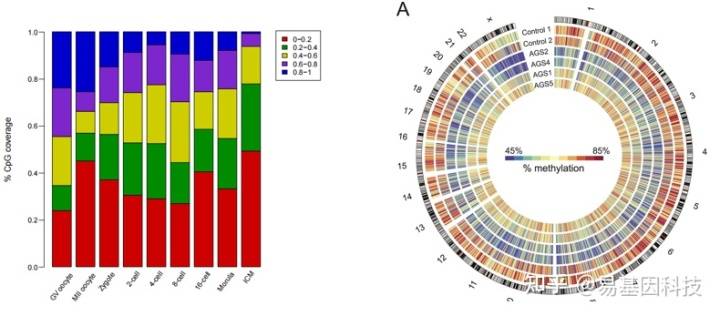
组间CpG甲基化水平分布比较
CGI相关元件
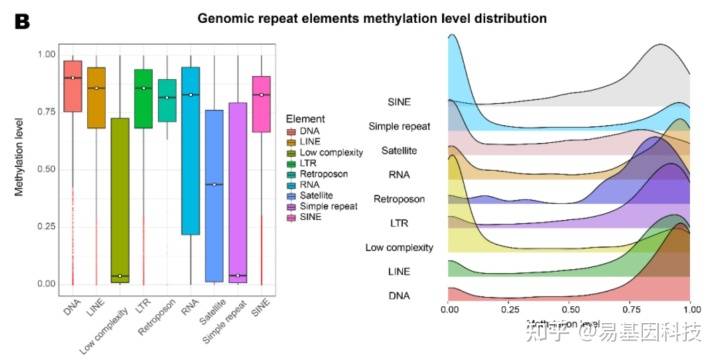
各类重复序列元件
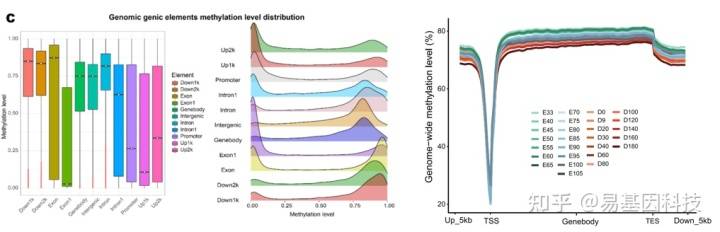
基因元件
(3)降维分析
降维分析尝试找到最能反映数据点真实分布情况的两个维度,以方便对数据进行直观把握。一般采用共同覆盖的5×以上位点进行分析:
主成分分析(PCA)
非度量多维标度法(NMDS)
主坐标分析(PCoA)
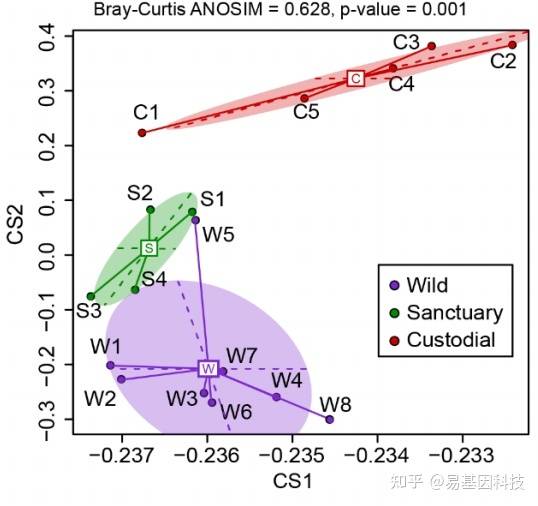
PCA
可采用统计检验分析组间差异的显著性:
相似性分析(ANOSIM)
置换多元方差分析(ADONIS)
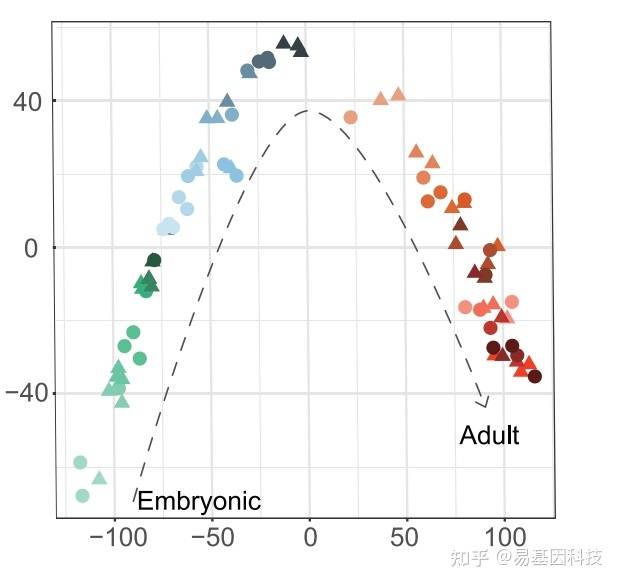
NMDS
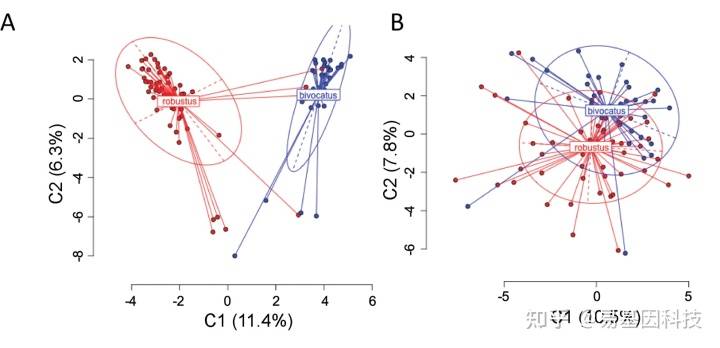
PCoA
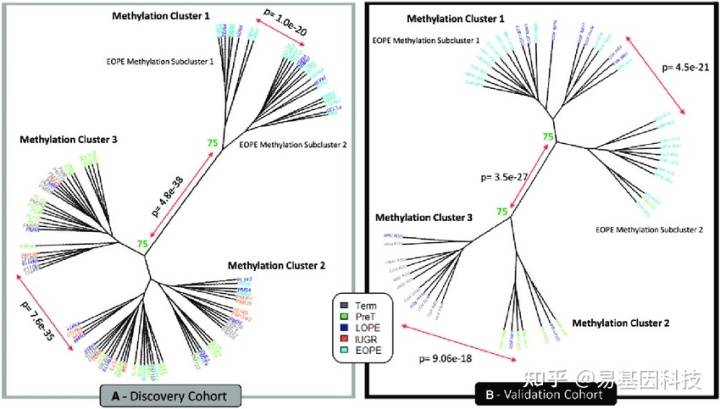
(4)聚类分析
聚类分析考虑的是各样本之间的距离,即不相似性。一般采用共同覆盖的5×以上位点进行分析。
与降维分析的差别在于,聚类分析更真实地反映样本的差距,而非仅考虑两个代表性维度。
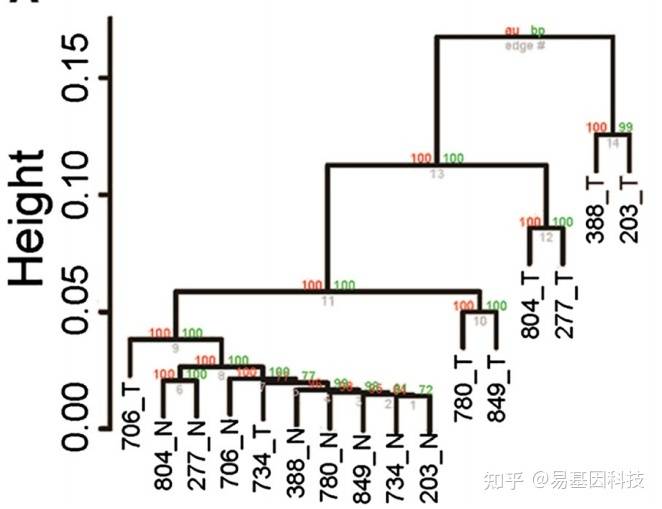
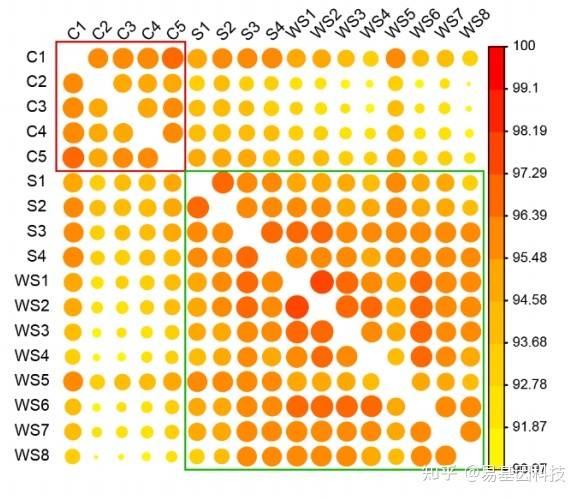
(5)相关性分析
相关性分析考虑的是各样本之间的相似性。一般采用共同覆盖的5×以上位点进行分析。
一般采用皮尔森相关系数
二、差异甲基化位点/区域分析DMC/DMR分析)
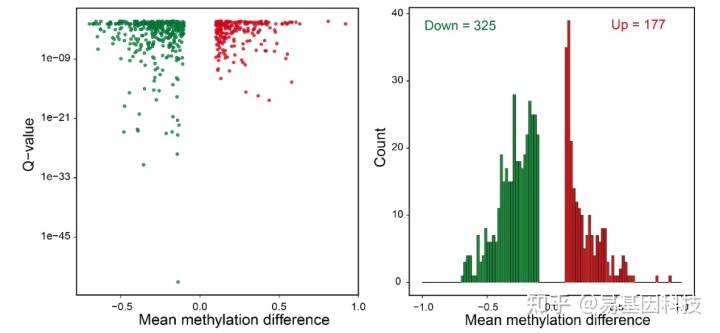
(1)DMC/DMR鉴定
差异甲基化位点:DMC
差异甲基化区域:DMR
(甲基化位点一般是与附近的位点一起起作用的)
鉴定实验组与对照组甲基化图谱的具体差异。
如果实验设计包括多个时间节点,也可以比较相邻时间节点/感兴趣的时间节点之间的甲基化图谱的差异。
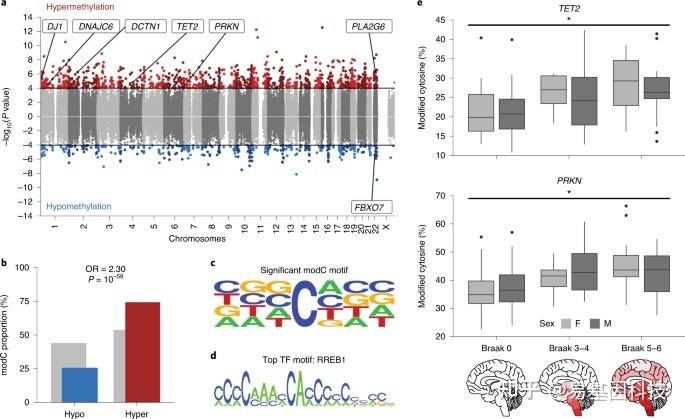
DMC在基因组上的分布
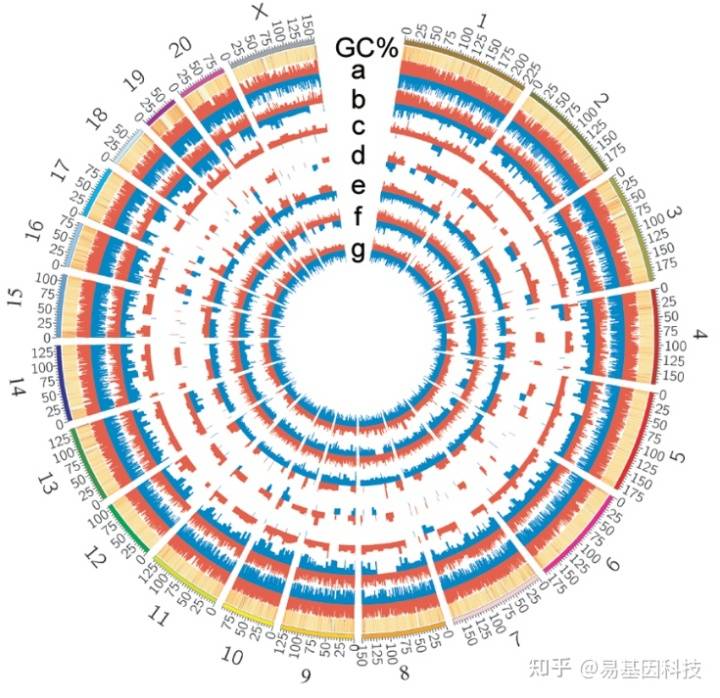
DMR在基因组上的分布
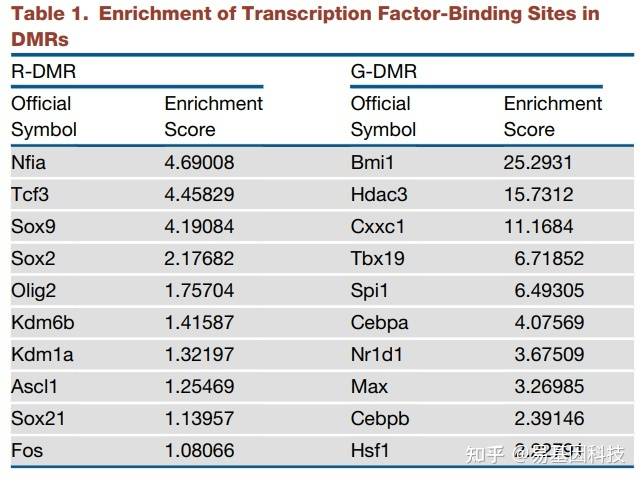
(2)DMC/DMR转录因子结合分析(TF binding motif )
主要关注Promoter和Enhancer等调控区域DMC/DMR的TF结合位点。
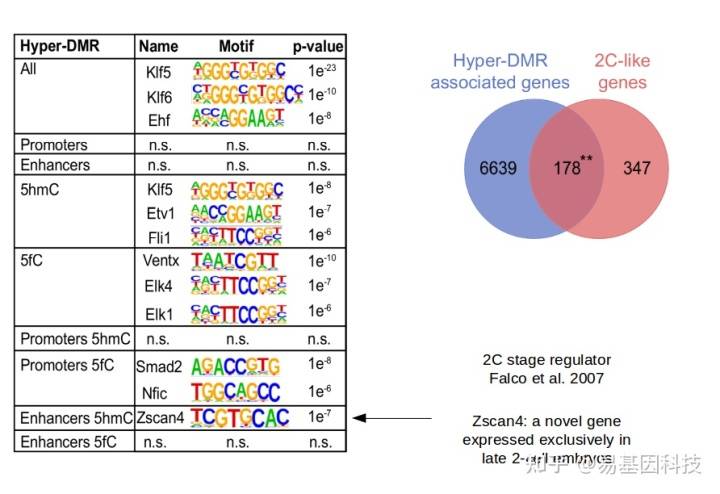
(3)时序甲基化数据的分析策略(Time Course)
比较相邻时间点的差异
直接筛选时间阶段相关的DMC和DMR
线性模型/混合线性模型
(可以排除混杂因素干扰,如性别)
共甲基化模式分析(阶段特异性Cluster筛选)
WGCNA(权重基因共表达网络分析)
MEGENA(多尺度嵌入式基因共表达网络分析)
mfuzz
... ...
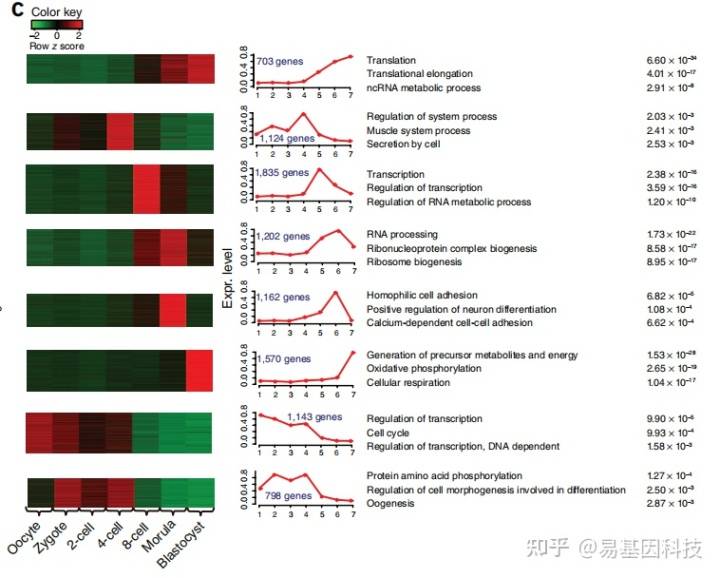
(4)DMC/DMR在基因元件上的分布
TE(转座元件):影响基因组稳定性
Promoter:影响基因表达
Genebody
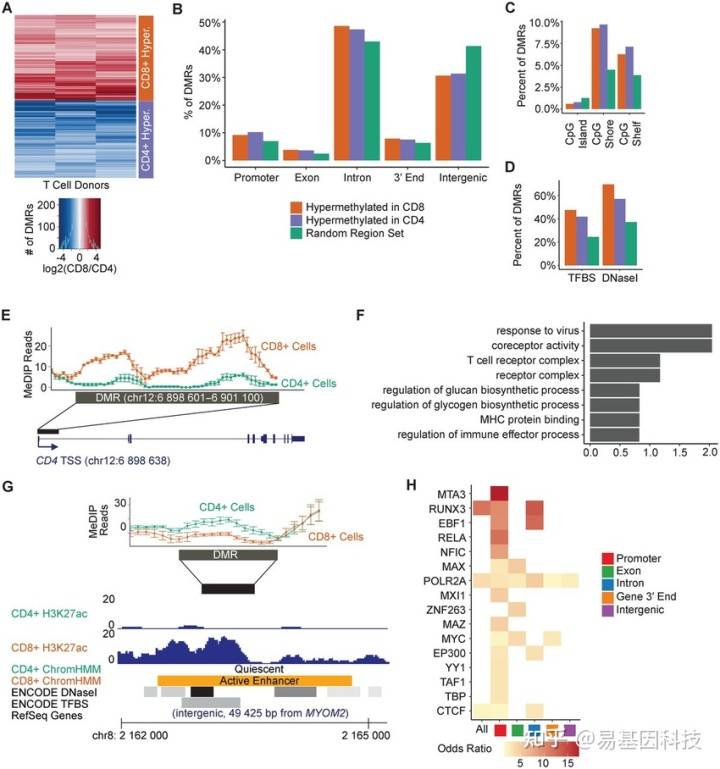
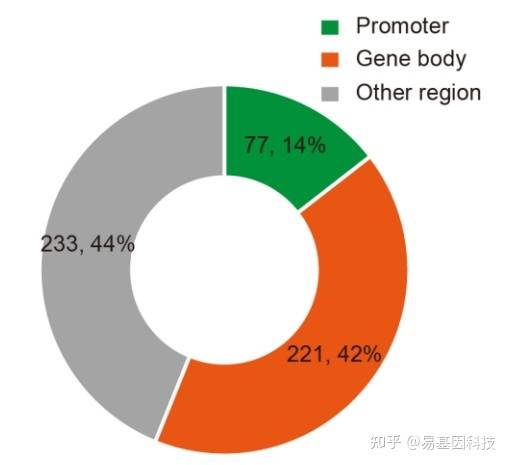
(5)差异甲基化基因集(DMGs)的功能分析
分析策略:
可以分为Hyper-DMG和Hypo-DMG
可以分为Promoter-DMG和Genebody-DMG
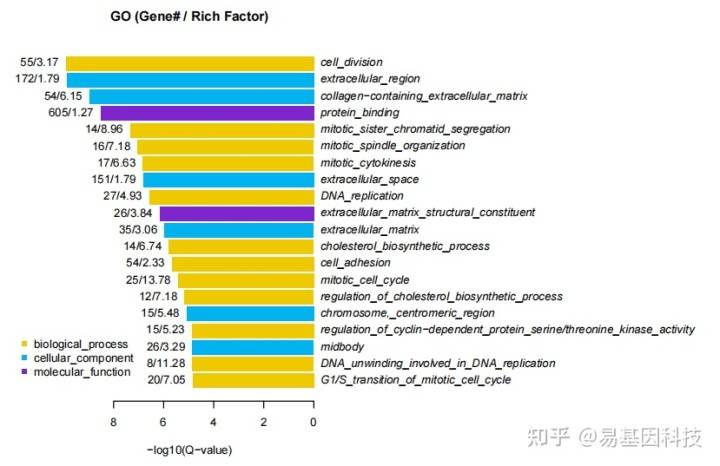
Gene Ontology
KEGG pathway
Reactome pathway
DisGeNET disease
Disease Ontology
三、组学关联分析:甲基化组学&转录组学
(1)Meta genes整体关联
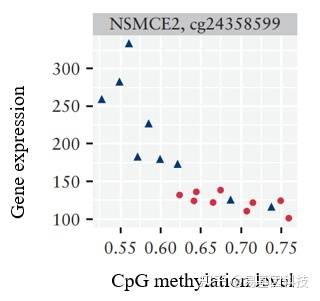
同一样本/组别内,所有基因的表达水平与对应基因的甲基化水平进行关联。
研究的是基因甲基化与表达的整体关系。
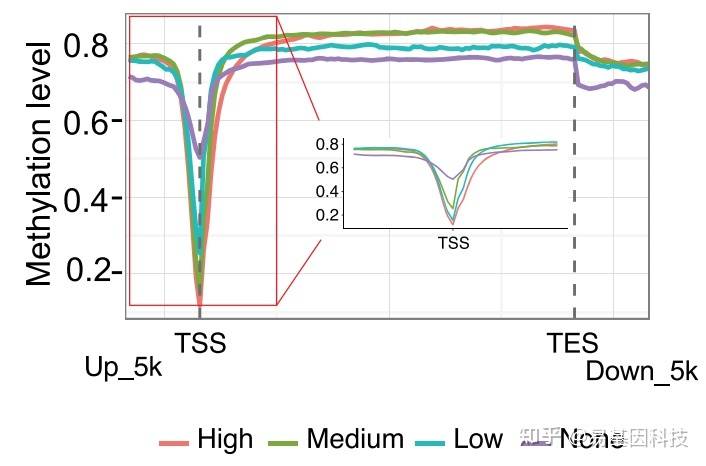
TSS位点附近负相关 Genebody区正相关
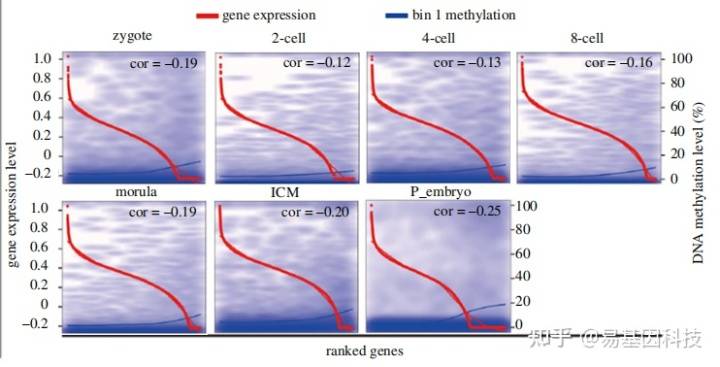
整体负相关
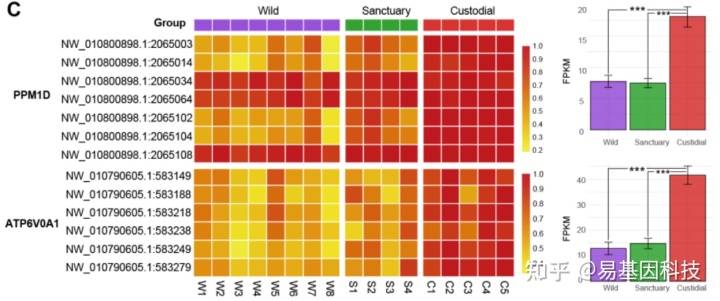
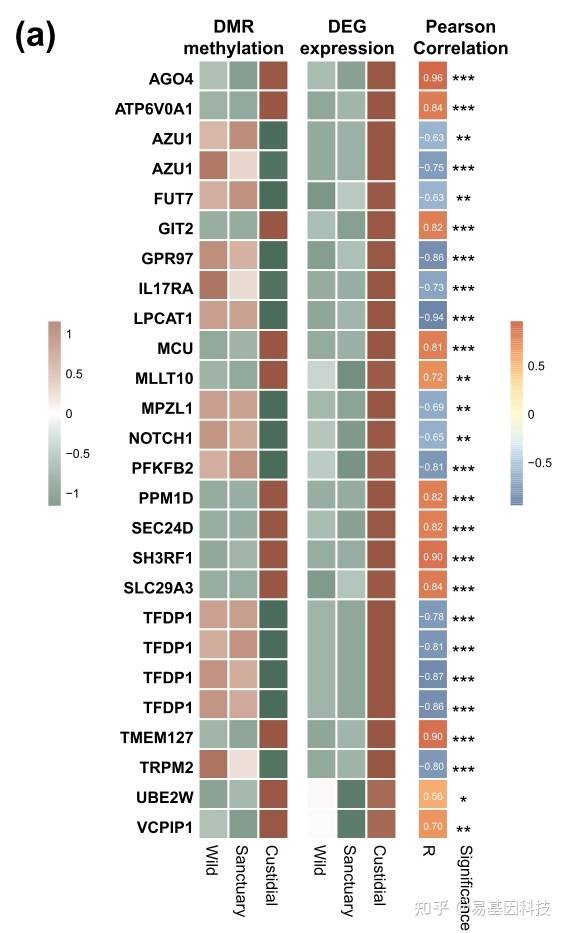
(2)DMG-DEG对应关联
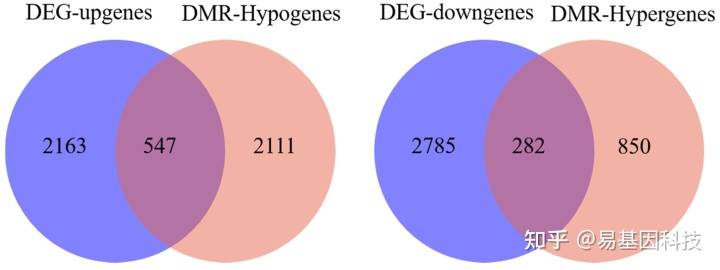
重叠分析: 特点:简单粗暴,也适用于样本量少的情况。 分析结果:韦恩图。
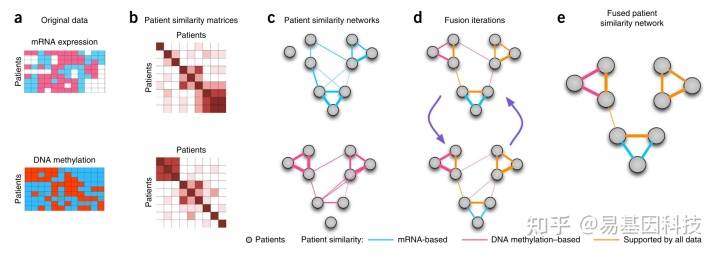
(3)网络关联基于基因表达具有功能和通路的富集性。有最低样本数量要求。
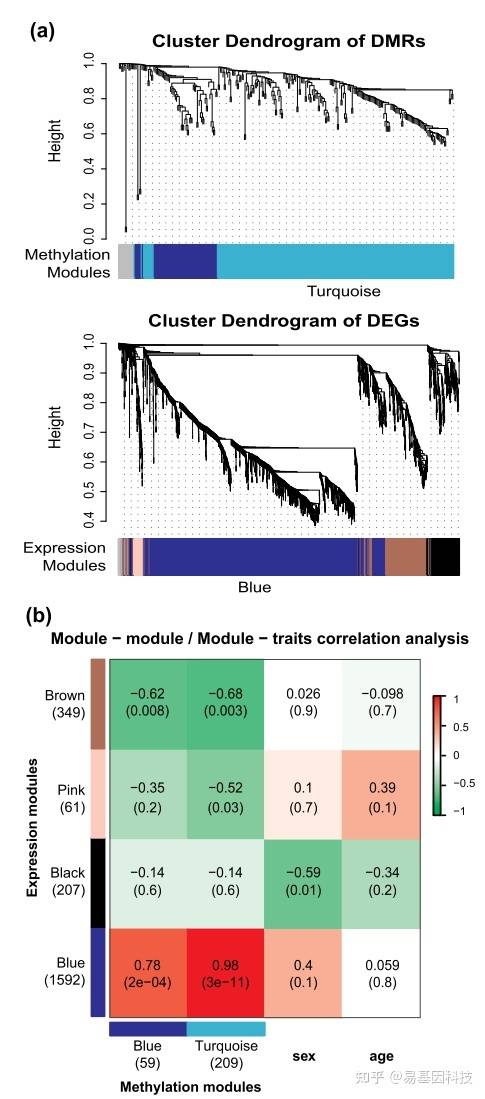
共表达-共甲基化网络关联:
WGCNA module correlation
EMDN algorithm
融合网络关联:
SNF algorithm
以上就是关于DNA甲基化测序的数据挖掘思路分享。
- 本文固定链接: https://maimengkong.com/kyjc/1371.html
- 转载请注明: : 萌小白 2023年1月29日 于 卖萌控的博客 发表
- 百度已收录
