本文转自组学大讲堂,侵删
KEGG数据库应该是我们最常见的、使用频率最高的数据库之一了,它是一个整合了基因组、化学和系统功能信息的数据库。由日本京都大学生物信息学中心的Kanehisa实验室于1995年建立。是国际最常用的生物信息数据库之一。

在KEGG分析结果中,总还是有人分不清例如K01000和ko01000这样的编号的生物学意义,下面我给大家再介绍一遍:K01000:由大写K+五位数字组成,对应的是KEGG数据库中某类蛋白的编号,即某些基因注释到了该蛋白,该编号代表着这一类功能的蛋白,一个基因对应一个这种大K编号;ko01000:由小写的ko+五位数字组成,是pathway的编号,对应的是某一条代谢通路;一条通路里可以有多个基因共同参与调控,所以会存在多个大K编号,同样,一个基因也可以参与多个代谢通路所以一个基因也可以有多个ko注释,这下应该明白了吧!
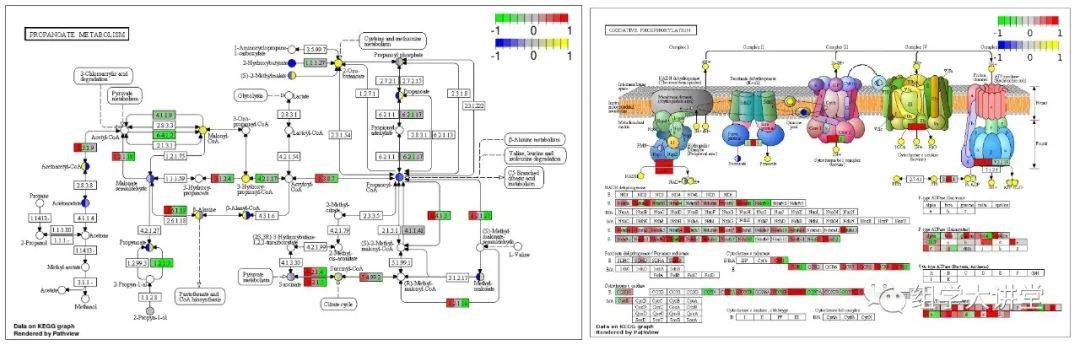
KEGG注释很是常见,几乎涉及到功能注释的项目都会有KEGG注释及富集分析,那么当我们找到某些感兴趣的基因或通路后,我们能不能仅用我们感兴趣的基因绘制漂亮的KEGG代谢通路图呢?就像下面这样的!当然,是我们自己做,而不是求助公司!答案是肯定的,今天小编就手把手教你自主绘制KEGG代谢通路图!
Pathview网页版简介:
Pathview是一个通路可视化友好的R包,支持多组学数据映射(基因/蛋白-代谢),更重要的是 Pathview还有一个网页版,这对于非生信人员真是重大好消息!
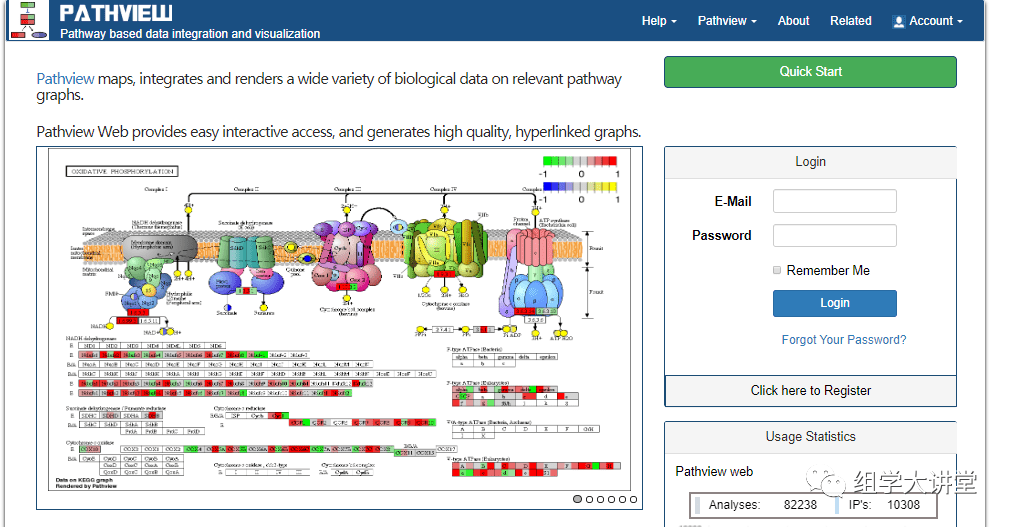
Pathview网页版具有如下优点:
1. 图形界面很是友好,访问速度快。
2. 有完整的通路分析流程,支持多组学数据整合分析,且数据库是最新的通路数据。
3. 结果是交互式的,可以更深入了解数据。
4. 可以游客身份登录也可免费注册,注册用户可以共享数据和保存分析历史。
今天我们仅介绍Pathview绘制单一数据kegg通路图,下面进入正题:
数据准备:
我们以6个人的转录组数据为例,我感兴趣的是某一分组中与MAPK signaling pathway有关的基因,于是我从该组差异基因列表中筛选出目标基因及FPKM值整理如下表。
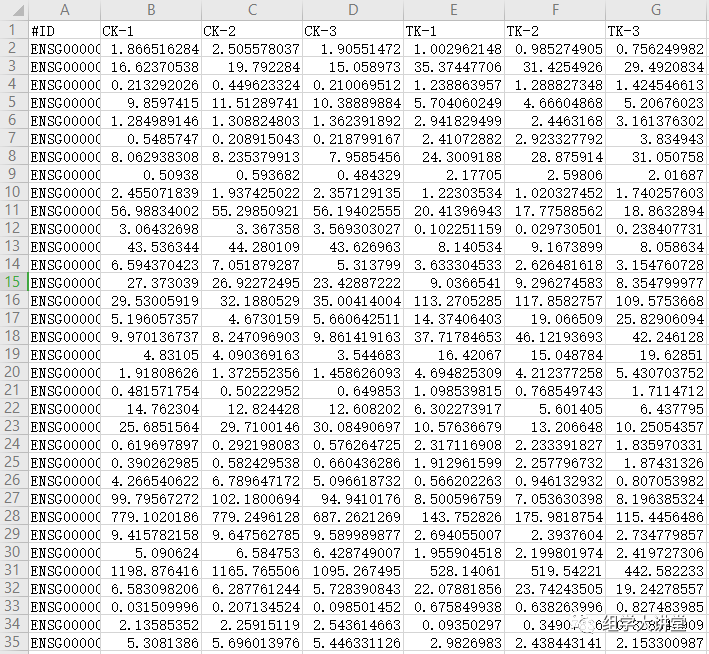
Pathview数据文件格式都是以tab或者逗号分隔的txt或者csv文件,EXCEL数据可以通过另存为(制表符分隔)TXT文件格式,如下:

数据提交与选项设置:
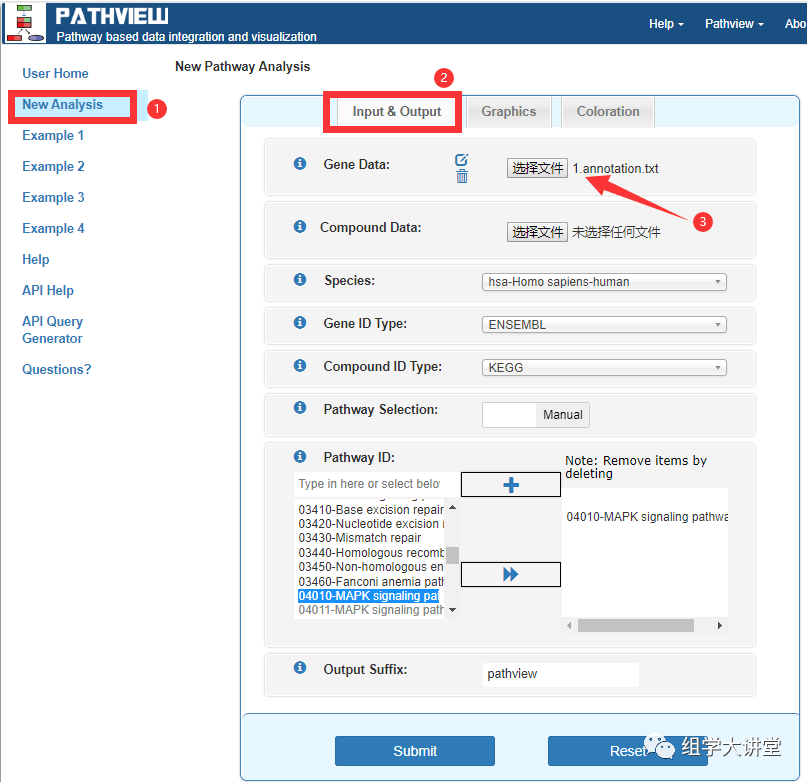
如下图:点击New Analysis进入分析页面,首先在input&output下提交数据,Gene Data项选择文件上传数据。
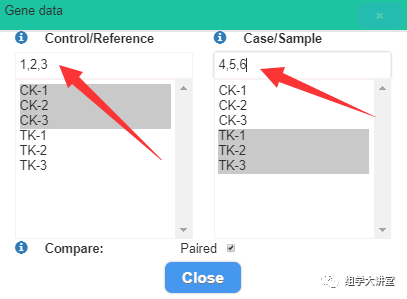
数据上传完页面会自动跳出样本分组对话框,如下图:箭头处填写样本顺序号,并用逗号隔开即可分组,完成后点击Close关闭对话框。
接下来就是选择参考基因及数据类型等,参考基因选择人类参考基因组,转录组分析时参考基因组来源于ensembl,所以基因ID类型选择ENSEMBL,其他数据按实际情况自行选择即可。
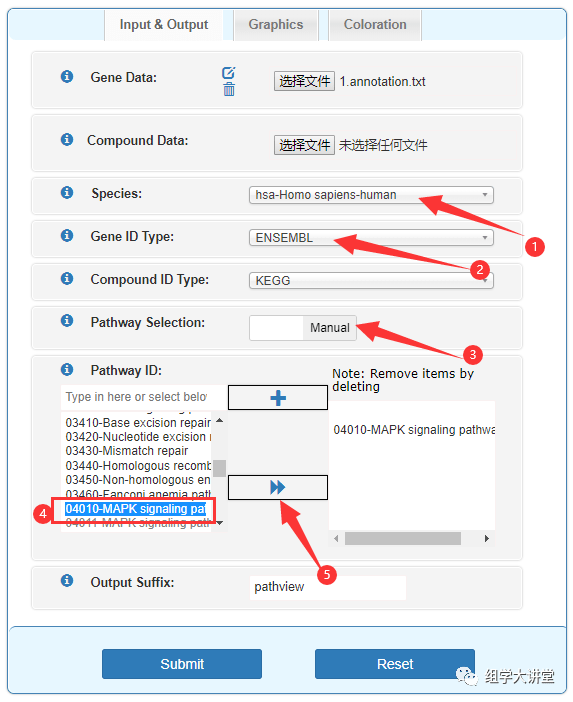
需要注意的是步骤③,Manual选项下,可以手动选择感兴趣的通路,此处也可以选择Auto,数据库会自动给出能注释到的所有通路结果。步骤④在左侧选择感兴趣的通路,选中,点击⑤即可添加到右侧。
选项Graphics,即图形参数,无特殊需求可默认;选项Coloration是颜色设置,可按需调整。
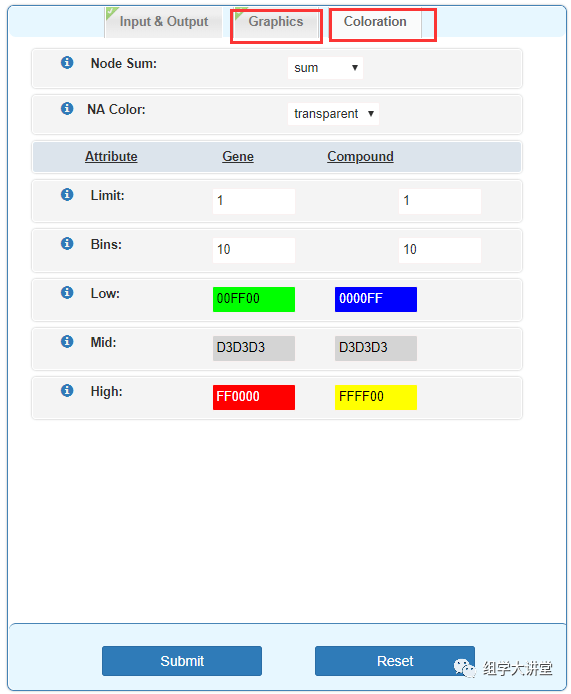
以上操作完成点击Submit提交任务。
结果展示:
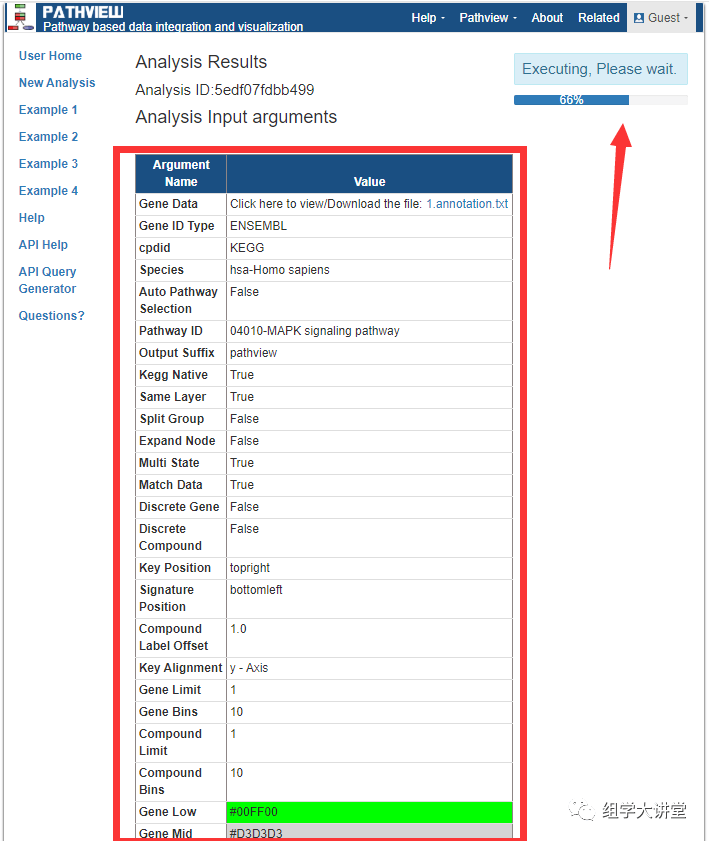
Pathview绘图速度很快,如下图,绘制完成后,进度条位置便会出现结果查看链接。
下图是生成的MAPK signaling pathway注释结果,点击链接即可查看绘图:

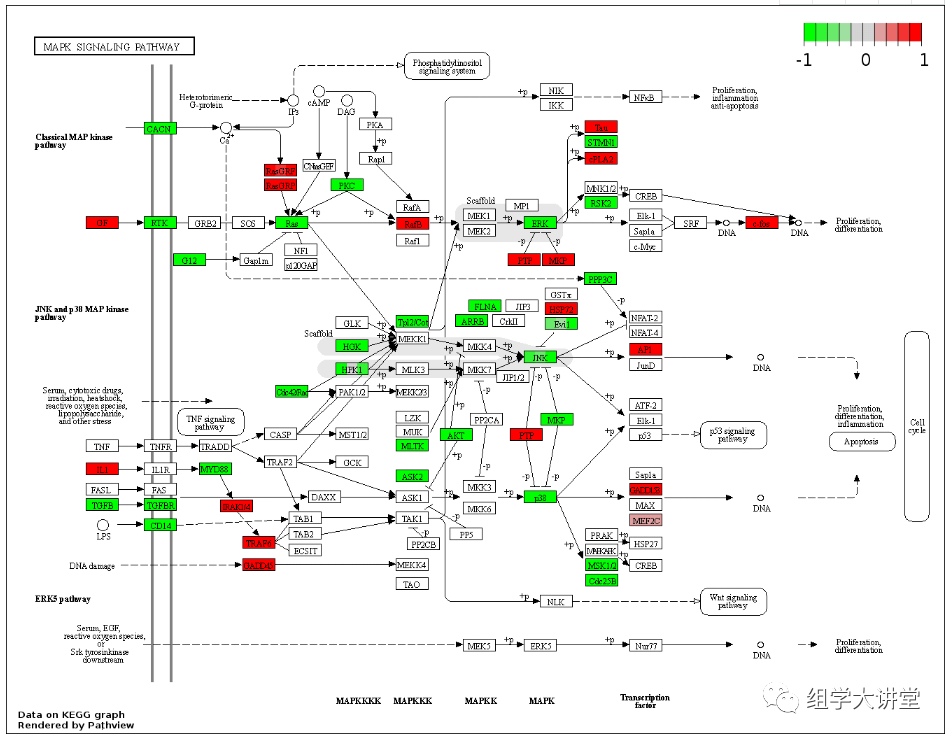
绘制的KEGG注释图是可交互式的,每个带颜色的term 都带有超链接,可点击它们转到更详细的解释,如下图:

下载绘图结果点击下方位置所示,下载结果中有PNG格式图片及keggID与基因ID对应文件。
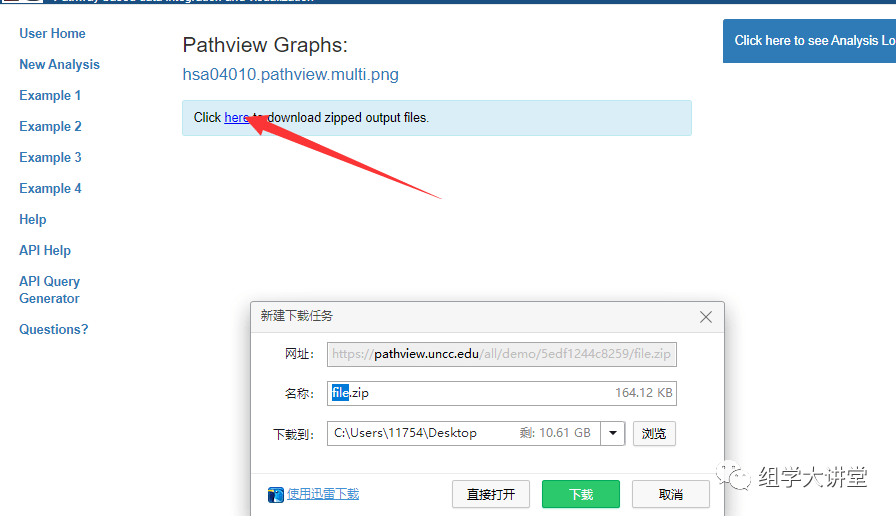
这样,重新绘制的部分基因kegg注释图就做完了,是不是很简单?Pathview支持上百种有参物种,适用性很广,感兴趣的话赶紧试一下吧!
Pathview网站地址:https://pathview.uncc.edu/- 本文固定链接: https://maimengkong.com/image/1064.html
- 转载请注明: : 萌小白 2022年6月29日 于 卖萌控的博客 发表
- 百度已收录
