2019年2月26日,中国科学院植物研究所北方资源植物重点实验室与北京百迈客生物科技有限公司等单位合作,成果以“A Chromosome-Scale Genome Assembly of Paper Mulberry (Broussonetia papyrifera) Reveals the Genetic Basis of Its Forage and Papermaking Usage”为题发表在国际著名植物学期刊Molecular Plant上。中国科学院植物研究所彭献军副研究员和刘辉博士后为本文并列第一作者,沈世华研究员和百迈客于海燕为论文的共同通讯作者,百迈客的刘敏、李绪明、戴鹤、王丽丽等人为共同作者。该研究应用最新测序技术,结合光学图谱,HIC测序和高密度遗传图谱,成功构建了高质量的构树全基因组谱图,达到了准染色体水平的组装,为进一步改良构树的农艺性状奠定了基础。此外,利用构树的全基因组序列以及转录组数据,对构树的进化历史、基因组功能注释和物种间的基因组共线性等进行解析;还对构树的纤维发育、木质素和黄酮类代谢、氮代谢以及金属耐受性和抗逆性等相关重要基因展开了深入研究。


摘要
构树因被用于古老中国伟大的四大发明之一——蔡伦造纸而世界闻名。目前,该植物被用作饲料以解决国家饲料短缺问题。为了加速构树的驯化、利用和推广,该研究使用多组学数据,包括Illumina和单分子实时测序数据,以及Hi-C,光学图谱和遗传图谱进行遗传背景分析和微生物鉴定。构树的基因组被组装成大小为386.83Mb,99.25%的序列被定向到13条染色体上。比较基因组分析显示:木质素合成基因家族收缩明显,降低了木质素的合成以及木质素单体比例S/G(syringyl lignin / guaiacyl ligin)值的提高,使得构树适用于医药、造纸和树皮衣;黄酮合成途径基因家族显著扩张,增加了黄酮的含量。宏基因组数据显示,构树共生菌主要为假单胞杆菌和根瘤菌,表明其作为饲料的氮的来源。这些结果说明,构树经历了适应性进化,黄酮合成的增强及木质素单体比例的调节有利于募集共生微生物,以促进构树的生长。本研究揭示了构树用于造纸、树皮衣和饲料养殖的遗传基础,将促进构树的驯化和利用。

背景研究
构树(Broussonetia papyrifera,2n=2x=26)属于桑科(Moraceae)构属(Broussonetia)多年生乔木,是我国乡土树种和先锋植物,有悠久的历史和文化,因为蔡伦用它造纸而世界闻名。构树的树皮和树干是造纸的优质原料,树叶还可以作为蛋白饲料,其根、茎、叶、果实及种子均可入药,富含黄酮类化合物;还是尾矿处理、生态绿化的理想树种。构树具有分布广、适应性强、生长迅速、种子水量极大、基因组紧凑、表型性状丰富,因此,可作为木本植物研究的模式材料。
当前,粗蛋白饲料的短缺已经成为畜牧业的瓶颈,生态恢复也成为了世界难题。因此,通过本研究团队构建了新的杂交构树用于饲料以补充目前青贮饲料的不足。然而,有关构树的研究主要集中于造纸、药理药化、养殖以及生态绿化等应用方面,基础生物学的研究很少。因此,构树栽培改良的第一步是获得其遗传背景,以便能更好地掌握其特有特征的生物学机制。
材料和方法
材料:5年的雌性构树
基因组denovo策略:
二代小片段文库(insert size:220bp和500bp)和mate-pair文库(insert size:3kb,4kb,5kb,8kb,10kb,15kb和17kb),测序仪器:Illumina HiSeq 2500,测序深度:311.73×;BioNano,数据量:118.42Gb,测序深度:~310×;三代20Kb文库,测序仪器:PacBio Sequel,数据量:8.54Gb,测序深度 :22.47×;遗传图谱:用与基因组测序的雌性亲本和未知雄性亲本杂交获得的CP群体(包含120个F1子代),通过SLAF-seq的方法构建遗传图谱,测序平台:Illumina HiSeq 2500,数据量:180.27M reads;Hi-C,数据量:15.28Gb,测序深度:38.56×;
研究方法:
基因组组装注释:1、基因组大小评估:a,基因组调研图:Genome Size = 17-mer number / 17 – mer peak depth,预估基因组大小 ~ 380.73Mb;b,流式细胞仪:预估构树基因组大小为386.48 ±4.47Mb。2、基因组组装:a,二代数据组装:ALLPATHS-LG,SSPACE,GapCloser;组装基因组(version 0.1)373,384,200bp,scaffold N50 = 1,034,263bp;b,光学图谱组装:BioNano Genomics ,RefAligner;组装基因组(version 0.2)384.85Mb;c,三代数据辅助组装:LoRDEC,Pbjelly;组装基因组(version 0.3)396.86Mb,contig N50 = 173.68kb;d,遗传图谱辅助组装:MAPS,图谱构建软件,SAMtools,GATK,ALLMAPS,由286.63Mb的scaffold锚定在染色体上,占基因组的72.2%,有128个scaffolds(268Mb)得到了定向;e,Hi-C辅助基因组组装:Hi-C-Pro,LACHESIS,最终组装出的基因组大小为386.83Mb,99.25%的序列被定向挂载到13条染色体上,Contig N5为171.17kb,Contig N90为38.90kb,Scaffold N50为29.48Mb。3、基因组评估:a,Hi-C互作热图评估;b,转录组完整性预测(构树叶片、茎干、根和花组织),Tophat2,Cufflinks,96.56%的reads回比到组装的基因组上;c,BUSCO评估,BUSCO(vertion 2.0.1)(embryophyta_odb9)。4、基因组注释:a,重复序列预测,采用同源预测和从头预测的策略对构树基因组进行重复序列预测,软件包括RepeatMasker (version open-4.0.5),PILER (version 1.0),RepeatScout (version 1.0.5),LTR-finder,MITE,PASTEClassifer;b,蛋白编码基因预测,利用从头预测(PASA,AUGUSTUS(vertion 3.0.3),SNAP,GlimmerHMM,GeneID,Genescan (version 1.1.0),)、同源预测(川桑,Genewise (version 2.2.0)和转录组预测(TopHat2 (version 2.0.7),Cufflinks (version 2.2.1),GeneMarkS-T (version 5.1))对构树蛋白编码基因进行预测;c,同源序列鉴定,GenBlastA (version 1.0.4);c,基因功能注释,InterProScan (version 5),Hmmscan (HMMER, version 3.0),BLAST2GO (version 2.5),BLASTP,Trembl;e,非编码RNA预测,tRNAscan-SE (version 1.3.1),Infernal cmscan (version 1.1.1)。
比较基因组分析:1、直系同源基因鉴定:利用软件OrthoMCL (version 2.0)鉴定直系同源基因,并利用BLASTP (Blast+ version 2.3.0)计算成对序列相似性;2、系统发育树构建与分化时间估算,利用14个物种(无油樟、亚麻、毛杨、棉花、拟南芥、黄瓜、苜蓿、桑树、构树、桃树、葡萄、番茄、毛竹和玉米)的单拷贝直系同源基因构建系统发育树,软件为MUSCLE、Gblocks (version 0.91b)和RaxML(version 8),MCMCTREE评估分化时间;3、基因家族扩张和收缩分析,CAFE(vertion 3.1);4、染色体共线性分析、4DTV检测及Ks值计算,MCscan。
共生微生物多样性分析:两周内收集中国10个地区的构树和根际土壤样品进行宏基因组测序,分析软件FLASH(v1.2.7)、UCHIME(v4.2)、UCLUST、QIIME(vertion 1.8.0)。
结果与讨论
1、基因组测序、组装和注释
本研究使用Illumina HiSeq和PacBio Sequel测序平台,用Hi-C、光学(BioNano Irys)和遗传图谱辅助,进行雌性构树的基因组测序,构树的测序和组装流程见图1。
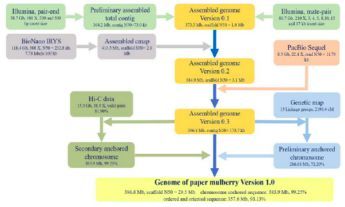
图1、构树的测序和组装流程图
组装出构树的基因组大小为386.93Mb,scaffold N50是29.48Mb(vertion 1.0);96.71%的基因组由contig序列组成,其结果与流式细胞仪和基因组调研图的评估相近。有99.25%(357.56Mb)的基因组被锚定在13条染色体上。大多数(98.03%)的核心真核基因(CEGs)和BUSCO数据库的基因(84.17%)得到了鉴定。另外,由转录组数据组装获得的93.73%的unigenes能够比对到基因组上。而且Hi-C热图也显示,所有的bins能够明确地分到13条染色体上,见图2。综上,证实该研究获得了精确且完整的,并达到染色体水平的高质量构树基因组。
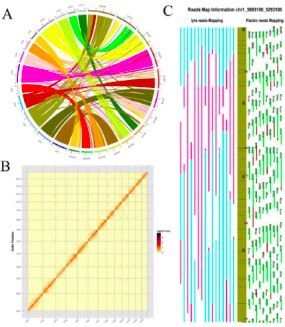
图2、使用不同策略产生的组装结果的相互校正
注:A,圈图显示Hi-C数据和遗传图谱之间共线性结果;B,通过计算热相互作用相关性验证Hi-C辅助染色体组装;C,通过比对BioNano IRYS分子和PacBio Sequel测序数据验证最终组装的染色体。
一共预测了30,512个基因,98.09%与已知基因同源并且得到了功能上的注释。共有27,778(91.04%)个基因锚定在13条染色体上,有27,134个基因显示在不同组织中差异表达,见图3。整合转录组数据后共预测到2,979个非编码RNA,几乎是川桑中的三倍。另外,构树基因组中有324个NBS抗性(R)基因,占构树基因的1.06%,该比例超过桑树(0.48%)、拟南芥(0.73%)、毛杨(0.88%)和番茄(0.77%),但低于咖啡(2.19%)、葡萄(1.76%)和玉米(1.51%)。来自多个类别的很多R基因成簇集中在1,4和13号染色体上。R基因的扩张表明存在强大的免疫系统,这是构树对病原菌或微生物相关分子模式的广泛适应性和抗性的原因,也可能是构树在引入到其他大陆时能够侵入性繁殖的主要原因。
一共鉴定到190.23Mb(49.18%)重复序列,与桑树(47%)和苹果(42%)相似,高于白杨(35%),低于高粱(~62%),超过水稻和拟南芥(14%)的三倍。与其他植物相似,构树中主要的长末端重复序列(LTRs)是Gypsy和Copia元件。然而,在构树中DIRS(5.61%)和LARD(8.68%)元件的比例高于其他植物。另外,共计2,327个简单重复序列(SSRs)均匀地分布在染色体上,这有利于分子育种。

图3、构树的染色体
注:III中RB(黄色,主根),SB(红色,木质化程度低的茎),SD(绿色,木质化程度高的茎),LA(蓝色,叶芽),LC(紫色,成熟叶片)
2、构树的基因组进化
系统发育树显示构树与桑树在同一分支,在大约3100万年前与桑树分开,与桃子的分化时间在大约7800万年前,见图4。
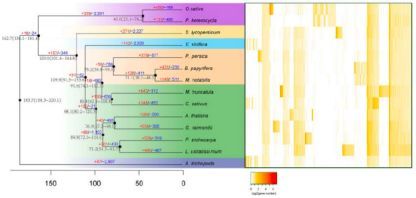
图4、基因组进化及构树与其他13个植物基因组的比较
注:左图,14个物种的系统进化树,右图,基因家族聚类热图
该结果与其他报道是一致的,也被4DTv的分析结果所证实,见图5。构树的4DTv峰值是1.9(见图5A),说明在构树的进化过程中仅发生了祖先γ全基因组复制(WGD)事件,该结果通过Ks分析也得到了进一步的证实,见图5B,C。以上结果表明,构树没有经历近代的α或βWGD事件。
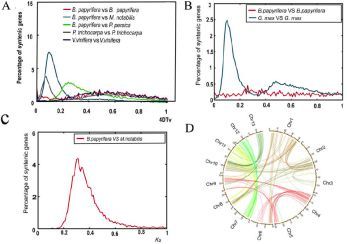
图5,基因组复制历史分析
注:A,构树基因组的4DTv分布;B和C,构树的Ks分析;D,构树基因组的三重同源基因分布。
在构树基因组中作者鉴定到7个主要三倍化区域对应于302个旁系同源关系,说明构树与其他双子叶植物,如:葡萄、苜蓿、桃子和桑树,共享古六倍化事件。此外,经自身比对鉴定到19,763个旁系同源基因,占构树基因组的56%,与葡萄、苜蓿和桃子相似,但是少于白杨和拟南芥,说明近代的βWGD事件发生在拟南芥和白杨中,但是在构树中没有发生。
根据已报道的双子叶植物祖先和谱系特异性WGD,本研究推测,古六倍化始祖的21条染色体至少经历了11次大的染色体融和(cfus)和2次染色体裂变后产生了桑科中间状态的12条始祖染色体,见图6。桑科的始祖染色体的数目与葫芦科和杨柳科是相似的,但是与蔷薇科(n = 9)、豆科(n = 6)、锦葵科(n = 16)和茄科(n = 16)是不同的。进化推演分析表明,构树的染色体是从桑科的12条始祖染色体经27次融合和28次裂变重构的,说明构树基因组在进化过程中至少经历了68次的染色体融合和裂变。
有趣的是,桑科、蝶形花科、蔷薇科和豆科的中间始祖染色体的数目是3的倍数,而茄科和锦葵科却不是,见图6。因此,本研究假设另一个共享染色体重构事件发生在豆科和古六倍化祖先之间或者所以豆科植物的祖先染色体数量是12。
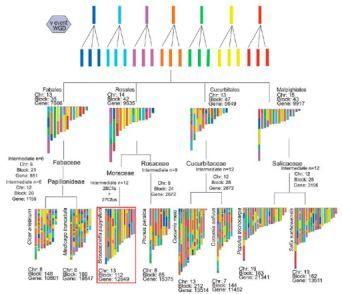
图6、构树和其他6种植物基因组重构的进化推演
进一步分析构树和其他植物染色体水平的同源基因对,显示构树与桃树有最强的共线性关系(已测序物种中,桑树与构树进化关系最近,但桑树没有组装到染色体水平,这个分析中没有用桑树),包含191个主要的共线性区域,分别占构树和桃树基因组的51.44%和26.12%,见图7。
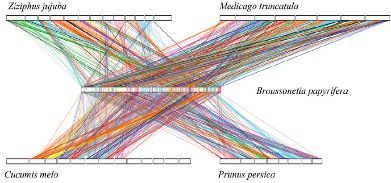
图7、种间共线性分析
3、比较基因组分析
在构树基因组中共发现15,254个基因家族,与桑树分化之后,有431个基因家族扩张,230个基因家族收缩,见图4,表明在适应进化过程中,构树中更多的基因家族经历了扩张而不是收缩。
扩张基因家族中有550个基因在KEGG中得到了注释,且大多扩张基因注释在植物-病原体互作、氰氨基酸代谢、黄酮类和三萜烯的合成中;有1432个基因在GO中得到注释,主要的代谢过程包括:五环三萜类生物合成、防御反应和黄酮类生物合成。还发现109个收缩基因聚集在30个KEGG通路中,包括苯丙烷类生物合成、淀粉和蔗糖代谢。收缩基因的GO terms主要与氧化还原过程、蛋白磷酸化和木质素分解代谢过程。扩张和收缩基因的功能注释分析解释了构树的多种性状,包括适用于造纸,强大的适应能力及高含量的黄酮类和萜类。
另外,与苜蓿、毛杨和甜橙相比,转录因子发生明显收缩(58个家族共1,342个转录因子,占蛋白编码基因的4.4%)。肌动蛋白在植物的生长和发育的很多层面扮演着重要的角色,在酵母和很多动物中,肌动蛋白仅被一个单基因编码。在构树中仅发现4个肌动蛋白,少于藻类、小立碗藓和无油樟。系统发育树显示,所有肌动蛋白基因都来自于一个祖先成员并被分成三个进化枝,见图8。构树肌动蛋白不存在古代进化枝中,其在过渡进化枝中出现并在高级进化枝中复制,就像桃树一样。因此,肌动蛋白家族与植物物种经历了非同步进化。
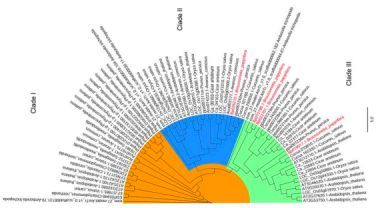
图8、肌动蛋白基因家族的系统发育树
4、构树中纤维素合成分析
制浆和造纸对原材料的要求主要是纤维的长度和宽度。纤维的化学成分由Klason木质素和果胶组成,表明构树十分适合造纸。纤维素由位于质膜的纤维素合酶(CesA)复合物(CSCs)合成。虽然已经预测高尔基体中的STELLO蛋白与CesA相关,但是CSC的合成机制一直都不清楚。在拟南芥中,初生壁CSCs主要包括CesA1、CesA3和类CesA6亚基(CesA2,5,6,和9),而次生壁CSCs由CesA4,7和8组成。根据构树中CesA基因的比对结果和表达谱发现,Bp02g1468,Bp08g1552和Bp12g0339编码CesA基因,进而形成初生壁CSCs,而Bp05g1241,Bp08g1551和Bp11g1484编码的CesA基因很可能组成了次生壁CSCs。因此,Bp05g1241,Bp08g1551和Bp11g1484被认为在纤维素的合成中发挥着重要的作用。
碳状态是影响细胞壁合成率的主要因素。在构树中,碳酸酐酶(CA)活性的功能是碳同化和光合作用,无论是正常生长还是胁迫条件下,构树中的CA含量均高于桑树。本研究的14个CA基因中,在幼嫩和成熟叶片中Bp11578的表达都很高,可能作为主要的CA基因,为细胞壁成分(例如纤维素)合成提供足够的碳。
UDP-葡萄糖是纤维素合成的底物,还是半纤维素和果胶合成必须的核糖核酸前体物质,蔗糖磷酸合成酶(SPS)和蔗糖合酶(SS)是UDP-葡萄糖合成过程中主要的酶。在构树中,UDP-糖基转移酶基因家族明显扩张,这意味着其在纤维素合成中的潜能。根据表达模式,Bp02g2347(编码SPS)和Bp11g1146(编码SS)主要调控构树树干中的纤维素的合成。
5、紫丁香基木质素主要影响构树在树皮布和造纸中的应用
很多研究已经证明,木质素含量及其单体组成是影响制浆和造纸的主要因素。尤其是紫丁香基木质素与造纸制浆率、饲料可消化性和生物乙醇的产量紧密相关。F5H或COMT的遗传调控能严重影响紫丁香基木质素的合成,从而提高制浆效率,COMT的下调能成功降低紫丁香基木质素的合成。在很多植物中,COMT的破坏可导致木质素单体S的严重减少,并伴随着不寻常的5-OH木质素单体G的进入。木质素单体的比例决定木本植物的制浆效率,S/G值越高,木质素越容易降解。
构树基因组中有53个木质素合成相关基因,而桃树和白杨中分别是87个和99个。基因编码C3H,CAD,CCR和F5H的基因数量显著低于其他植物。然而,COMT基因家族成员的数量与其他植物相比增加了近两倍。此外,淀粉和蔗糖代谢、苯丙烷合成和激素信号转导在树干中的差异表达基因中占了很大的比例(见图9)。
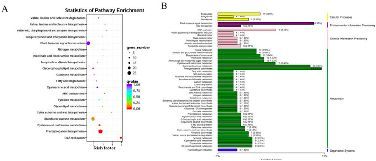
图9、扩张基因家族的功能注释
注:A,KEGG注释统计;B,GO注释统计
因此,作者提出木质素合成相关基因家族的收缩降低了构树木质素合成生产力。与此同时,COMT基因家族的扩张增加了木质素S单体的水平,从而增强了制浆率。该结论被之前构树S/G值(2.7)高于白杨(1.1)和柳树(1.8)的研究报道所证实。这些遗传机制可能解释了构树为什么从古代就是树皮衣和造纸的合适选择。
6、构树中黄酮类和木质素生物合成的遗传基础
苯丙氨酸是黄酮类和木质素的共同前体物质。令人惊奇的是,在黄酮类合成通路中,4个主要的酶家族(CHS,F3'H,I2'H和DFR)显著扩张。这些家族的基因数量几乎比得上大豆、苜蓿和葡萄,远远超过桑树、白杨、拟南芥和桃树。这些扩张的基因家族可能部分地揭示了构树的黄酮类丰富及对疾病强大的抗性。同样的现象也出现在银杏科。
在类黄酮合成中,查耳酮合酶是第一个限速酶,见图10,在构树中已经扩张到16个基因。在大多数植物中(除了葡萄和豆科植物),该酶目前没有超过10个拷贝数。序列比对说明这些基因是通过连续串联重复扩张的。

图10,苯丙烷代谢途径说明类黄酮生物合成的增强和紫丁香基木质素单体比例的增加
注:红色和绿色字体分别代表基因家族的扩张和收缩
与木质素合成相关基因相比,根中参与类黄酮合成中的大多数关键基因具有高表达和竞争力,证明了根中有最低的木质素含量。然而,与类黄酮合成相关基因相比,树干中参与木质素合成相关大多数基因是高度表达的,因此,在竞争且达到平衡后,树干中有高的木质素含量和低的类黄酮含量,这可能解释了构树叶片中类黄酮总含量是4%,而根中的木质素含量是10.5%。以上结果表明,构树中黄酮类化合物的合成比木质素的合成增加,特别是在根和叶中。
7、构树与微生物的共同进化和相互作用
从构树根相关微生物中共鉴定到1,895个细菌OTUs和533个真菌OTUs,见图11。在构树内部,科水平注释到的大多数细菌是根瘤菌科(21.5%)、假单胞菌科(16.6%)和肠杆菌科(13.6%)。种水平上,根瘤菌(19.1%)和假单胞菌(18.8%)是优势菌。在根际,假单胞菌在种水平上是最丰富的。在内部和根际分别有67和170个常见和核心OTUs,包括鞘脂单胞菌科、假单胞菌科和根瘤菌科。该组成与豆科植物很相似,但是与白杨差异较大。
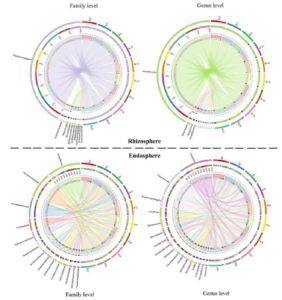
图11、构树根际和内生的共生细菌群落
构树根际和内生的真菌群落是相似的,3个优势群是担子菌门(~69.2%)、子囊菌们(~24.3)和接合菌门(~4%)。另外,在内生和根际都发现了类似的常见和核心PTUs,包括Agaricales、Coprinellus, Tremellales, Hannaella 和Tuber。同样地,大豆中子囊菌门的比例高于担子菌门。美洲黑杨的内生真菌包括子囊菌门(60%)和担子菌门(30%),而其根际真菌是子囊菌门(50%)和担子菌门(20%)。
不同种群的构树也具有细菌和真菌的共同核心和优势OTUs,见图12。尽管所处的生长阶段和条件不同,但是构树根际微生物在所有样本中有很大的相似性,这表明这些共生微生物在很长的历史时期内与构树共同进化和协作,形成了一个遗传上的共生关系。
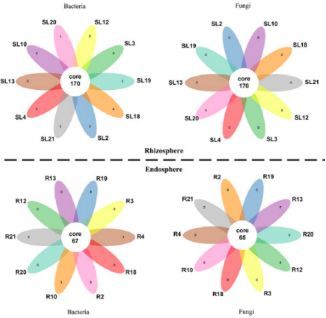
图12、韦恩图展示共生微生物的特有和共有OTUs
类黄酮是植物-根瘤菌共生微生物的信号分子。研究证明,根系分泌物中的黄酮类是几种致病共生植物-微生物相互作用的信号化合物,并且黄酮类渗出物的增加改善了固氮作用和菌根共生。由构树合成的大量各类黄酮和异黄酮,成为共生微生物的信号分子。把黄酮类化合物的扩张考虑在内,作者推测构树通过增加黄酮类的合成可能富集微生物并形成互利关系。
此外,黄酮类在植物防御中起重要作用。许多假单胞菌也可以增强共生植物的抗性,并帮助植物从土壤中吸收磷和钾等矿质元素,从而在植物生长促进中发挥作用。因此,作者提出黄酮类和共生微生物增强了构树对各种环境胁迫的适应性。
黄酮类化合物还具有抗菌、抗真菌、抗病毒和抗癌活性。黄酮类和异黄酮类已经被证明是药用成分,可以解释构树的药用价值。很多食用和药用真菌与构树共生,这些生物体的代谢产物也具有抗炎和抗肿瘤的作用。因此,作者推断,由于构树高含量的黄酮类化合物以及与微生物的共生,其被用作传统医药和饲料。
8、共生微生物可能有助于构树的固氮作用及紫丁香基木质素可能调节其作为饲料的消化性
植物为微生物提供碳能,反过来微生物为植物提供氮和磷等营养物质。即使在非豆科植物中,大琪固氮也占玉米氮营养的29-82%。研究发现,黄酮类化合物作为信号分子在促进共生菌形成结节方面起着至关重要的作用。在本研究中,共有36个结瘤信号或类结瘤蛋白基因在组织中表现出差异表达,这意味着它们可能被类黄酮和其他组织特异性的化合物所诱导;这些基因被考虑作为通过黄酮类分泌形成的构树和细菌间共生关系的关键候选基因,其可能参与相关的固氮以提供足够的氮源。
最重要的是,构树被用作饲料来源,不仅仅是叶子中高蛋白含量。其他营养成分的组成包括粗纤维、木质素单体比例、碳水化合物、脂肪、钙、铁和维生素,共同作用使得构树比苜蓿和其他木本饲料更易于消化和吸收。许多研究已经证明,木质素含量和单体组成,尤其是紫丁香基木质素,是影响饲料消化率的因素。F5H或COMT的遗传调节可显著影响木质素的生物合成,从而提高饲料的消化率。因此,COMT基因家族的扩张可能是其作为饲料易于消化的原因。
简而言之,本研究发现构树在如何处理代谢前体方面存在偏好性。优良的纤维质量和合适的S/G木质素比例,使得构树的茎比其他木本植物更适合于树皮衣制作、造纸、制浆、动物消化和蘑菇生长。黄酮类和异黄酮类赋予构树重要的医学价值,募集更多的共生微生物以提供氮和其他有机营养或矿质元素,反过来也增强了构树的抗性和适应性。此外,与豆本植物相似的共生细菌组成,产生丰富的粗蛋白,使构树成为一种优质的饲料。构树的特性与相互促进和制约的古老中国哲学是一致的,见图13,表明构树是一种非常复杂非凡的树种。此外,人类可以从构树和微生物的共生中受益。

图13、在物种进化历史中构树与周边生态环境的共同进化、协作与相互促进关系
- 本文固定链接: https://maimengkong.com/zu/982.html
- 转载请注明: : 萌小白 2022年6月5日 于 卖萌控的博客 发表
- 百度已收录
