我们接着讲生物信息学的资源,之前我们介绍了NCBI,欧洲也有一个非常大的centralized resource,叫EBI 。
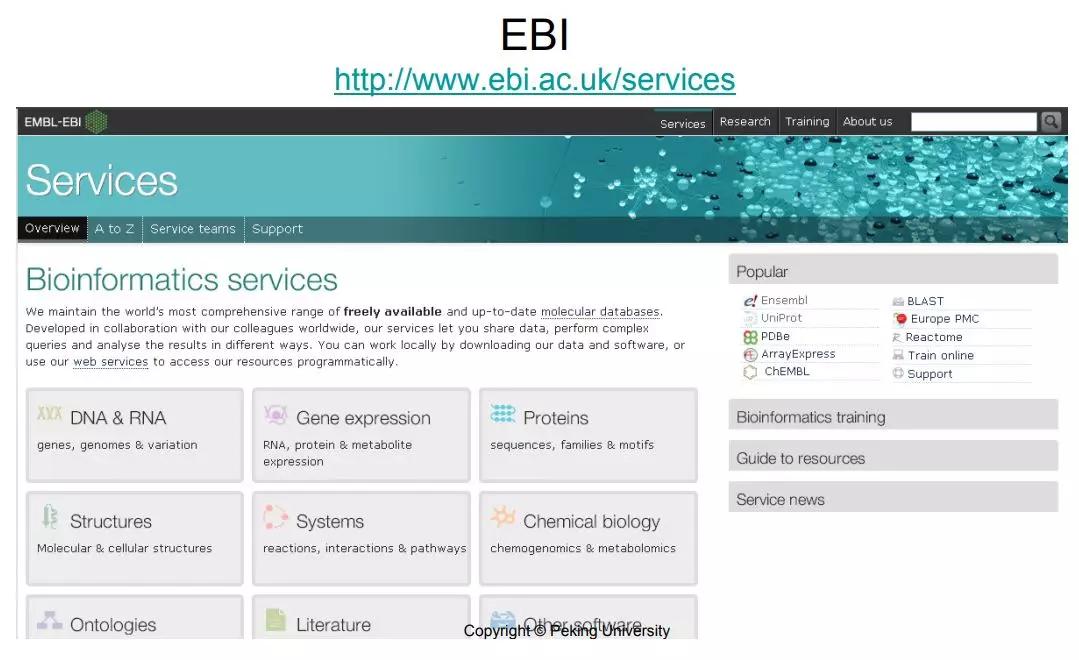
和NCBI有点类似,也是一个包含各种各样数据库这样的资源,它主要也是针对从序列到蛋白到蛋白结构到表达到通路到Ontology各个方面。

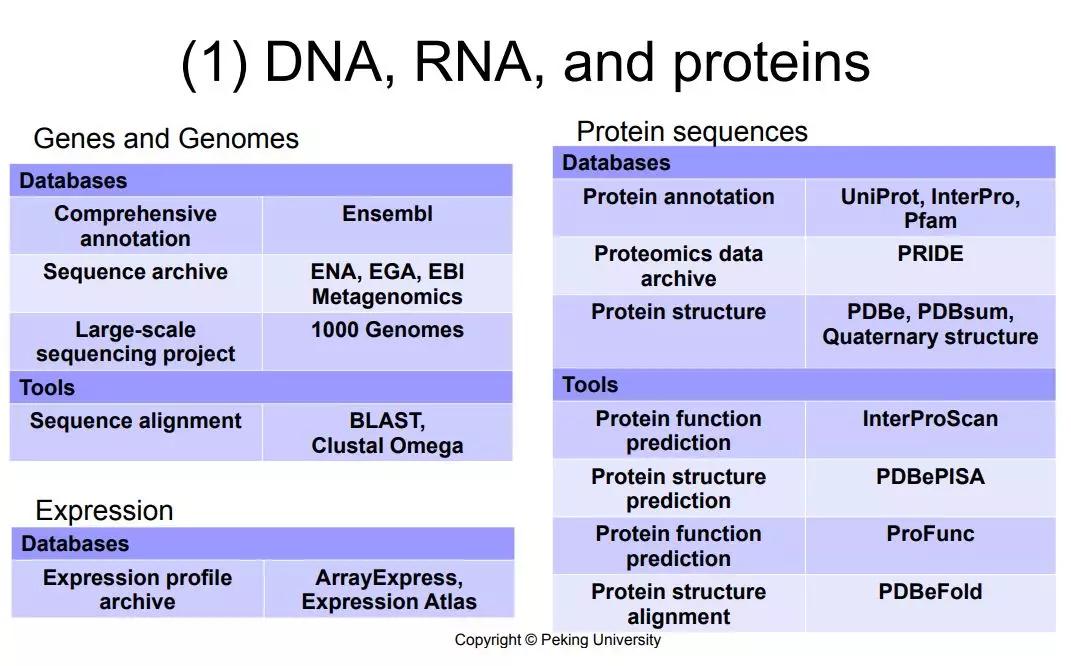
首先在DNA,RNA和蛋白的方面,基因和基因组最主要是有一个ensembl,很多人不知道EBI也会知道ensembl,是一个整合的基因和基因组的一个资源,另外也有一个原始数据,比如说像Metagenomics的一些数据,还有大规模测序数据的一些资源,比如说千人基因组。工具上有BLAST,Clustal Omega等等。
在蛋白层面,有UniProt,这是蛋白的一个Reference 和InterPro,这是所有蛋白功能区间整合的一个数据库,蛋白质组学的数据也有很多的数据,主要在PRIDE里面可以找到,EBI也有一系列蛋白的三维结构的数据库。基于它前面的原始数据和二级数据,EBI也有一系列的软件工具,包括InterProScan,就是针对InterPro收集的这些蛋白家族构建的功能区域 的motif来预测你输入的序列有没有哪些已知功能区间,还有以下几个蛋白功能预测的工具等等,在表达方面也存储了很多表达数据,在前面的这些年主要是芯片的数据,现在主要是RNA-seq的数据。
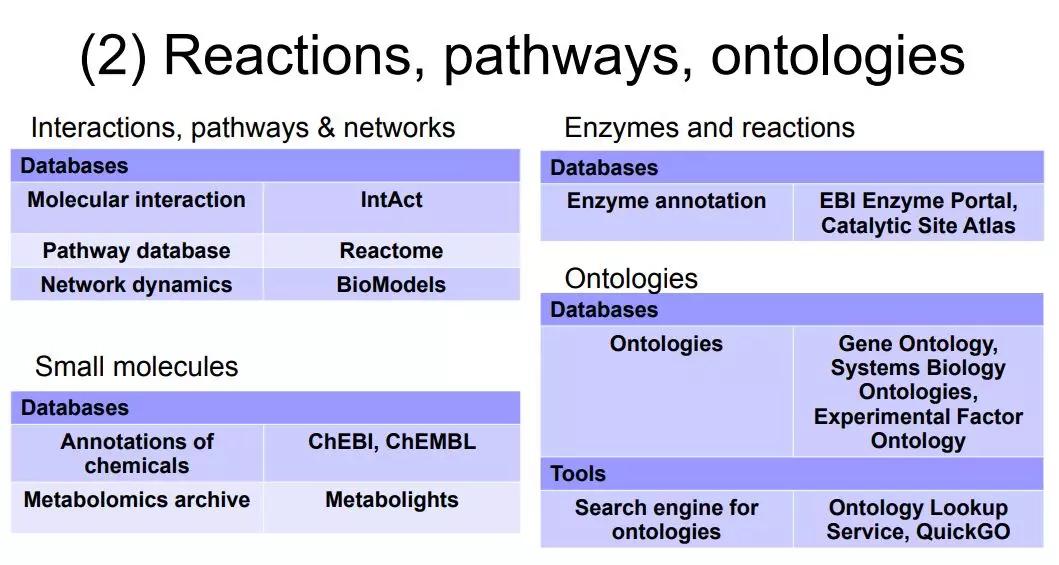
EBI做得比较强的一点是有很多相互作用,通路和反应Reaction的数据,比如说有IntAct分子相互作用的数据,有Reactome通路的数据库和BioModel, 主要是网络的动态特征和模型。它有一个比较庞大的蛋白酶数据集,另外还有若干个小分子数据库。Gene Ontology是我们上一次课讲得比较多的,在EBI的资源里,还有系统生物学本体论,和实验生物学本体论等等 。


选其中的几个简单地做一个介绍 首先是大家用的很多的Ensembl,Ensembl界面有点介于NCBI和UCSC的资源,它也整合了很多很多的物种的数据。

像这里列出来的是整合的一部分物种,还不是完整的列表,这个页面可以下载到完整的列表,可以看到有没有感兴趣的物种。

如果来看人的数据,现在一共有30多亿的碱基对,比较强的证据大概有20000多个基因 另外有9000多短的非编码基因,有13000多长的非编码基因,还有14000多的假基因,假基因是指原来是coding gene,后来由于功能的冗余, 或者说他们上面逐渐累积突变,fitness没有明显降低,所以会留下来,另外还有各种不同的遗传变异,都在ensemble的数据库可以找到 ensemble的data主要有下面几大类。

一大类是有专职人员读文献来建reference就是专家看过并且整合好的数据集,还有是没有人看过的高通量实验产生的原始数据集,还有不是高通量也没有看过的用户提交的一些数据。另外也有一些完全用计算方法生成的数据集, 这几类各自都各自的意义,在ensemble里都有整合,每一类都有不同的标识 。

举几个例子,比如说序列就有clone set ,里面有一些预测出来的转录起始位点,variation在UniProt里面有人 给看过的表型的信息,像HapMap测了很多人种的不同的 个体的基因变异,这个很多没有一个一个地看过就属于Large-scale project的数据 那另外还有就是James Watson的基因组可以在Ensembl里面看到。
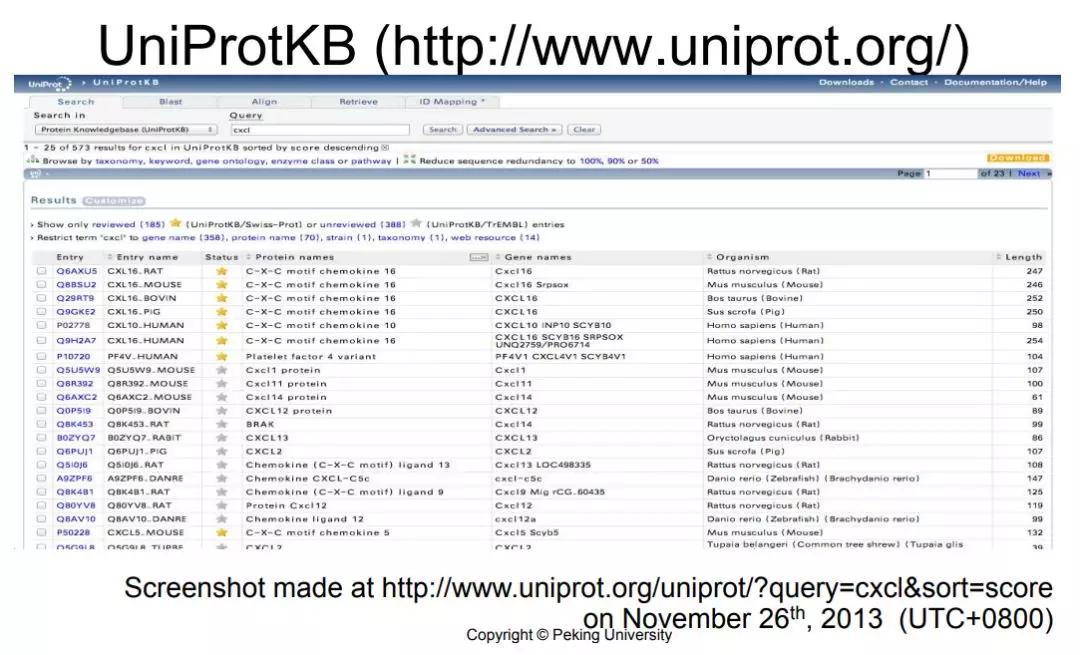
另外还有UniProtKB,它的前身是PIR,再前身是1965年时,Dayhoff 最先出版的一本书Protein atlas 后来逐渐演变成网上数据库 后来基本上现在都是整合到UniProt里面,Unified protein knowledge base,所以看到大一统也是领域的趋势之一。
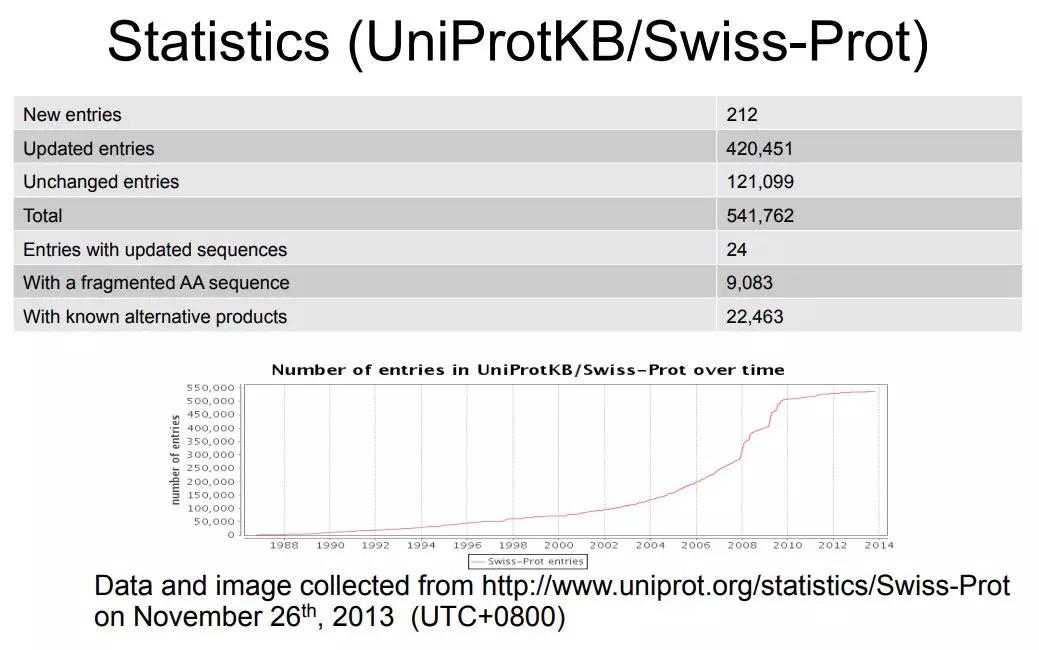
现在UniProt数据库里又分两类,一类是Swiss-Prot,还有一类叫TrEMBL,这两类有什么差别呢?Swiss-Prot是人工校对过的蛋白的集合,现在一共有54万条 看到也是一个增长的趋势。
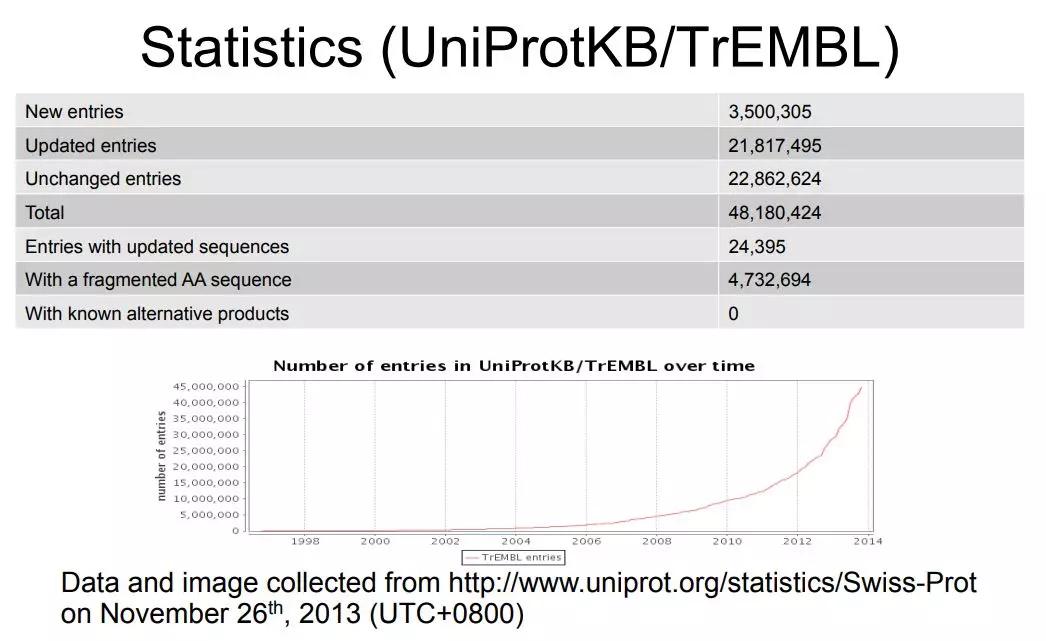
TrEMBL存储的是还没有来得及人工校对的序列,所以 TrEMBL一共有4800万,这个增长趋势就更加陡峭。

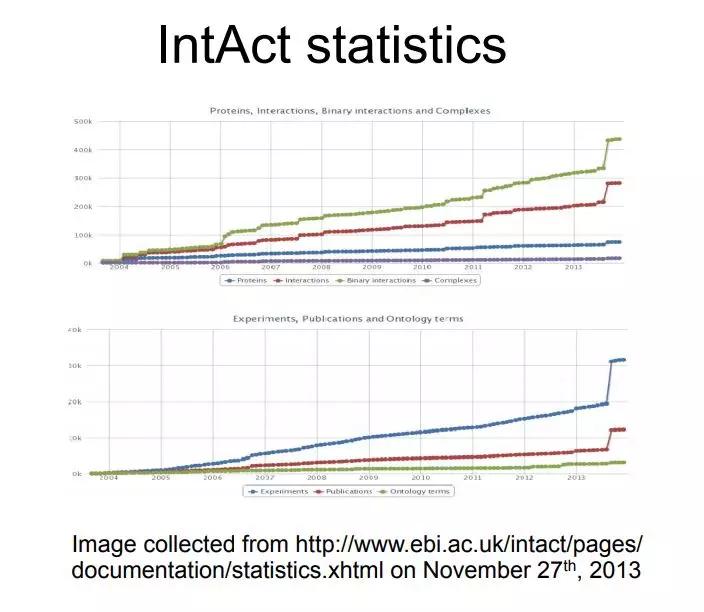
EBI另一个有用的资源叫IntAct IntAct存储的是分子之间相互作用,目前是有43万个不同的相互作用,其中涉及到将近8万个蛋白和小分子。他们的信息来自于10000多文献,这些文献报道了30000多的实验,所以可以看到这么大量资源对于你的研究应该是很有帮助的,因为不管你现在研究的是什么物种和什么家族,如果上网仔细搜索,都会有很多相关的数据和信息,如果你没有上过生物信息学的课,不知道这些数据和信息存在的话,其实是非常危险的。不光是说浪费点时间,有可能这个课题有的信息你就不知道,整个课题就会有很致命的伤害,所以我们建议大家,即使将来不做生物信息学的研究,也要把这些数据当作文献一样,在做一个课题之前,都会去检索一下文献, 看看别人已经研究什么,做完文献检索之后,马上就要检索一下数据,看看已经有什么数据了。
已经知道了什么数据,有什么低通量的数据,有什么高通量的数据,这应该成为研究的一部分。

IntAct这个数据库也是有较快的一个增长, 它的实验技术包括tandem affinity purification,酵母双杂,pull down等几大类实验方法。相互作用类型最多的是 物理上的藕联,还有一些是不确定哪一种association,有一些是co-localization,只是说他们共同的定位 涉及的物种最多的是人和酵母,然后是小鼠果蝇线虫E.coli等等 其实也是围绕着最重要的这些模式生物。
EBI很重要的一个资源就是Clustal Omega,是做多序列比对的一个资源,另外一个是InterProScan 。

就是输入一个序列,找这个序列是否包含任何已经知道的蛋白功能区域 看到这里的显示,InterProScan整合了324个新的方法, 如果没有InterPro的话,你可能就要把这300多方法一个一个地跑一遍,他们结果的格式 也不一样,还要逐个看一遍,所以这样整合的资源价值是很大的 ,下周我再用一个Unit给大家介绍一下UCSC的Genome Browser。
- 本文固定链接: https://maimengkong.com/zu/1576.html
- 转载请注明: : 萌小白 2023年6月17日 于 卖萌控的博客 发表
- 百度已收录
