群体进化第一期带大家实战了基于群体SNP的PCA分析,相信大家都已经练得很熟了,那我们今天带大家进行实战进化树分析,依然含分析和绘图代码哦。
系统进化树(Phylogenetic tree,又称Evolutionary tree进化树)是描述群体间分化顺序的分支图或树,用来表示群体间的进化关系。根据群体的物理或遗传学特征等方面的共同点或差异可以推断出它们的亲缘关系远近。
实战之前我们先熟悉一下进化树的分类。
有根树和无根树
有根树:是具有方向的树,包含唯一的节点,将该节点作为树中所有物种的最近共同祖先。反映时间顺序。
无根树:是没有方向的,只指明种属的相互关系,没有确定共同祖先,线段的两个演化方向都有可能,树枝长短表示两个物种之间进化距离的远近。反映进化距离。
标度树和非标度树
标度树:分支的长度代表分化程度。非标度树:分支只表示进化关系,支长无意义。
物种树和基因树
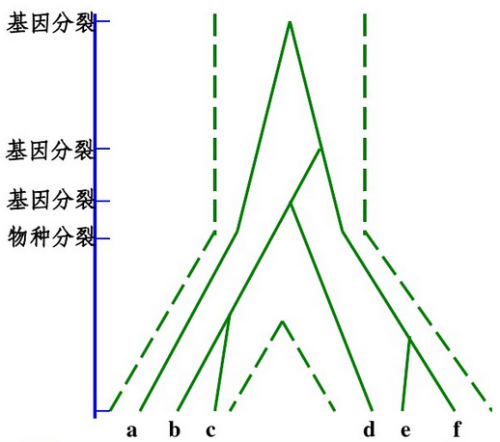
物种树:代表一个物种或群体进化历史的进化树,两个物种分歧的时间即是两个物种发生生殖隔离的时间。
基因树:由来自各物种的一个基因构建的进化树,分歧时间代表基因分离的时间。
举例来说:对于某一被研究的基因,可能存在种内多态性,即在物种分化之前,该基因可能已经开始分化。两物种间该基因的分化时间可能早于这两个物种的分化的时间,根据这个基因计算而来的分支长度(分歧时间)可能偏离。
进化树分析实战
群体进化分析中的进化树构建是基于群体SNP的VCF文件进行的,如果样本数目比较多,VCF文件也会比较大,通常在构树前都要对原始的VCF文件进行过滤,过滤的标准有:SNP在样本中的缺失率、质量值、多态位点、深度、次等位基因频率等。SNP过滤后得到用于构树的VCF文件,接下来就是进化树构建的实战部分了!
以下为实战数据链接:https://pan.baidu.com/s/1flth4p-ORJakQbQqwkXdxQ 提取码:k6tr
VCF文件格式转换为fasta格式
下载一个python脚本:(https://github.com/edgardomortiz/vcf2phylip),然后用vcf2phylip.py将VCF文件转成fasta格式(也可以选择转为phylip、nexus格式,如果有外群也可以指定外群),该脚本用法如下:
usage: vcf2phylip.py [-h] -i FILENAME [-m MIN_SAMPLES_LOCUS] [-o OUTGROUP] [-p] [-f] [-n] [-b]
-h, --help show this help message and exit
-i --input 输入vcf文件,甚至是可以压缩后的vcf文件
-m --min-samples-locus 缺失的个数,每个位点至少有多少个SNP,默认值为4
-o --outgroup 输入外群样本ID,这样外群样本会放在第一个。
-p, --phylip-disable 输出格式为phy
-f, --fasta 输出格式为fasta
-n, --nexus 输出格式为nexus
-b, --nexus-binary 输出格式为二进制
具体使用命令:
./vcf2phylip.py -i test.vcf -f
得到的结果文件test.fasta。
MEGA进行进化树构建
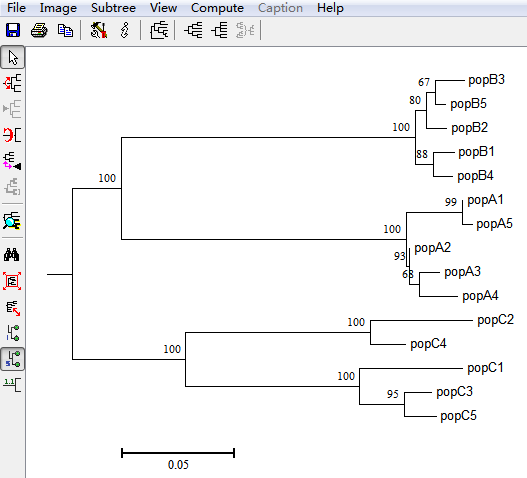
用于构建进化树的软件有很多(比如:MEGA、Phylip、RaxML、MrBayes等),这里我们使用大家常用的MEGA软件来进行进化树构建。具体步骤是:打开MEGA软件,File--导入文件test.fasta--Analysis,Data Type选择Nucleotide Sequences,然后建树方法选择neighbor-joining(NJ),点击“PHYLOGENY”→“Construct/Test Neighbor”计算模型可以选择p-distance,最终将构建的树保存为test.nwk。
进化树美化
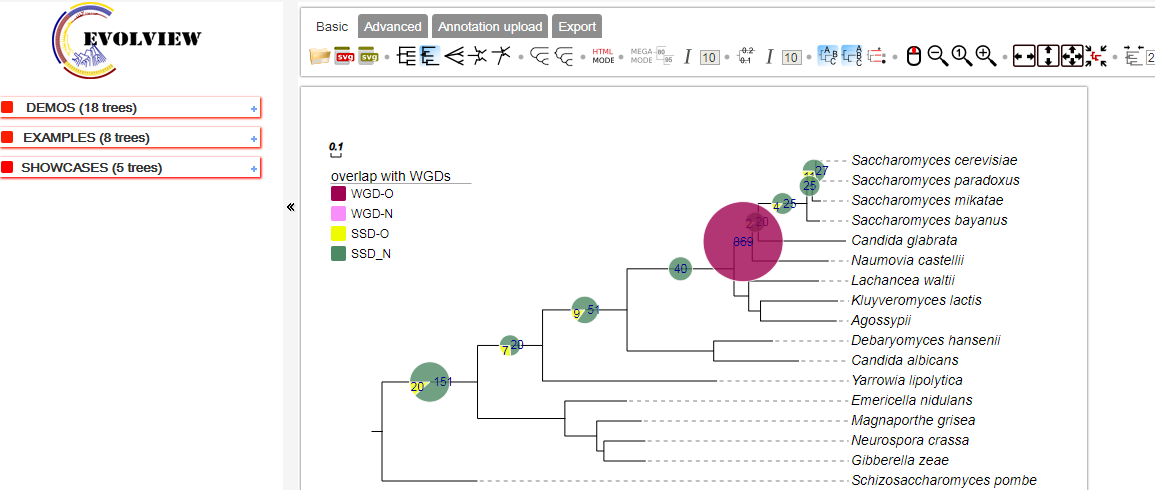
因为MEGA显示的进化树不是很美观,我们需要借助一些软件来美化一下。这里给大家推荐一款在线软件Evolview:
(https://evolgenius.info//evolview-v2/)。该软件不支持IE和360浏览器,建议用谷歌打开。下面是登录注册后,打开的页面如下:
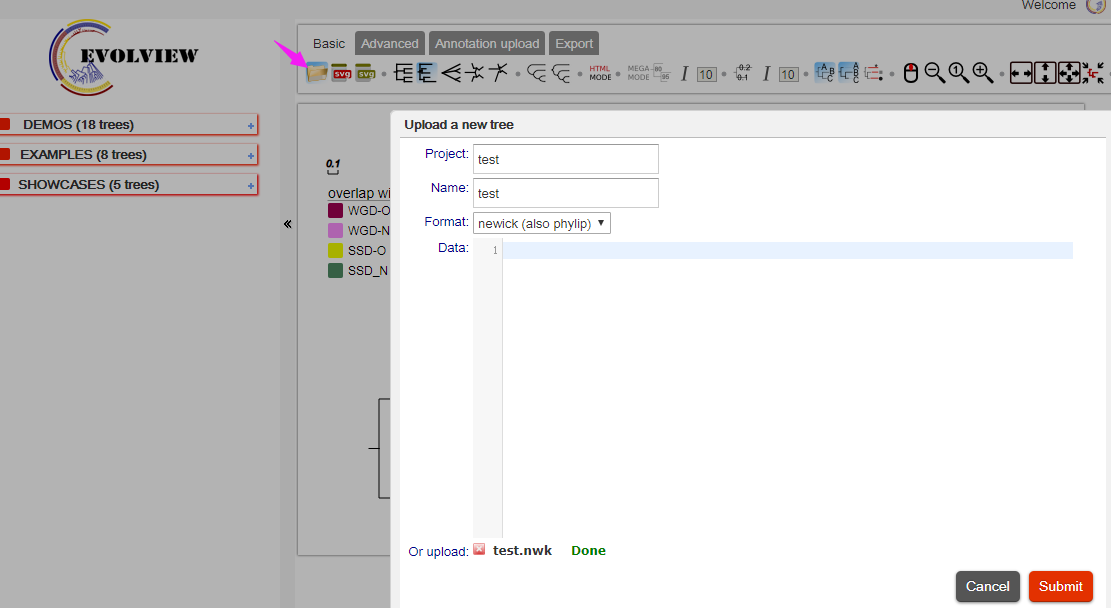
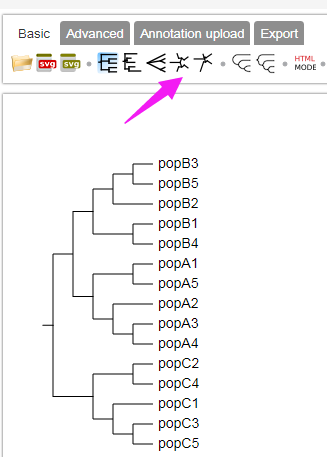
Step1:点击“Use without an account”→“upload tree file”→“test.nwk”(##点击下图箭头位置,导入进化树文件,点击“Submit”上传树文件)。
Step2:点击箭头所指的位置,修改树的形状为圆形。
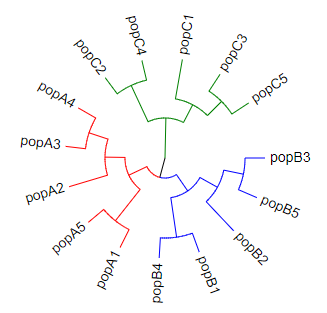
Step3:对连续的树枝进行上色。点击“Annotation upload”→“unload data for coloring branches(help)”→“branch.txt”,配置文件branch.txt中列与列之间采用Tab键进行分割,具体文件格式如下:

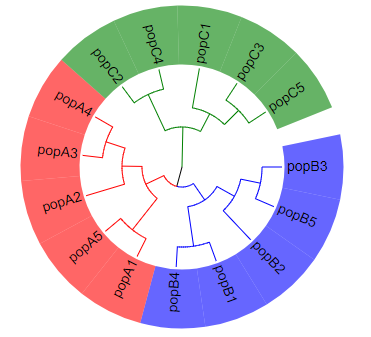
Step4:对连续的树枝背景进行上色。点击“Annotation upload”→“unload data for leaf label(help)”→“branch.txt”,为了保持群体分支和背景颜色一致所以依旧用branch.txt。
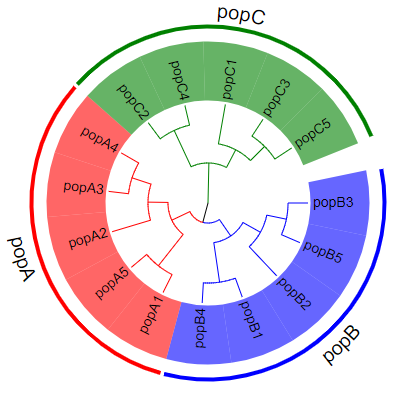
Step5:按照样本分组指定群体。点击“Annotation upload”→“unload data for group label(help)”→“group.txt”。
以上就是基于群体SNP进行进化树分析和图形美化的方法,如此简单,赶快用自己的数据试试吧!
如果大家实战分析的过程中有任何疑问,可在文末留言或者邮件交流(genome_support@personalbio.cn),欢迎和我们一起探讨。下面是动植物产品线的产品类型,如果对其他产品比较感兴趣的,也可以邮箱反馈我们,后续我们一一为大家安排。
那下一期《群体进化之群体遗传结构分析》再见喽!
- 本文固定链接: https://maimengkong.com/zu/1425.html
- 转载请注明: : 萌小白 2023年4月12日 于 卖萌控的博客 发表
- 百度已收录
