已经陆续为大家介绍了ceRNA研究的建库方法(文章:),还有ceRNA之间的调控关系(文章:),这篇就来总结一下miRNA、 lncRNA、circRNA鉴定及靶向关系预测常用的分析工具。
miRNA鉴定
去除低质量的数据,得到clean data后,还需要和Genebank、Rfam等数据库比对去除rRNA,scRNA,snoRNA,snRNA,tRNA等其它小RNA的tags,最后再进行miRNA的鉴定,鉴定包括寻找已有的miRNA和预测可能的新的miRNA。
①鉴定已有miRNA
和miRBase数据库中收录的miRNA比对进行鉴定。
miRBase(http://www.mirbase.org/)是目前收录最全面的miRNA信息的数据库,可以在线输入miRNA的accession、name检索miRNA,或通过序列信息进行blast比对。也可以免费下载数据库信息,进行本地分析。结果会给出对应的茎环结构和成熟的序列、验证方法(实验或者测序)、预测的靶基因等信息。
利用数据库和已经收录的对应的物种miRNA进行比对,鉴定已知的miRNA。对于目前数据库未收录的物种,或者只收录了部分信息的物种,也可以和数据库中其他已知的植物或动物miRNA进行比对,利用miRNA在不同物种中的保守性进行鉴定。
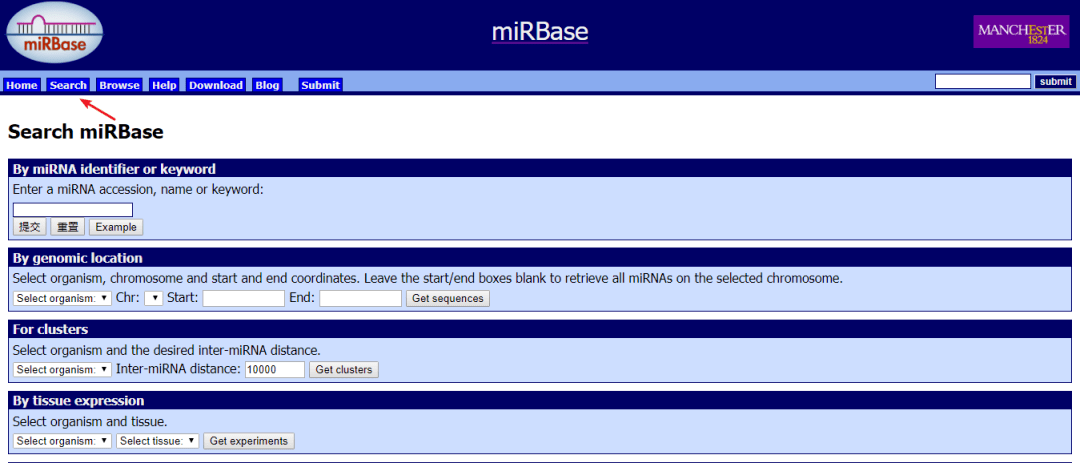
图1 miRBase数据库搜索界面
②预测新miRNA
利用miRDeep2预测新的miRNA。
miRDeep2(https://github.com/rajewsky-lab/mirdeep2)是2011年推出的用于NGS分析的软件,到目前为止参考文献已经引用了1500+次,不仅可以对miRNA进行定量,而且有了测序结果和参考基因组,就可以鉴定和预测新的miRNA,是目前最常用的软件之一。
成熟的miRNA是由特殊的茎环结构前体加工而来,所以在有参考基因组的情况下,挑选出能够比对上基因组的tags获得位置信息,然后使用miRDeep2软件,预测miRNA的特殊的二级结构,鉴定新的miRNA。
除了上述提到的两个工具,这里给大家推荐一篇2019年发表在《Briefings in Bioinformatics》上的miRNA分析的综述文章,“Trends in the development of miRNA bioinformatics tools”[1] ,里面详细列举和介绍了常用miRNA生信分析工具,有本地的工具也有数据库,请按需取用。
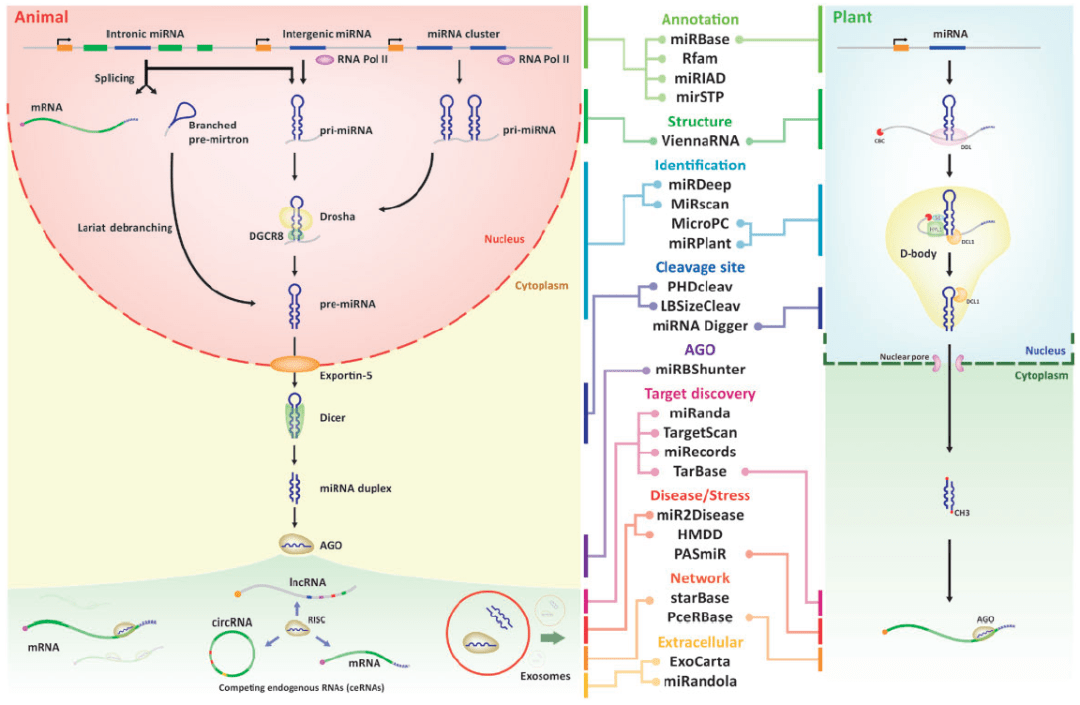
图2 miRNA常用分析工具
lncRNA鉴定
lncRNA的鉴定首先过滤掉低质量数据,得到clean data,和数据库比对去除rRNA,将剩余的reads和参考基因组进行比对。再利用stringtie重构转录本,由于lncRNA长度大于200bp,所以保留转录本长度≥200bp的转录本进行后续分析。使用CPC(http://cpc2.cbi.pku.edu.cn)、CNCI(https://github.com/www-bioinfo-org/CNCI)、PfamScan(http://pfam.xfam.org/)、CPAT(http://rna-cpat.sourceforge.net/)等软件预测新转录本的编码能力,去除掉可编码的RNA,取两个或多个软件分析出的没有编码潜能转录本交集作为lncRNA的预测结果。根据新lncRNA在基因组上相对于蛋白编码基因的位置,再将lncRNAs划分为不同类别。方法同样适用于三代全长数据的分析。
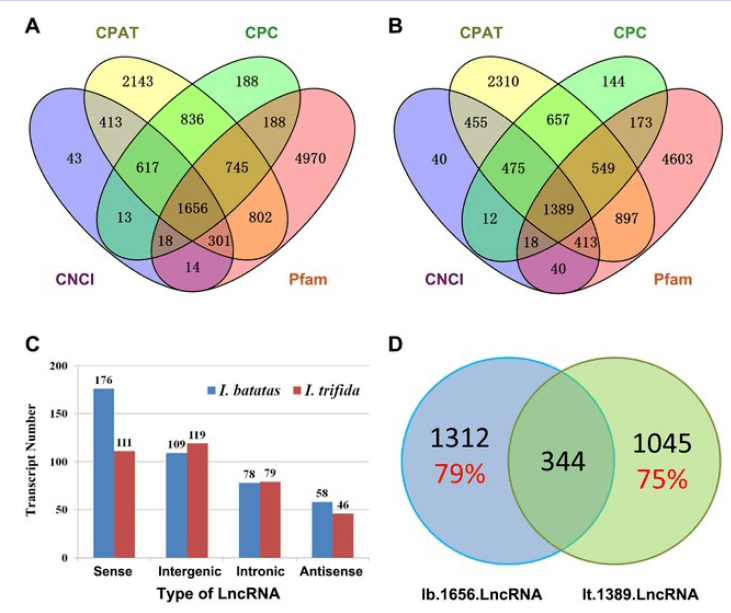
图3 马铃薯中lncRNA预测及分析 [2]
除了利用工具预测,lncRNA也有很多如下的数据库收录了相关信息,可以在线搜索相关信息:
lncRNA分析常用数据库

CircRNA鉴定
由于circRNA与线性RNA的不同点在于环状RNA成环打断后会使成环断点上下游序列方向相对于成环前恰好相反,因此在测序过程中,若成环断点位于某一read上(back-spliced junction),则会造成该read的一端与另一端方向相反,这种reads在比对的时候是没有办法比对到基因组上的。而circRNA的分析正需要这类reads,所以需要从上述的比对结果中提取unmapped reads,然后提取这些reads两端的序列,然后将短reads以单端read为单位比对到参考基因组。这些比对上的reads,称之为Anchors reads。Anchors reads比对到参考基因组上后,将所有样品的比对结果进行合并,得到合并后的结果后使用find_circ(https://github.com/marvin-jens/find_circ)等主流软件进行circRNA鉴定。
以下是其他常用circRNA鉴定和分析工具:
circRNA分析常用工具

上面提到的发表miRNA综述的研究团队,2020年还发表了一篇circRNA的综述,“The bioinformatics toolbox for circRNA discovery and analysis ”[3] 详细列举和介绍了circRNA分析常用的工具,有需要也可以观摩一下。
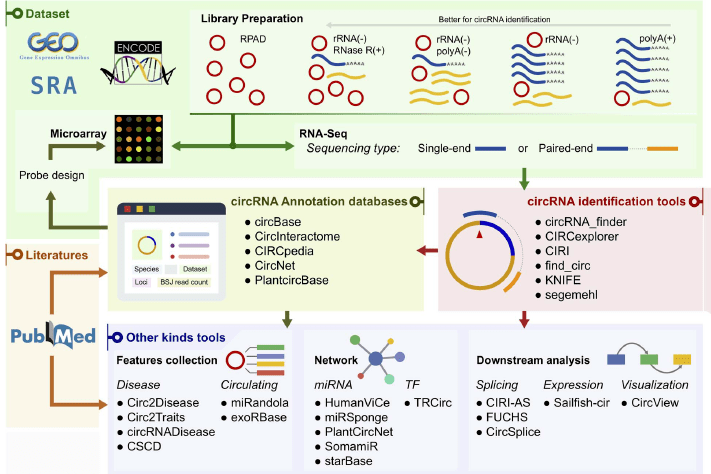
图4 circRNA分析工具流程图
miRNA靶向mRNA、lncRNA、circRNA鉴定
鉴定到了各类ncRNA,接下来还要了解它们之间的关系。
①动物
对于动物样品,miRNA和mRNA为部分结合,产生抑制作用,可以使用基于二级结构等分析的软件进行靶基因的预测。
RNAhybrid(https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid)是基于miRNA和靶基因二聚体二级结构开发的miRNA靶基因预测软件;miRanda(http://34.236.212.39/microrna/home.do)预测依据主要是序列匹配、miRNA 与mRNA 双链的热稳定性以及靶位点的跨物种保守性;TargetScan(http://www.targetscan.org)通过搜索与每个miRNA的种子区匹配的保守位点而预测miRNA的靶基因,同时非保守位点也可预测,并提供每个预测的靶点排名。可以取上述不同方法得到的靶基因预测的结果的交集作为靶基因预测的结果。
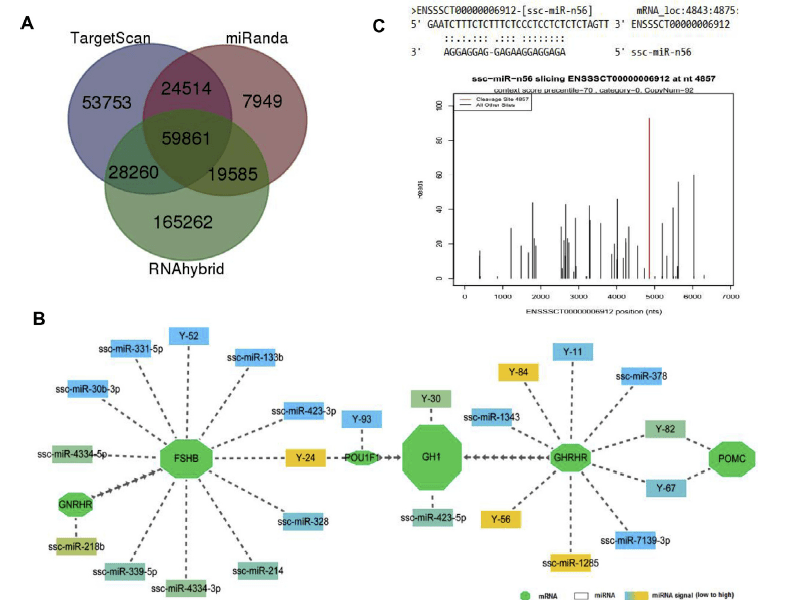
图5 西藏小型猪垂体前叶中miRNA和mRNA互作分析 [4]
②植物
由于在植物中,miRNA和mRNA可以完全配对结合,产生降解作用,可以使用CleaveLand4(https://github.com/MikeAxtell/CleaveLand4)同时可以用于降解组分析的软件、patmatch(https://www.arabidopsis.org/index.jsp)利用拟南芥数据库分析的软件、TargetFinder(https://github.com/carringtonlab/TargetFinder)等软件来进行小RNA和靶基因的互补配对,再通过程序筛选预测得到最终结果。
其他实用miRNA靶基因预测的工具如下:
miRNA靶基因鉴定常用网站

lncRNA靶向mRNA鉴定
lncRNA和mRNA之间的靶向关系分为三种,可用鉴定方法如下:
①Cis:lncRNA位于相同链mRNA的上游或者下游10kb内。
②Antisense:一部分来自于反义链的lncRNA可能因为与正义链的mRNA结合调控可变剪切模式或产生siRNA进而沉默基因、影响其转录及mRNA的稳定性。利用RNAplex的ViennaRNA包, 根据其热力学结构计算最小自由能来预测最佳碱基配对关系。
③Trans:LncRNA和编码基因在位置上没有关系,可能位于不同染色体或者染色体远端的位置,但与其在表达量上存在正相关或者负相关。当样本数≥6时,采用Pearson相关性分析样本间lncRNA与蛋白编码基因的表达相关性。
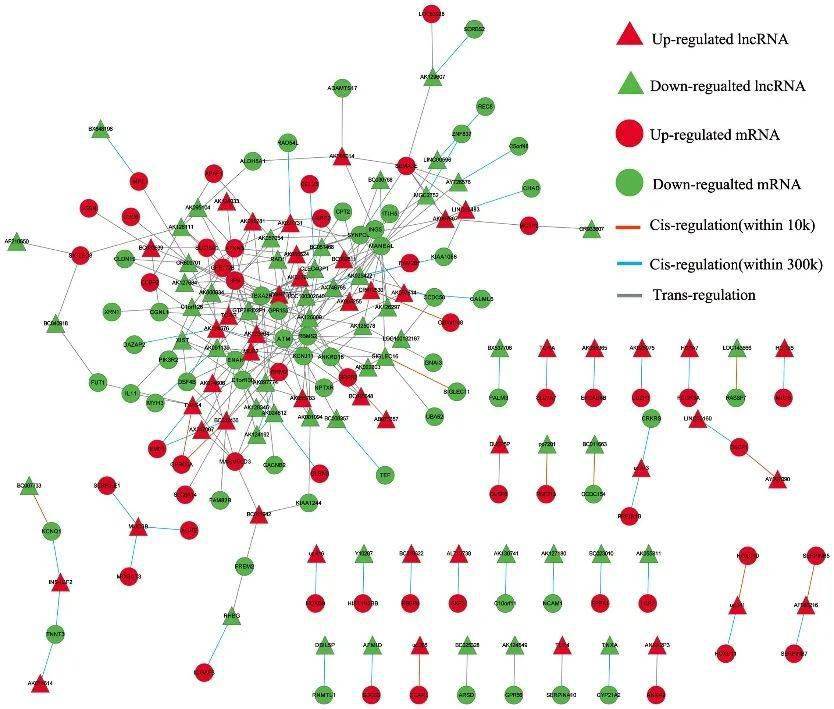
图6 胃癌发病机制研究中lncRNA与mRNA之间的互作关系
此外,前面列举的数据库很多也包含实验验证或者预测出的靶向关系,大家可以自己去探索挖宝了。里面一些数据库还给了TCGA、GEO数据库的信息,不仅有详细的注释,还有表达量。希望上述内容能给大家解决部分不知道靶基因如何找,找到的基因不知道有没有人验证过等等问题。
转自:基迪奥
- 本文固定链接: https://maimengkong.com/zu/1119.html
- 转载请注明: : 萌小白 2022年7月10日 于 卖萌控的博客 发表
- 百度已收录
