LncACTdb 2.0数据库食用指南
Hello,小伙伴们大家周三好~本周我们继续来讨论一下lncRNA的一个大热功能,也就是lncRNA的ceRNA机制。ceRNA是个啥,相信各位小伙伴们已经非常清楚了,无论是酸谈老师的直播还是文献品谈会都介绍了大量的ceRNA的案例文献。在ceRNA的机制中,一共有三个变量参与,以lncRNA为例,最最常见的ceRNA结构就是lncRNA—miRNA以及miRNA—mRNA啦。那如果我们自己有全转录组测序的数据想要分析潜在的ceRNAs,是不是就得分开预测lncRNA—miRNA与miRNA—mRNA,然后合并呢?欸,还真不是,我们今天介绍的主角,lncACTdb数据库就可以把上面三步化作一步解决,一库解决五个数据库的麻烦,简直不要太爽~前面我科老师在挑圈联靠也介绍过了LnCeVar数据库与LnCeCell数据库,同样也是介绍ceRNA关系的数据库,今天我们就站在巨人的肩膀上,继续来看看lncACTdb数据库又有哪些亮点吧~
数据库的网址为
http://www.bio-bigdata.net/LncACTdb/
大家使用的时候不要忘记引用参考文献
Wang P, Li X, Gao Y, Guo Q, Wang Y, Fang Y, Ma X, Zhi H, Zhou D, Shen W, Liu W, Wang L, Zhang Y, Ning S, Li X. LncACTdb 2.0: an updated database of experimentally supported ceRNA interactions curated from low- and high-throughput experiments. Nucleic Acids Res. 2019 Jan 8;47(D1):D121-D127. doi: 10.1093/nar/gky1144. PMID: 30476305; PMCID: PMC6324071.
lncRNA 系列传送门
circ和miRNA都有了,lnc也不能少~
听说你还在为lncRNA的亚细胞定位秃头?
太彪了!一个数据库=一大把时间,精力与金钱
一键搞定高分文章里的装逼技能!超酷炫数据库,各种操作好用到哭!
吹爆这个数据库!能让你懒到极致的就是坠厉害的!
新的套路已经出现,不学你就Out了!!!
两大生信热点的强强联合,这才是发文章的首选套路,你学废了吗?
生信辅佐课题设计,基操,坐下!
一、LncACTdb 2.0数据库基本介绍
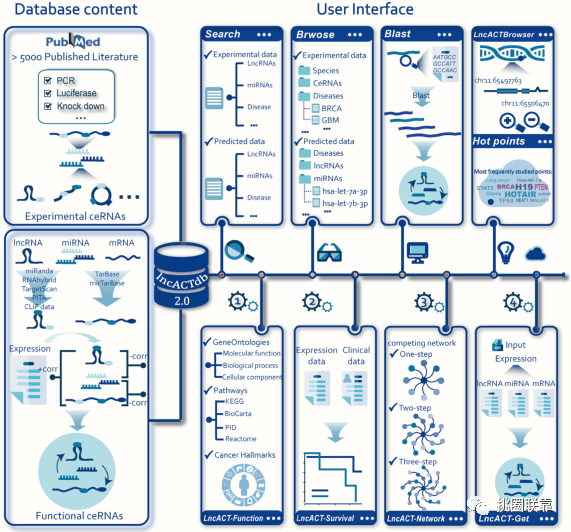
同LnCeVar数据库,LncACTdb 2.0数据库同样由哈尔滨医科大学开发,整个数据库界面清晰简洁,对国人友好。我们可以从下面这张图中看出数据库的构建原理与功能有哪些。大的方向来看,针对ceRNA网络构建,数据库一共提供了两种方法,一种是人工整理审核2018年10月之前的ceRNA相关文章,并从中总结了2663个经过实验验证的ceRNA关系对,包括23个物种,213种疾病的312个lncRNA,131个编码基因,59个circRNA以及16个假基因。其中实验验证包括PCR,WB以及荧光素酶报告基因实验等。另一种即通过生物信息学分析。针对lncRNA—miRNA,LncACTdb 2.0数据库综合了miRANDA,RNAhybrid,Targetscan,PITA,以及Encori五个数据库的结果,而针对miRNA—mRNA,则综合了TarBase与mirTarBase两个以实验验证为基础的miRNA数据库的结果。LncACTdb 2.0数据库进一步结合表达矩阵分析ceRNA中三个分子的相关性来确定最终的ceRNA关系对。因此,在后续的数据库实操讲解中,大家可以看到Predicted data与Experimental data两个模块的内容,因为他们来自不同的分析方法。图片右边则展示了数据库的几个主要功能,我们接下来逐一进行介绍。
二、LncACTdb 2.0数据库实操演示
输入网址http://www.bio-bigdata.net/LncACTdb/,进入数据库主页。

上方菜单栏显示了数据库的功能,包括“Home”,“Search”,“Browse”,“BLAST”,“LncACTBrowser”,“Tools”,“Download”,以及“Help”。Home即为当前主页面,其他几个功能又分为几个子菜单,因此整个数据库的功能加在一起相当丰富。
往下即为快速检索的输入框,用户可以在其中输入lncRNA,miRNA,gene,以及疾病,病变部位等关键词来进行快速检索。此处的快速检索结果同菜单栏其他几个功能检索的结果,一般情况下我们直接进行快速检索即可,若想进一步限制检索条件,则需要在菜单栏其他几个功能中使用高级检索。右侧为LncACTdb 2.0数据库的高频检索词,大家可以点击其中滚动的词条直接链接到相应页面,也是快速检索的一种手段。
1
快速检索
我们首先通过演示快速检索来解读LncACTdb 2.0数据库的结果页面。之后其他几个功能可以链接到相同的结果页面,就不再赘述了。
我们以明星lncRNA HOTAIR为例,点击“Search”:
页面刷新后,显示两个板块的内容:“Predicted data”与“Experimental data”,分别为预测的结果与实验验证的结果,如前文所述,不再赘述。我们首先看预测的结果,下方的表格从左到右依次为详细结果页面链接,预测的ceRNA对三个分子的ID。这里对ceRNA1/2的标识做出一个说明,在常规的lncRNA-miRNA-mRNA关系对中,如果以miRNA作为中心分子,那左侧的lncRNA即为ceRNA1,右侧的mRNA为ceRNA2。因此,表格中的ceRNA1为lncRNA的ID,ceRNA2为mRNA的ID。Activity score为数据库给出的ceRNA对的评分,评分越高,lncRNA与mRNA对于miRNA的竞争性结合越强。因此大家在筛选结果时,可以将activity score优先按照从大到小进行排列,选择前几个关系对使用。“Disease”为该ceRNA对参与的疾病,“Network”,“Function”,“Hallmark”,“Survival”则为LncACTdb 2.0数据库提供的四个主要功能,分别为ceRNA对网络结构图,检索分子富集分析结果,肿瘤生物学过程标志以及生存分析的结果。分别点击这四列下方的小图标可以进入相应的结果页面,点击第一列“Detail”下方的链接同样可以查看相应结果。此处我们则以“Detail”的结果为例进行说明。检索结果可以通过点击“Excel”,“CSV”进行下载。
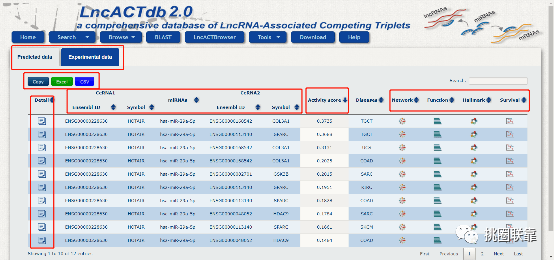
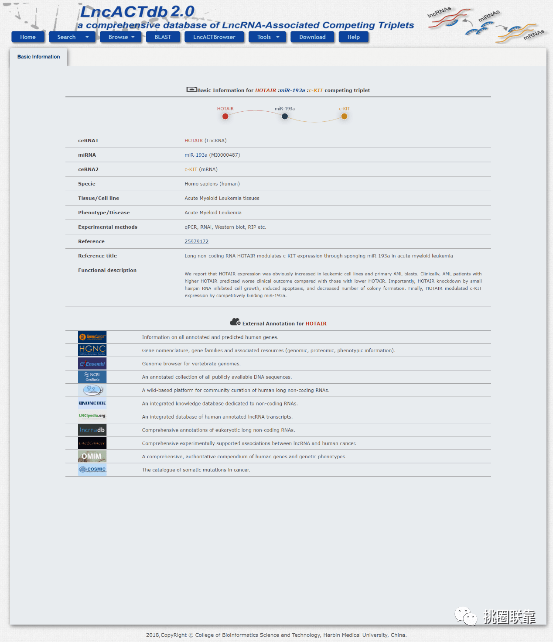
我们以第一条结果“HOTAIR—hsa-miR-29a-5p—COL3A1”为例进行说明。点击“Detail”下方的小图标,跳转到如下界面:
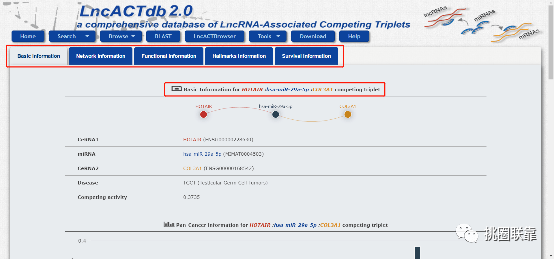
页面刷新后可以看到结果一共分五个部分进行呈现,分别为Basic Information(基本信息简介),Network Information(网络结构图),Functional Information(富集分析结果),Hallmarks Information(肿瘤生物学过程标志),Survival Information(生存分析结果)。Basic Information包括ceRNA中三个分子的基本情况,所研究的疾病及评分:
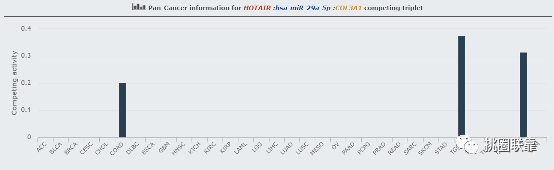
该ceRNA关系对在不同癌症中的评分:
lncRNA与miRNA以及miRNA与mRNA的结合位点预测:

外源数据库对于lncRNA的注释:

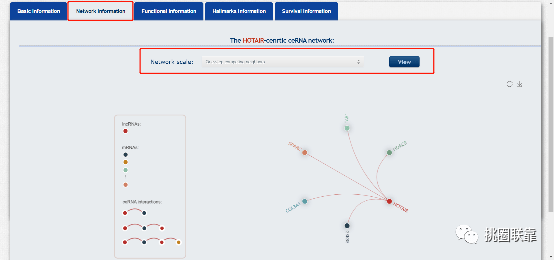
点击“Network Information”可以查看所有与HOTAIR有竞争性结合机制的mRNA:
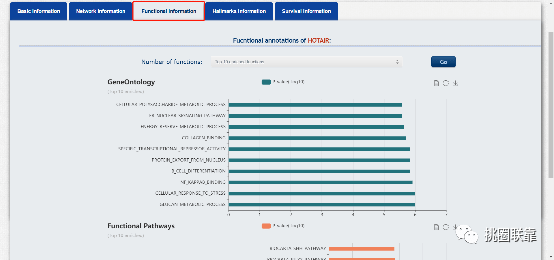
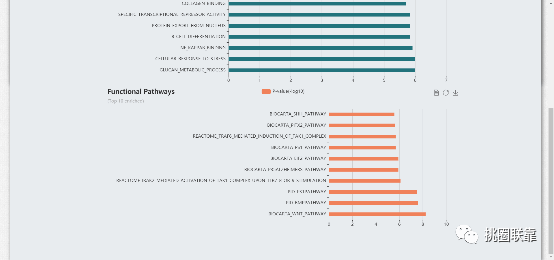
点击“Functional Information”可以查看数据库对于HOTAIR的富集分析结果,包括GO,KEGG等:
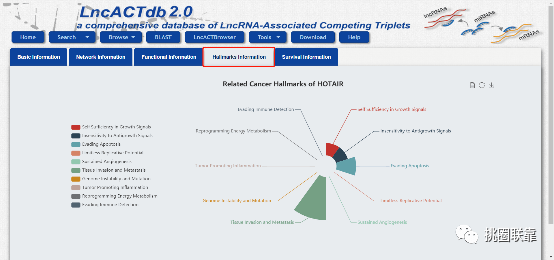
点击“Hallmarks Information”可以查看HOTAIR参与的肿瘤生物学过程:
回到最开始的检索结果页面,点击“Experimental data”可以查看已有文献报道的HOTAIR相关ceRNA对:
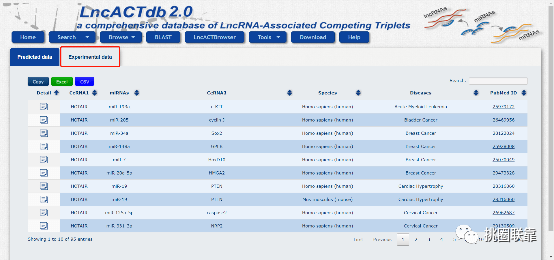
我们以第一条结果“HOTAIR—miR-193a—c-KIT”为例进行说明。点击“Detail”下方的小图标,跳转到如下界面,展示了文献中关于该ceRNA对的内容,包括研究的疾病/细胞系,实验验证的方法,文献的题目与简要概述。
2
“Search”功能
点击“Search”可以看到LncACTdb 2.0数据库将检索分为预测和实验验证两个部分。两个部分的操作相同,此处以“Predicted data”为例进行说明。
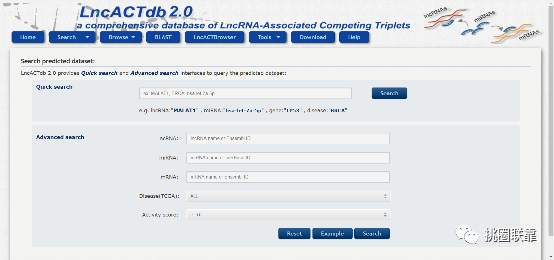
点击“Predicted data”,跳转到如下界面。上方为Quick search快速检索,同前文介绍,此处不再赘述。下方为高级检索,用户可以对疾病与评分进行一定的限制来进行检索:
3
“Browse”功能
点击“Browse”可以看到LncACTdb 2.0数据库将检索分为预测和实验验证两个部分。
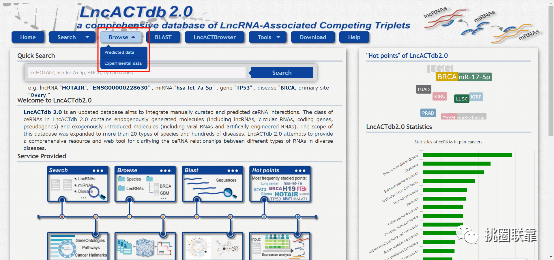
点击“Predicted data”,跳转到如下界面。用户可以根据lncRNA,miRNA,genes以及疾病来进行检索。检索结果同前文所述,点击相应的小图标可以进一步查看结果。
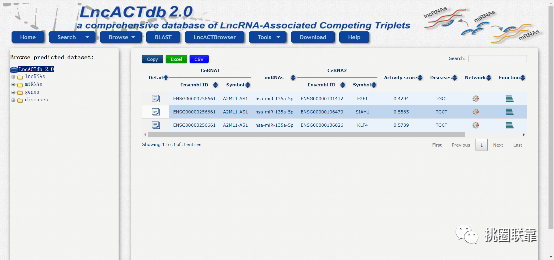
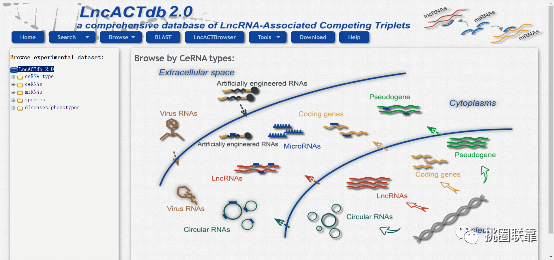
点击“Experimental data”,跳转到如下界面。用户可以在左侧列表或者直接点击右侧图案检索不同类型的RNA参与的ceRNA对。
4
“BLAST”功能
点击“BLAST”,用户可以通过在对话框中输入相应的lncRNA分子序列进行检索。此处我们输入MALAT1分子的序列,点击“BLAST”:
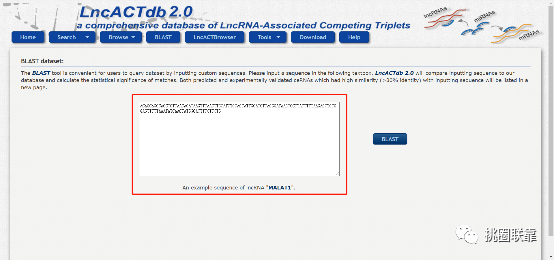
页面刷新后显示出Blast结果,提示该序列为MALAT1分子:
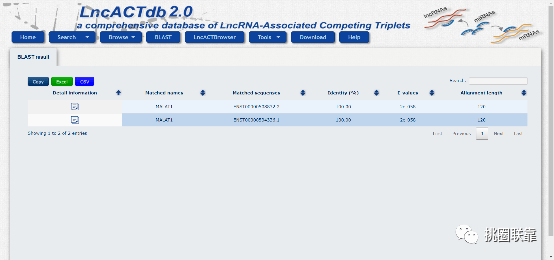
进一步点击“Detail information”下的图标可以显示MALAT1参与的ceRNA关系对:
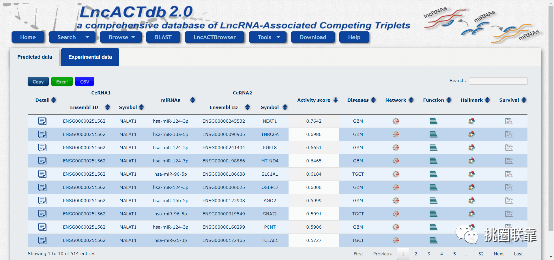
5
“LncACTBrowser”功能
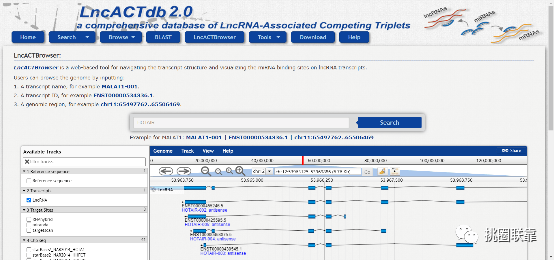
点击“LncACTBrowser”跳转到如下界面。用户可以通过在对话框中输入转录本名称,ID或染色体位置进行检索。
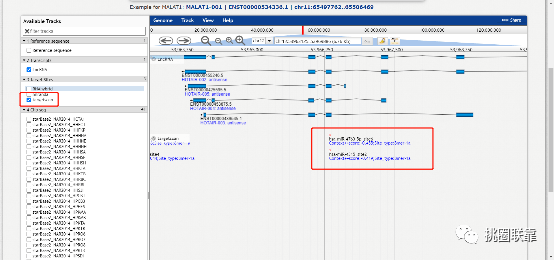
左侧在“Target Sites”中点击“targetscan”,右侧即可显示Targetscan数据库中收录的相应miRNA与lncRNA结合位点。同理点击“RNAhybrid”与“miranda”也可显示相应的结果。
6
“Tools”功能
点击“Tools”可以看到LncACTdb 2.0数据库提供了四个小工具,包括“Function”,“Survival”,“Network”,“Get”。前三个功能同上文介绍,不再赘述。个人推荐第四个小工具“LncACT-Get”对于大家处理全转录组的数据非常方便。
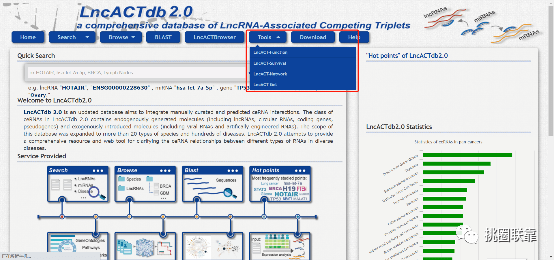
点击“LncACT-Get”,跳转到如下界面:
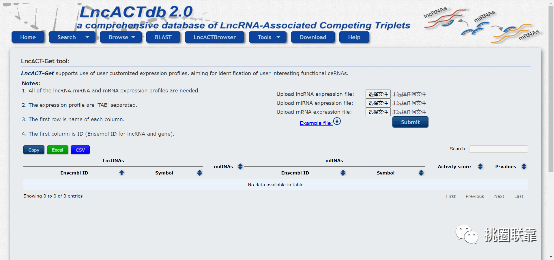
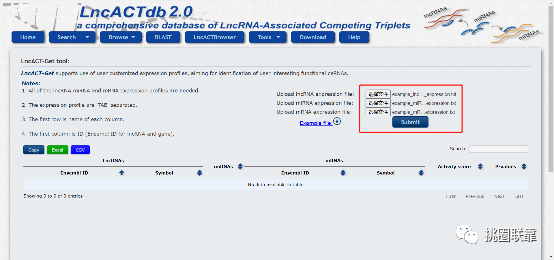
可以看到使用这个工具的话需要用户上传三个文件,包括lncRNA,miRNA与mRNA的表达矩阵。上传完毕后,数据库可以根据三种分子的表达矩阵计算相互之间的相关性,结合生信数据库的预测,进一步分析出潜在的ceRNA关系对,简直是全转录组数据分析的神器~
我们以示例数据为例,上传完毕后,点击“Submit”:
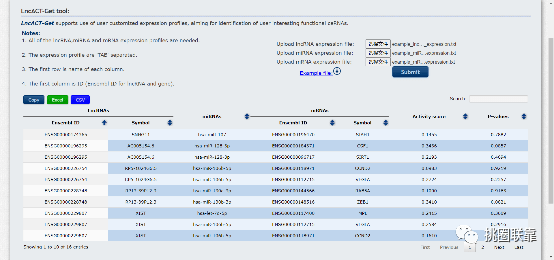
页面刷新后即显示出所有预测的ceRNA关系对。数据库自动计算相关性与生物信息学分析,超方便有木有!
好啦,关于lncACTdb
2.0数据库我们就介绍到这里啦~如前文所述,数据库整体上分为两个大的部分,包括预测和实验验证,但是从文献收录得到的实验验证部分仅截止到2018年,所以个人认为预测部分的功能对大家帮助最大。最后还是给“LncACT-Get”打个call,别看他蜷缩在数据库的小角落,但是对于潜在ceRNA关系对的挖掘属实过于方便,大家不要忽视哦~
转自:挑圈联靠
- 本文固定链接: https://maimengkong.com/zixun/1503.html
- 转载请注明: : 萌小白 2023年5月1日 于 卖萌控的博客 发表
- 百度已收录
