by 老马
上几期我们介绍了lncRNA的研究:1)什么鬼,lncRNA原来这么分析?2)实例解读|高分文章|lncRNA-miRNA作用于心脏肥大;3)实例解读| lncRNA在胃癌中的作用;4)最全长链非编码RNA数据库介绍(上);5)GWAS+lncRNA是否能成为后基因组研究的宠儿?反映不错!大家对lncRNA的研究很感兴趣。这一期小编继续为大家分享一篇lncRNA+miRNA+mRNA的RNA深度测序的好文[1]。题目是:Deep RNA sequencing reveals dynamic regulation of myocardial noncoding RNAs in Failing Human Heart and remodeling with mechanical circulatory support. Kai-Chein Yang et al. 2014. Circulation (IF=19.309)
大家都知道,基因芯片已经广泛用于对复杂疾病的研究,但其有限的基因组覆盖度难以得到详尽的转录组特征。采用二代深度测序能够很好的覆盖整个基因组。为了更好的更全面的研究心力衰竭以及辅助装置下重塑过程相关的心肌细胞lncRNA、miRNA和mRNA动态变化,本文主要采用转录组测序来研究心力衰竭。
在讲述主文之前,我们给大家稍微介绍一下何谓心力衰竭?心力衰竭(heart failure)简称心衰,是指由于心脏的收缩功能和(或)舒张功能发生障碍,不能将静脉回心血量充分排出心脏,导致静脉系统血液淤积,动脉系统血液灌注不足,从而引起心脏循环障碍症候群,此种障碍症候群集中表现为肺淤血、腔静脉淤血。心力衰竭并不是一个独立的疾病,而是心脏疾病发展的终末阶段。其中绝大多数的心力衰竭都是以左心衰竭开始的,即首先表现为肺循环淤血。
本文作者们分别对40个样本同时进行了lncRNA-seq、miRNA-seq和RNA-seq测序。样本包括:非缺血性心力衰竭植入辅助装置前后的左心室样本8对,缺血性心力衰竭植入辅助装置前后的左心室样本8对,正常心肌左心室样本8个。
文章的分析流程图如下:
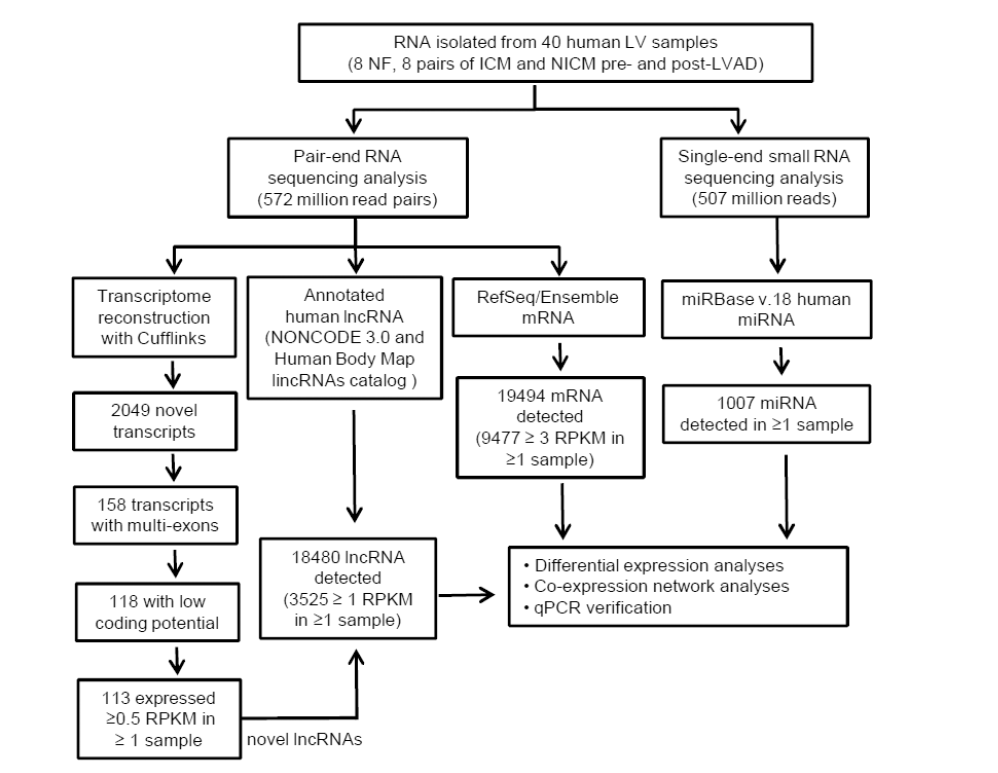
注解:NICM:非缺血性;ICM缺血性;NF:正常样本;pre-LVAD:植入辅助装置前;post-LVAD:植入辅助装置后。
结果分析:
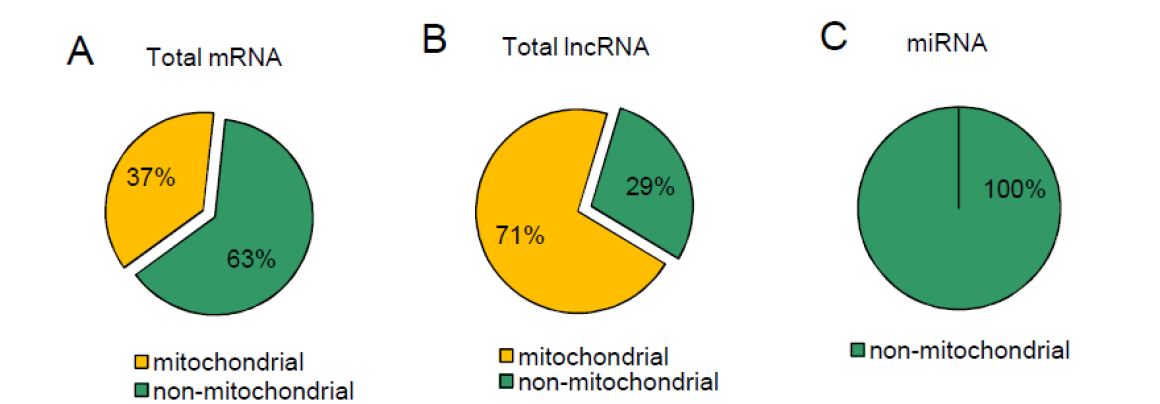
1. 转录组分析后发现,心脏的mRNA和lncRNA中分别有37%和71%比例的来自线粒体DNA。而miRNA则不同,所有miRNA都来自非线粒体。
2. 根据表达水平,作者们把转录分为3类,以mRNA转录为例,高表达(RPKM>3)的比例为40%;中等表达(RPKM,1-3)比例为18%;低表达(RPKM<1)比例为42%。同样,在lncRNA和miRNA也发现转录本中占绝大部分的是低表达和高表达的部分,特别是lncRNA低表达转录本的比例高达88%。

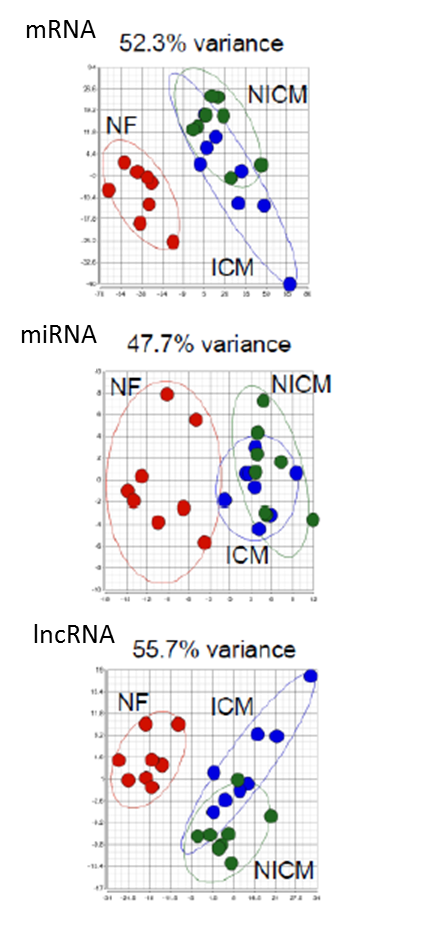
3. 作者将基因名字和PMMR/RPKM的比值输入到MultiExperiment Viewer软件中,用于做Heatmap图和hierarchical clustering test. 如下图,我们可以看出,mRNA、miRNA和lncRNA在心肌疾病中的表达模式与正常样本中显著不同。其中,lncRNA的表达还可以区分ICM和NICM病人。

进一步,作者利用Partek Genomics Suite软件进行主成分分析。同样,发现心肌病人的mRNA、miRNA和lncRNA表达异常不同于正常人。如下面主成分分析图:
同样,利用独立心肌样本,进行heatmap-unsupervised hierarchical clustering聚类分析lncRNA表达。作者们验证了lncRNA可以区分开ICM和NICM病人,如下图所示。总结起来,心肌转录组分析发现lncRNA能够明显区分心肌衰竭的不同类型,而且对植入辅助装置前后的变化比较灵敏。这里注意:此处验证用到的数据是从GEO公共数据(GSE1145)下载,是芯片数据。证据显示之前设计用于protein-coding gene 分析的芯片中约有10%~30%的probe实际上map to lncRNA. 这里利用Gatexplorer软件,作者们从传统的芯片中提取了5635个lncRNA用于验证分析。
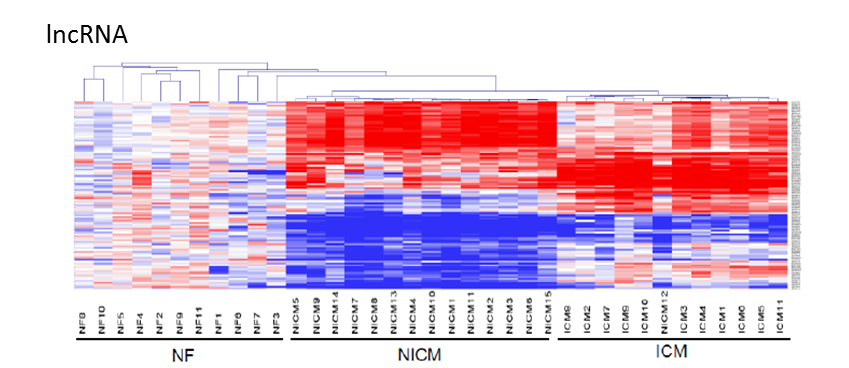
接着作者们分析了植入辅助装置(LVAD support)下的miRNA、mRNA和lncRNA的表达变化。植入辅助装置是一种将血液由静脉系统或心脏引出直接泵入动脉系统部分或全部代替心室做功的人工机械装置。

他们发现了许多差异表达的RNA,如在ICM和NICM病人中分别发现了160和147个差异表达的miRNA,2262和1929个差异表达的mRNA,679和570个差异表达的lncRNA。有趣的是,利用unsupervised hierarchical clustering方法对lncRNA表达谱进行聚类,作者们发现lncRNA的表达特征不仅可以区分正常样本和心肌症左心房的样本,而且可以判别LVAD治疗前后的样本,如下图。
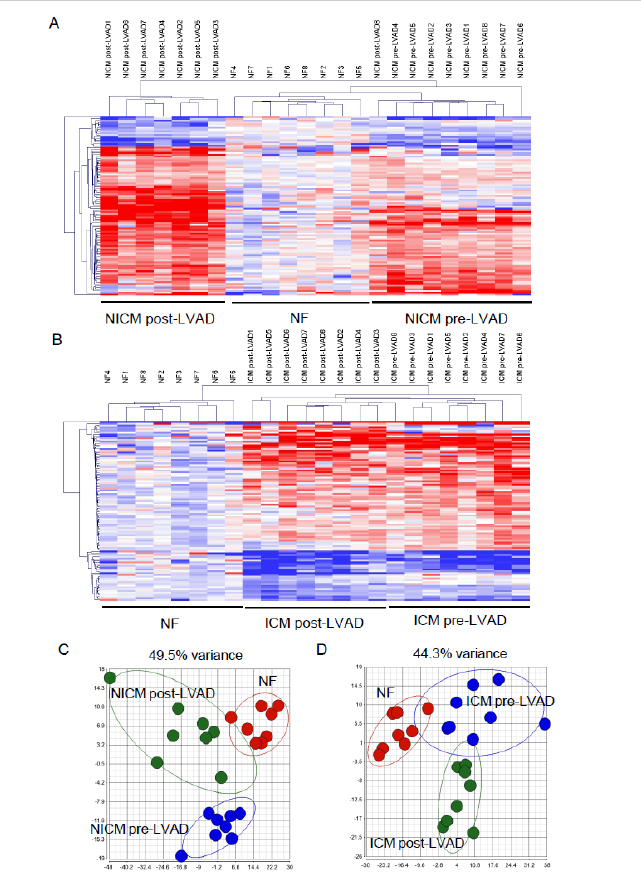
总结以上结果,可以看到相比于其它两种RNA, lncRNA的表达很大程度上参与心衰调节,可能作为很有用的生物标记来评估不同疾病状态。到这里,作者们阐述了lncRNA在心衰中的重要性,下面开始以lncRNA为主要视角展开研究。
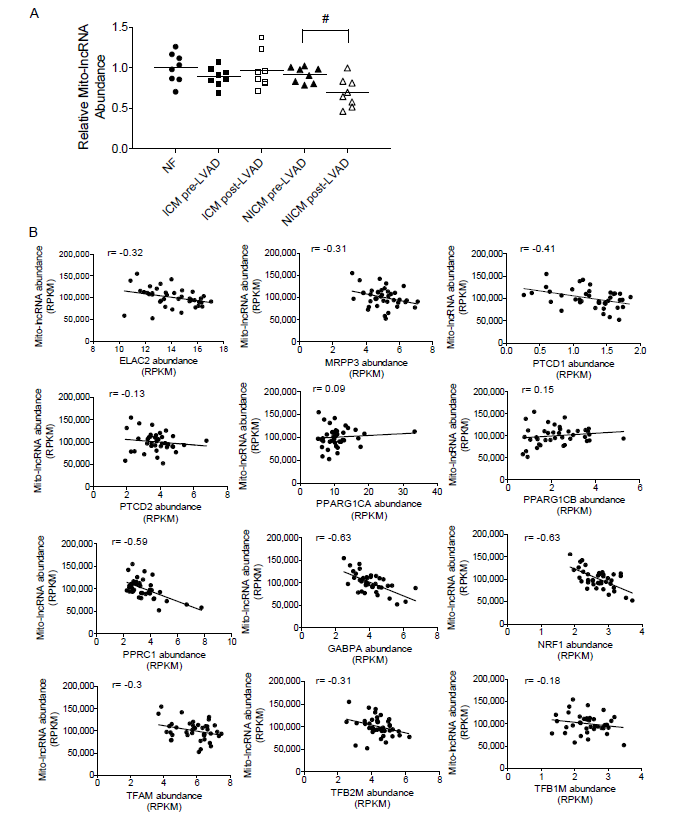
前面已经提到线粒体中也有很大一部分lncRNA (mito-lncRNA)。作者挑选了已知调节mito-lncRNA表达或者mito-gene转录的基因,包括ELAC2, MROO3, PTCD1, PTCD2, PPARG1CA, PPARG1CB, PPRC1, GABPA, NRF1, TFAM, TFB2M, TFB1M [2, 3],进行Pearson’s correlation分析。作者发现这些基因与mito-lncRNA都是负相关。
此前的研究已经发现,lncRNA能够调节邻近的mRNA/Coding genes (cis-mRNA)。利用Pearson’s correlation分析方法,作者计算lncRNA表达水平与cis-mRNA之间的相关系数,并与random gene pairs进行比较。作者们发现:random gene pairs之间没有显著相关性,但lncRNA与cis-mRNA是显著正相关。qRT-PCR验证了其中一些lncRNA-cis-mRNA的相关性。
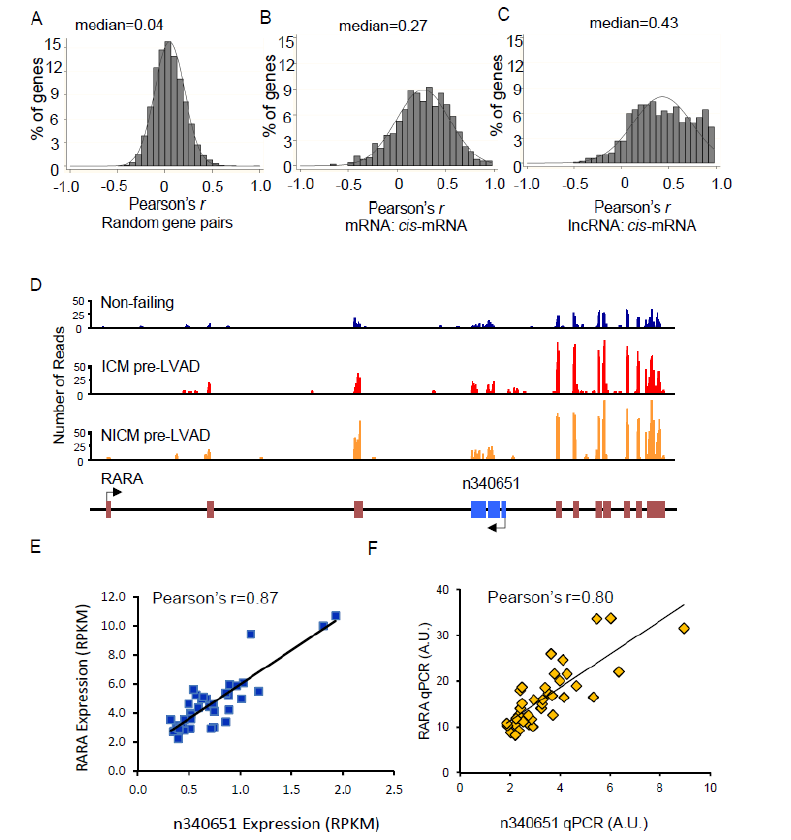
尽管许多lncRNA是cis-调节的功能(因此与邻近的编码基因有很多的相关性),但也发现一些lncRNA能够与远端的基因互作或参与调节,即具有trans-调节的功能(距离大于1Mb或在不同的染色体上)。本文为了探究lncRNA可能的trans-调节功能,对所用可以发现的lncRNA和mRNA进行了pairwise correlation分析。计算了约100万对lncRNA-mRNA和mRNA-mRNA的Pearson相关系数。结果发现在人类左心室细胞中lncRNA调节远端mRNA(lncRNA-mRNA)远没有trans-mRNA interactions程度高,如下图:
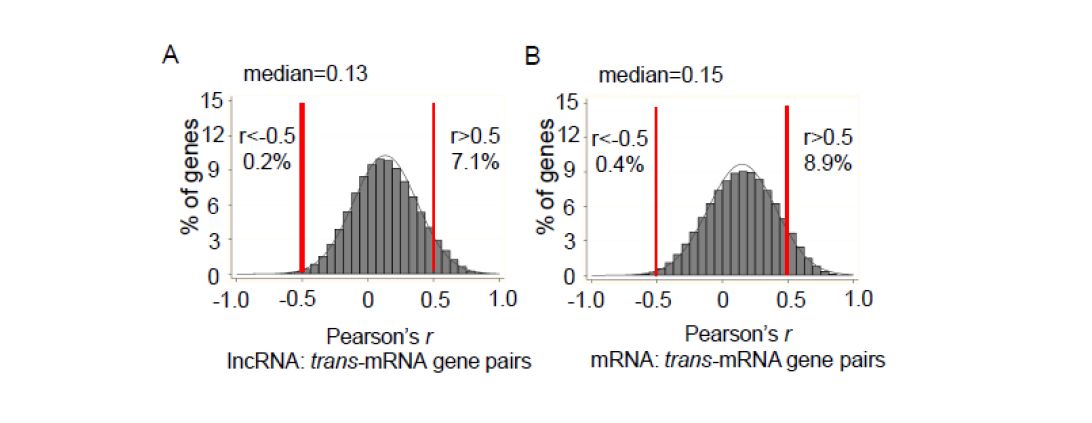
小结:本文进行了转录组测序发现lncRNA,miRNA和mRNA在心肌细胞中的表达情况。通过差异分析找到了许多与疾病相关的差异表达的RNA,并利用三种RNA进行聚类分析和主成分分析区分疾病。其中,lncRNA相比于其它两种RNA很大程度上参与心衰调节,可能作为有用的生物标记来评估不同疾病状态。同时作者又进行了cis-和trans-mRNA的相关性分析。全文思路清晰,所用到的方法比较简单常见,从整体水平探索lncRNA表达水平与心力衰竭疾病之间的关系。
参考文献:
1. Yang, K.-C., et al., Deep RNA sequencing reveals dynamic regulation of myocardial noncoding RNA in failing human heart and remodeling with mechanical circulatory support. Circulation, 2014: p. CIRCULATIONAHA. 113.003863.
2. Rackham, O., et al., Long noncoding RNAs are generated from the mitochondrial genome and regulated by nuclear-encoded proteins. RNA, 2011. 17(12): p. 2085-2093.
3. Hock, M.B. and A. Kralli, Tranional control of mitochondrial biogenesis and function. Annual review of physiology, 2009. 71: p. 177-203.
生信草堂
- 本文固定链接: https://maimengkong.com/moreshare/1124.html
- 转载请注明: : 萌小白 2022年7月15日 于 卖萌控的博客 发表
- 百度已收录
