想要做性状定位,
家系群体、自然群体傻傻分不清楚?
单性状、多性状无从下手?
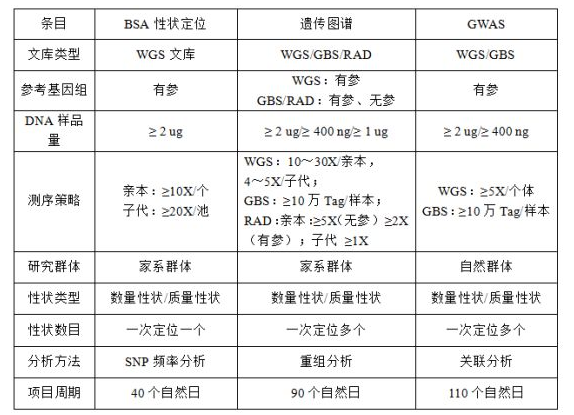
BSA、遗传图谱、GWAS不知该如何选择?
不要着急,今天小编为大家带来性状定位干货分享,教大家正确选择适合自己研究的性状定位方法。
►BSA性状定位(Bulk Segregate Analysis, BSA),指利用混池分组分析法,对家系群体目标性状表型极端的子代分别混合成两个基因池,同时对两个性状差异明显的亲本进行测序,检测与性状相关联的位点并注释,分析基因控制目标性状的机制。BSA是目前应用最广泛的一种单性状定位方法。
►适用范围:BSA用于定位具有极端表型的单性状,一般适用于质量性状单基因及数量性状主效基因的定位。
►BSA研究策略:选取极端表型性状亲本—构建群体—构建极端表型池—高通量测序—基于SNP-index进行关联分析—候选基因定位
文献案例一
BSA方法鉴定木豆抗枯萎病和花叶病基因[1]
◆ 研究背景:木豆(Cajanus cajan)是热带和亚热带区域发展中国家的一种重要的豆科粮食作物。但是,木豆的产量受到枯萎病(FW)和花叶病(SMD)的严重影响。因此,培育抗FW和SMD的木豆植株具有重要意义。
◆ 研究材料:亲本 ICPL 20096(抗)x ICPL 332 (易感)的构建RILs;取分离群体中极端表型的个体各16株构建抗性池(R)和易感池(S);
◆ 测序策略:Illumina Miseq平台;子代池约14 ×,亲本约15×。
◆ 研究结果:揭示了位于4个基因上的4个候选nsSNPs 与FW抗性相关,位于3个基因上的4个候选nsSNPs与SMD抗性相关。并且蛋白分析和表达谱分析鉴定出2个最可能的候选基因。
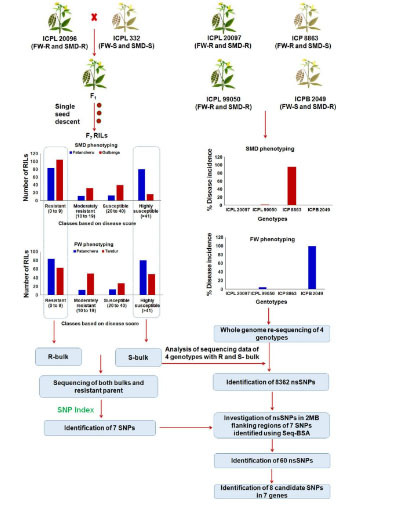
图1. 木豆BSA鉴定抗枯萎病和花叶病基因技术路线图
►遗传连锁图谱(Genetic Linkage Map),指基因或其他DNA序列在染色体上的相对位置与遗传距离。通常以基因或DNA片段在染色体交换过程中分离的频率厘摩(cM)来表示。其最重要的应用是鉴定目的基因或QTL在染色体上的位置。遗传连锁图谱是最经典的性状定位方法。
►适用范围:
➀ 遗传图谱主要用于数量性状位点的定位,获得与目标性状连锁的基因组片段 。
➁ 其次,遗传图谱可用于辅助动植物基因组的组装,提高基因组图谱的完整性。
►遗传连锁图谱研究策略: 选配亲本—构建家系分离群体—高通量测序—开发和筛选标记—基于子代重组事件的连锁分析—QTL定位
文献案例二
GBS测序构建小麦遗传图谱及三雌蕊基因的定位[2]
◆ 研究背景:利用GBS测序技术构建F2代的遗传图谱,定位三雌蕊基因(Pis1),为 Pis1 基因的克隆奠定基础,为研究小麦重要性状和标记辅助育种提供重要工具。
◆ 研究材料:亲本川麦CM28 × CM28TP 构建的200份F2代分离群体。
◆ 测序策略:Illumina HiSeq2000 进行GBS测序。
◆ 研究结果:构建了含有2917个GBS-SNP标记的小麦高密度遗传图谱,总长度为2371.40cM,平均标记密度为0.81cM。成功绘制了 Pis1 基因,鉴定了与该基因紧密连锁的两个侧翼SNP标记M70和M71。
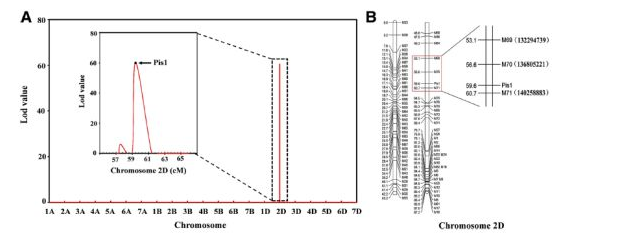
图2. Pis1 基因在小麦2D染色体上精细位置图谱
►全基因组关联分析(Genome-Wide Association Study, GWAS),是对遗传多样性丰富的群体中的每个个体进行全基因组重测序,结合目标性状的表型数据,基于一定的统计方法进行全基因组关联分析,可以快速获得影响目标性状表型变异的染色体区段或基因位点。GWAS关联分析是当前最热门的性状定位方法。
►适用范围:GWAS(全基因组关联分析)一般应用于自然群体上的全基因组范围内多性状定位及目标性状的生物学基础研究。在动植物中广泛应用,如:牛、羊、猪、鸡等家畜;水稻、玉米、大豆、高粱、番茄、辣椒等经济作物;在水产类动物、林木类植物及拟南芥、果蝇等模式生物中也有应用。
►GWAS研究策略: 群体选择—表型数据的鉴定—高通量测序—标记与表型关联分析—性状定位—基因注释
文献案例三
GWAS解析大豆复杂农艺性状间遗传网络[3]
◆ 研究背景:大豆是人类和动物油脂及蛋白质的主要来源,现代栽培大豆大约在5000年前由野生大豆驯化,继而进一步选育而成。大豆的产量、品质等大都是多基因控制的复杂性状,因此解析复杂性状间耦合的遗传调控网络,明确关键调控单元,对分子设计育种具有重要意义。
◆ 研究材料:809份大豆品种,表型收集于两年(2013-2014、2014-2015)和三个环境(周口、北京和牡丹江)84个性状的数据。
◆ 测序策略:Illunina HiSeq 2000/HiSeq 2500平台 平均测序深度8.3×/个体
◆ 研究结果:鉴定出245个显著关联位点,其中95个关联位点和其它位点存在上位性效应;这些关联位点很好地解释了单个性状的遗传调控,同时形成复杂的多性状多位点调控网络,揭示不同性状间相互耦合的遗传基础。
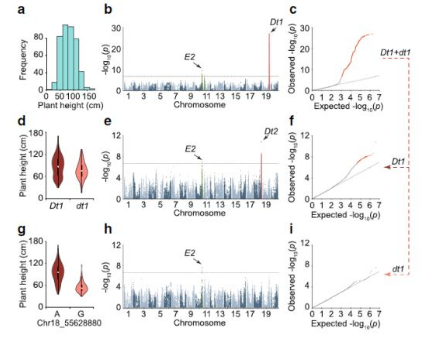
图3. 大豆株高的GWAS分析
参考文献:
[1] Singh V K, Khan A W, Saxena R K, et al. Next-generation sequencing for identification of candidate genes for Fusarium wilt and sterility mosaic disease in pigeonpea (Cajanus cajan)[J]. Plant Biotechnology Journal, 2016, 14(5):1183-1194.
[2] Yang Z, Chen Z, Peng Z, et al. Development of a high-density linkage map and mapping of the three-pistil gene (Pis1) in wheat using GBS markers[J]. Bmc Genomics, 2017, 18(1):567.
[3] Fang C, Ma Y, Wu S, et al. Genome-wide association studies dissect the genetic networks underlying agronomical traits in soybean.[J]. Genome Biology, 2017, 18(1):161.
- 本文固定链接: https://maimengkong.com/morejc/1217.html
- 转载请注明: : 萌小白 2022年10月2日 于 卖萌控的博客 发表
- 百度已收录
