之前跟大家分享了《转录组测序结题报告怎么看?点进来就知道啦!》,看懂结果是解读数据的第一步,如何把结果跟最终的课题相关联,即数据的挖掘是数据到文章的转化关键,咱们今天就来聊聊如何在转录组结果中挖掘关键基因。
有参转录组
有参考基因组的物种进行转录组研究,因为基因的信息是已知的,所以在挖掘基因上会相对简单。
1、通过参考文献和前期实验获得了一些预期基因的信息,可以直接在转录组的差异分析结果中看基因的表达变化,是否为差异基因。文章思路参考《BmSUC1调节家蚕的糖代谢途径》。
———是,直接整理差异基因的GO和KEGG结果,明确目标基因通过什么功能和代谢通路发挥作用,富集在相同功能和通路上的其他基因以及在聚类分析中表达模式跟目的基因相近的其他基因也可以一起研究。
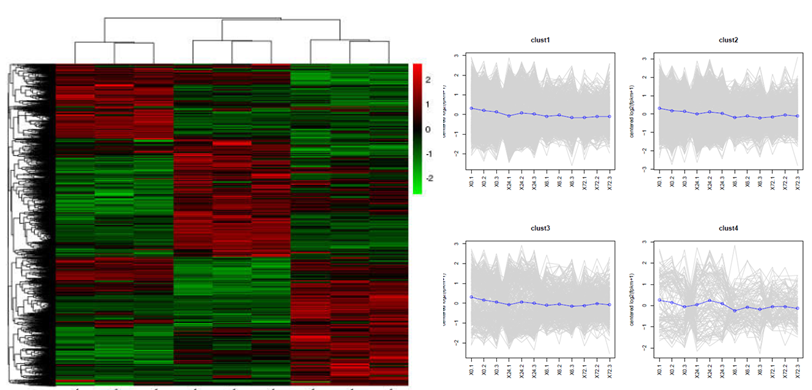
———否,看差异分析的标准是否需要调整,(调整下面的DESeq表格中的foldchange值),把目的基因扩充进来,之后参照差异基因的整理方法即可;如果目的基因几乎没有变化,可以通过寻找目的基因的同源基因,关联基因等信息来扩充研究的范围,或者通过现有的差异分析的富集的结果,看富集的功能和代谢通路是否可以跟目的基因相关联。
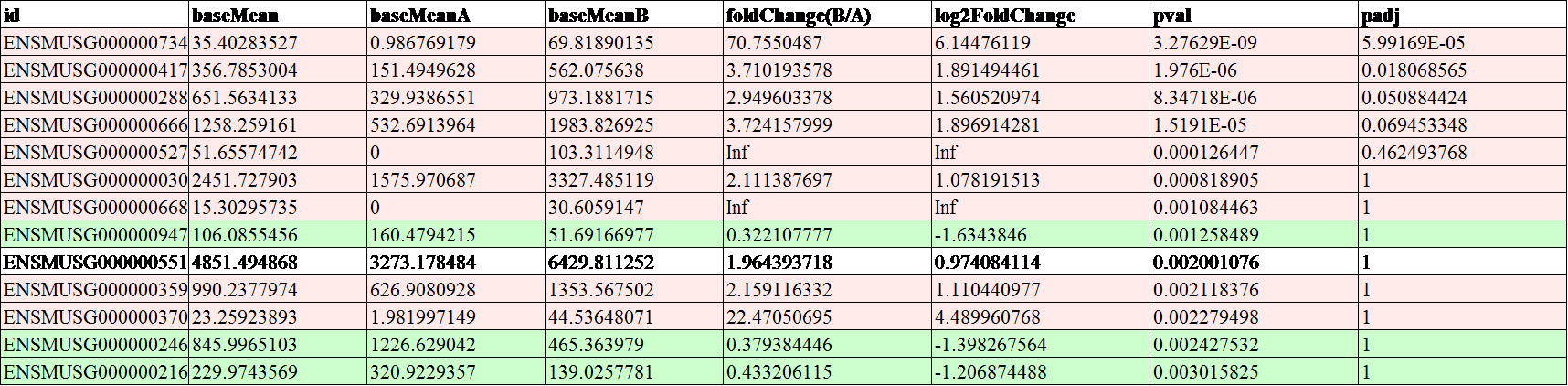
2、前期并没有什么目的基因,想要通过转录组的结果来看对照和处理的可能机制,那么直接整理项目的差异分析结果,说明差异基因的上下调情况,和具体的显著富集的功能和代谢通路即可;如果想要在转录组结果中筛选一些关键的基因,则需要通过GO和KEGG的富集分析结果,筛选显著差异的条目进行分析,来缩小研究的差异基因的范围。例如下表中p值<0.05的GO显著富集的条目有三条,那么这三个条目上富集的基因可以重点关注。文章思路参考《肉质品冷藏变质是谁在捣鬼?气调包装为啥能延长保鲜期?》。

无参转录组
无参考基因组的物种进行转录组研究,因为基因的信息是未知的,所以在挖掘基因上会相对有参的物种麻烦一些。建议可以在无参物种的注释结果中添加近缘有参物种的同源基因信息或者模式物种的同源基因信息,便于后续的基因查找。
1、通过参考文献和前期实验获得了一些预期基因的信息或功能,在无参的结果中如何获得目的信息:
——在unigene的总的注释表格中通过关键词进行查找,可以直接获得注释为该目的基因的unigene;在注释结果的GO和KEGG整理信息中获得目的功能和通路的信息,进而获得目的unigene;
——通过目的基因或蛋白的序列信息blast查找unigene中的同源基因,获得目的 unigene;
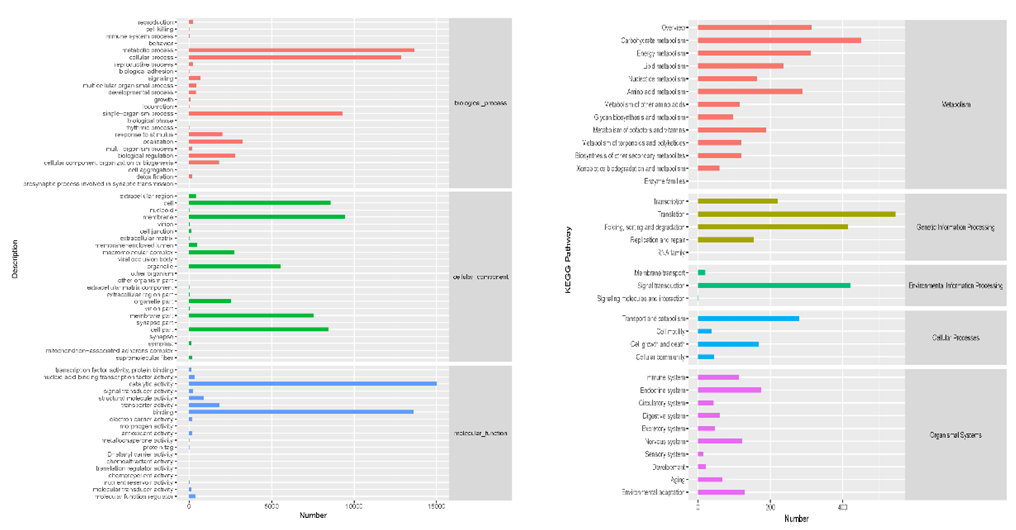
对于获得的目的unigene,可以在差异分析的结果中查看是否属于差异基因,之后的分析参考有参基因组的关键基因查找的思路即可。
2、前期没有特殊的目的基因,想要通过转录组的结果来看对照和处理的可能机制,这种情况跟有参的项目这种情况类似,直接整理项目的差异分析结果,说明差异基因的上下调情况,和具体的显著富集的功能和代谢通路即可;如果想要在转录组结果中筛选一些关键的基因,则需要通过GO和KEGG的富集分析结果,筛选跟研究目的相关的条目进行分析,来缩小研究的差异基因的范围。文章思路参考《RNA-Seq+qRTPCR,转录组文章轻松发》。
- 本文固定链接: https://maimengkong.com/kyjc/884.html
- 转载请注明: : 萌小白 2022年4月30日 于 卖萌控的博客 发表
- 百度已收录
