基因序列到进化树:轻松掌握多物种DNA序列比对与进化分析
基因序列的多物种比对与进化分析是研究物种间遗传关系和功能保守性等的关键步骤。今天,我们将以T2R41基因为例,手把手教大家如何下载基因序列、进行多序列比对,以及构建分子进化树。
1. 搜索基因并下载序列
首先,打开NCBI官网[1],在搜索栏输入目标基因名称T2R41。

进入搜索结果页面后,选择Gene类型,可以看到该基因在13个物种中有记录。
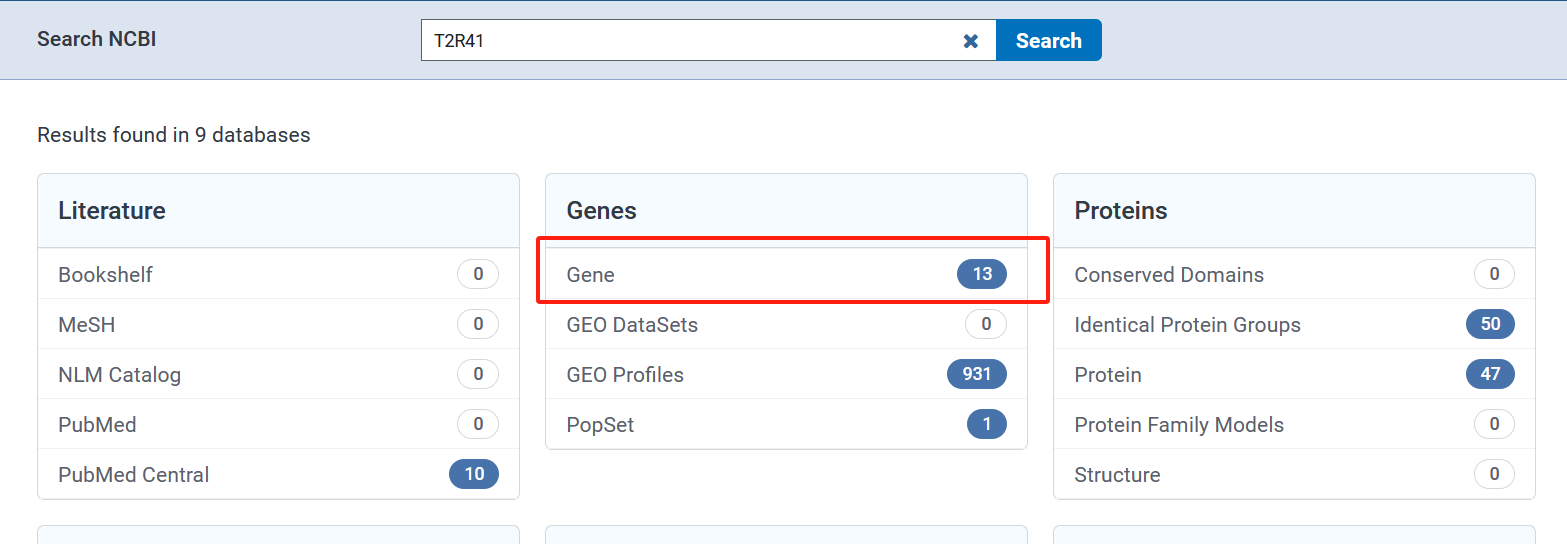
在检索到的Gene结果表格中,选择感兴趣的物种分别点击链接进行下载。
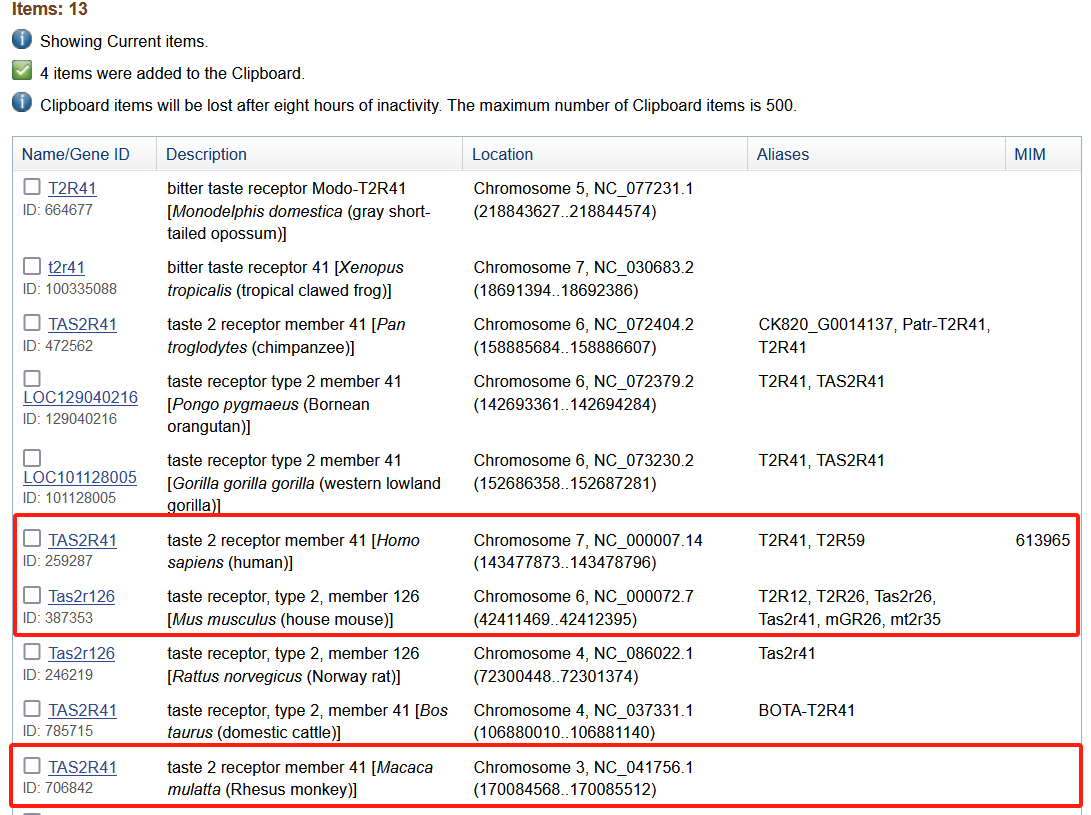
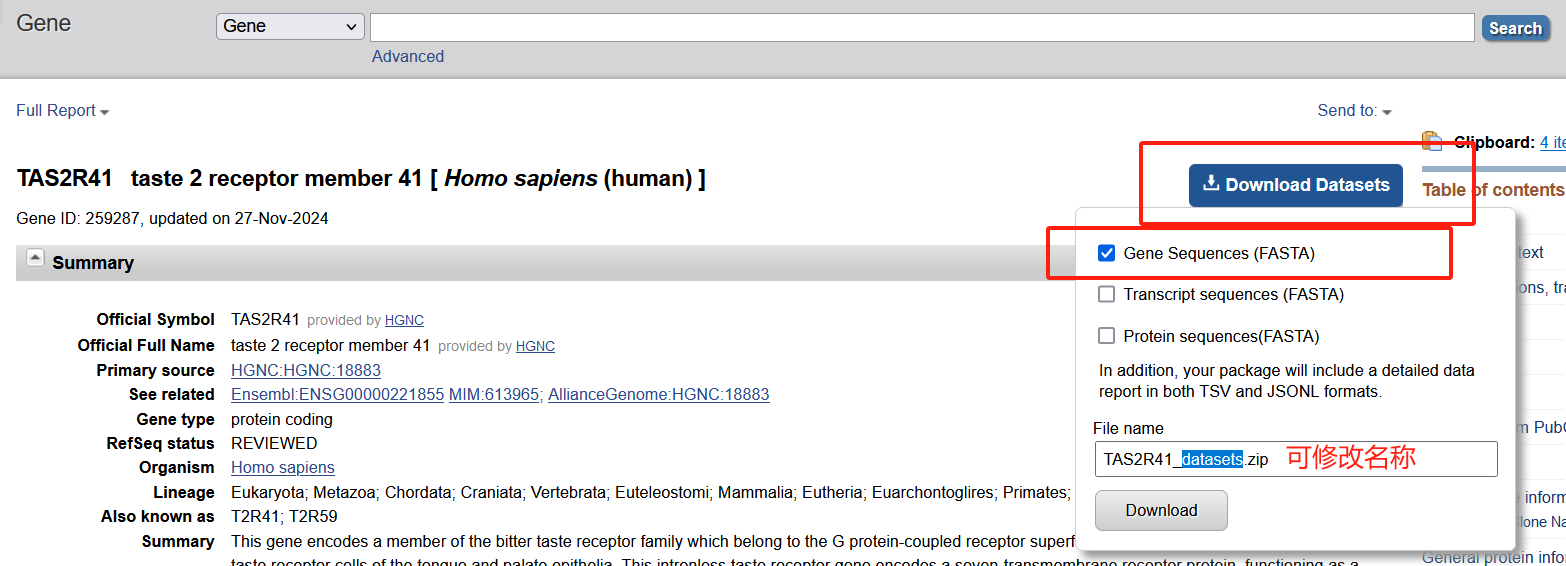
我们这里以human(人类)、mouse(小鼠)、monkey(猴子)为例,进入基因页面并点击右上角的Download Datasets按钮,选择Gene Sequence (FASTA)格式下载基因序列。建议将下载文件重命名为易识别的名称,方便后续整理。
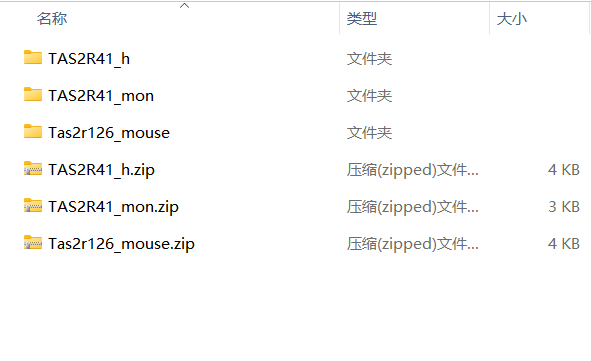
下载得到序列后,我们分别将其解压到各自文件夹。
2. 整理序列数据
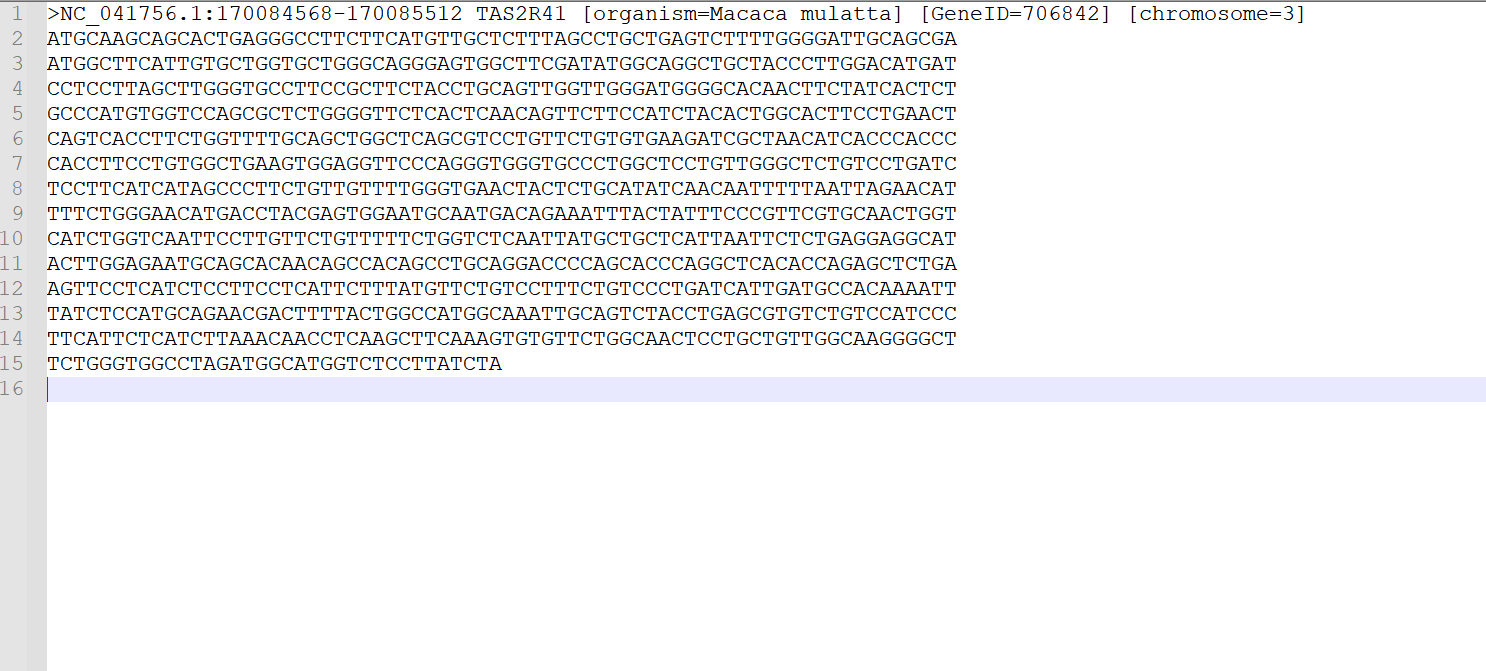
下载得到的压缩文件解压后,基因序列会存储在gene.fna文件中,如下图。
将各个物种的序列文件整理到一个总文件中,我们在此创建一个新的All.fa文件.。打开记事本,将每个gene.fna文件中的内容复制粘贴到All.fa,并对>后的描述信息进行简化,如下图:

3. 多序列比对
我们打开MEGA软件,将刚刚整理好的All.fa文件拖入软件。
拖入后会询问是否是比对或对比对结果分析,因为我们这是原始数据,所以需要先选择Align进行比对,点击后序列被自动导入比对窗口。
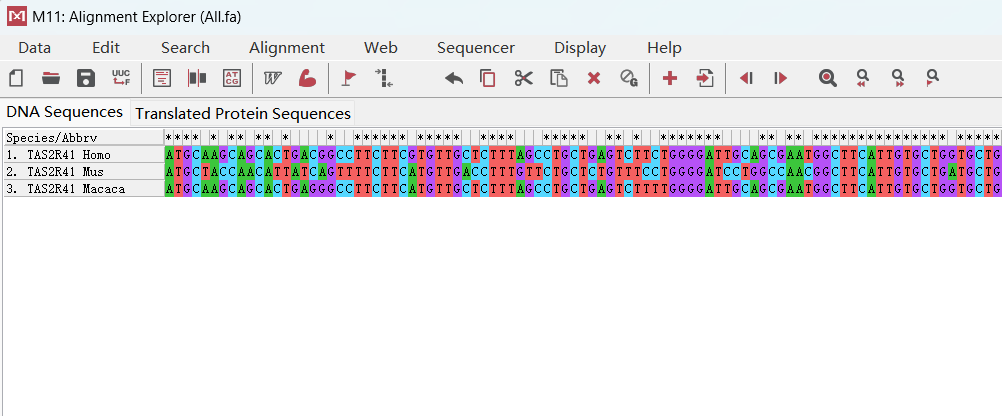
目前展示的序列并没有被对齐,因此我们选择Alignment菜单中的 Align by ClustalW进行比对。
Tips: ClustalW和MUSCLE都是比对序列的算法,可以相互替换,都可以尝试。
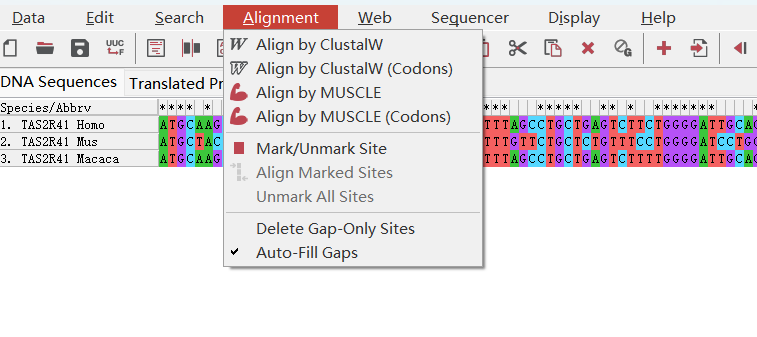
点击后,会跳出窗口,选择OK,全部序列进行比对。
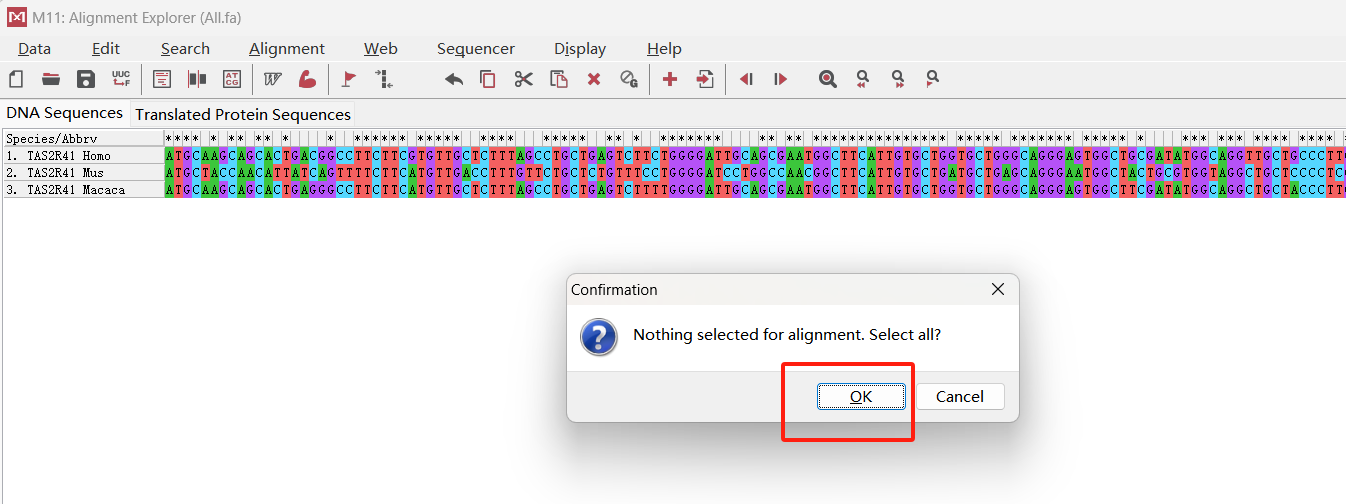
接着弹出参数设置对话框,参数默认即可。
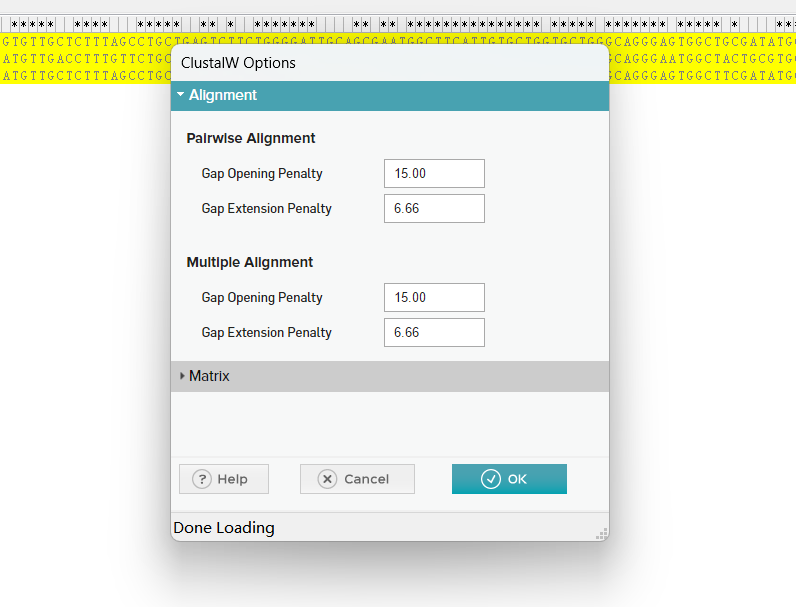
等待数秒后,接着我们会发现序列经过比对,已经被对齐。
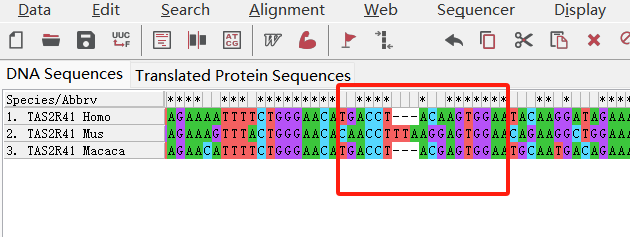
比对结果中,不同的背景颜色代表不同的碱基,最上方有*号的代表这个位置的碱基在物种间保守。
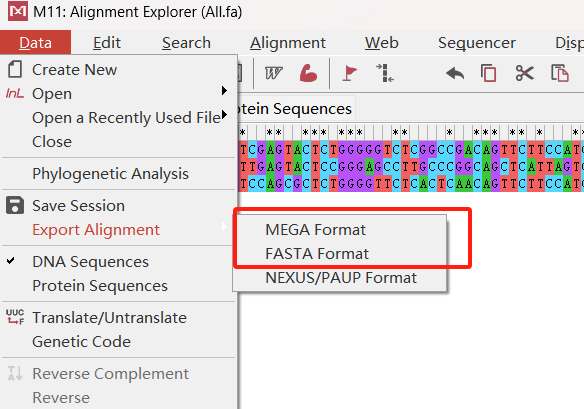
在Data菜单中,我们可以选择Export Alignment中的MEGA Format或FASTA Format导出,如果后续分析仅计划在MEGA软件则导出第一种MEGA Format,如果后续还打算导入结果到其他软件推荐FASTA Format。
4. 构建分子进化树
接着我们返回软件主页面,点击DATA图标打开刚才生成的结果,导入刚才的结果。
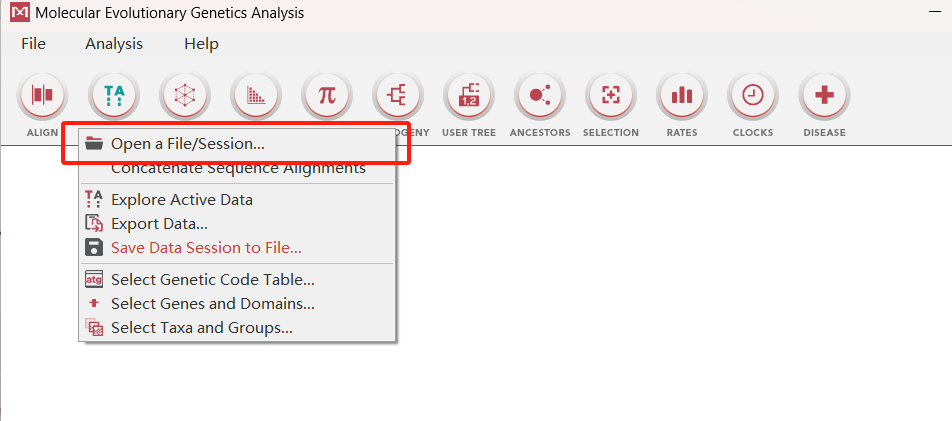
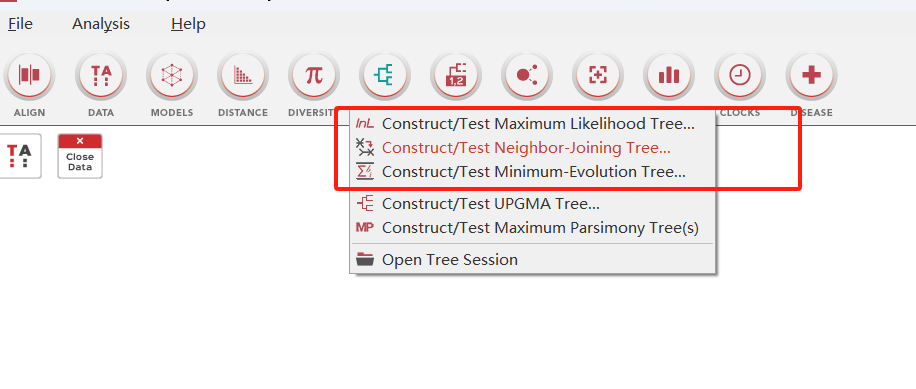
接着选择PHYLOGENY > Constract/Test Neighbor-Joining Tree,使用邻接法(NJ)构建进化树。
Tips:进化树构建有三种方法:若有合适的分子进化模型可供选择,则最大似然法获得的结果较好;对于近缘物种序列,通常情况下使用最大简约法;而对于远缘物种序列,一般使用邻接法或最大似然法。在这里我们选择邻接法进行展示。
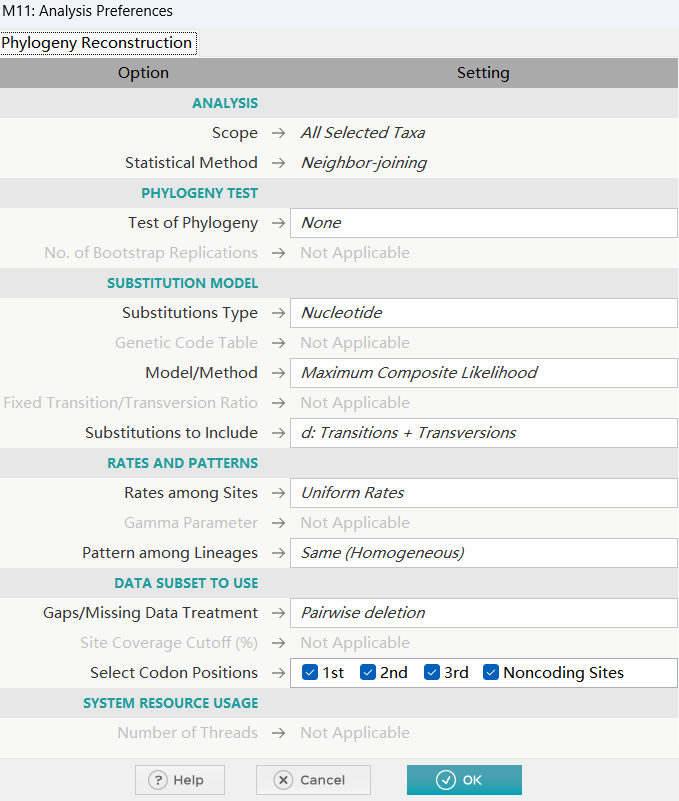
接着进入分析选项窗口,参数默认,选择OK即可
接下来我们就得到了分子进化树,如下图,我们可以在左侧工具栏调整效果,如文字大小,进化树长度等设置。
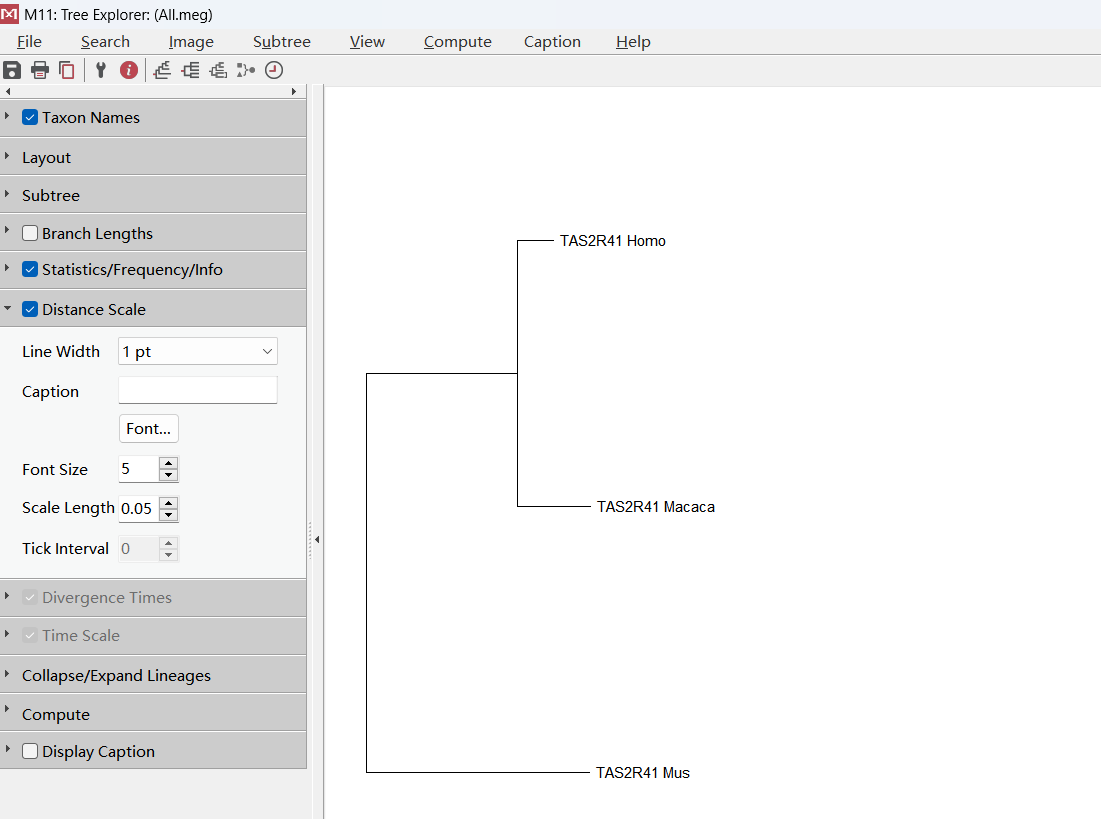
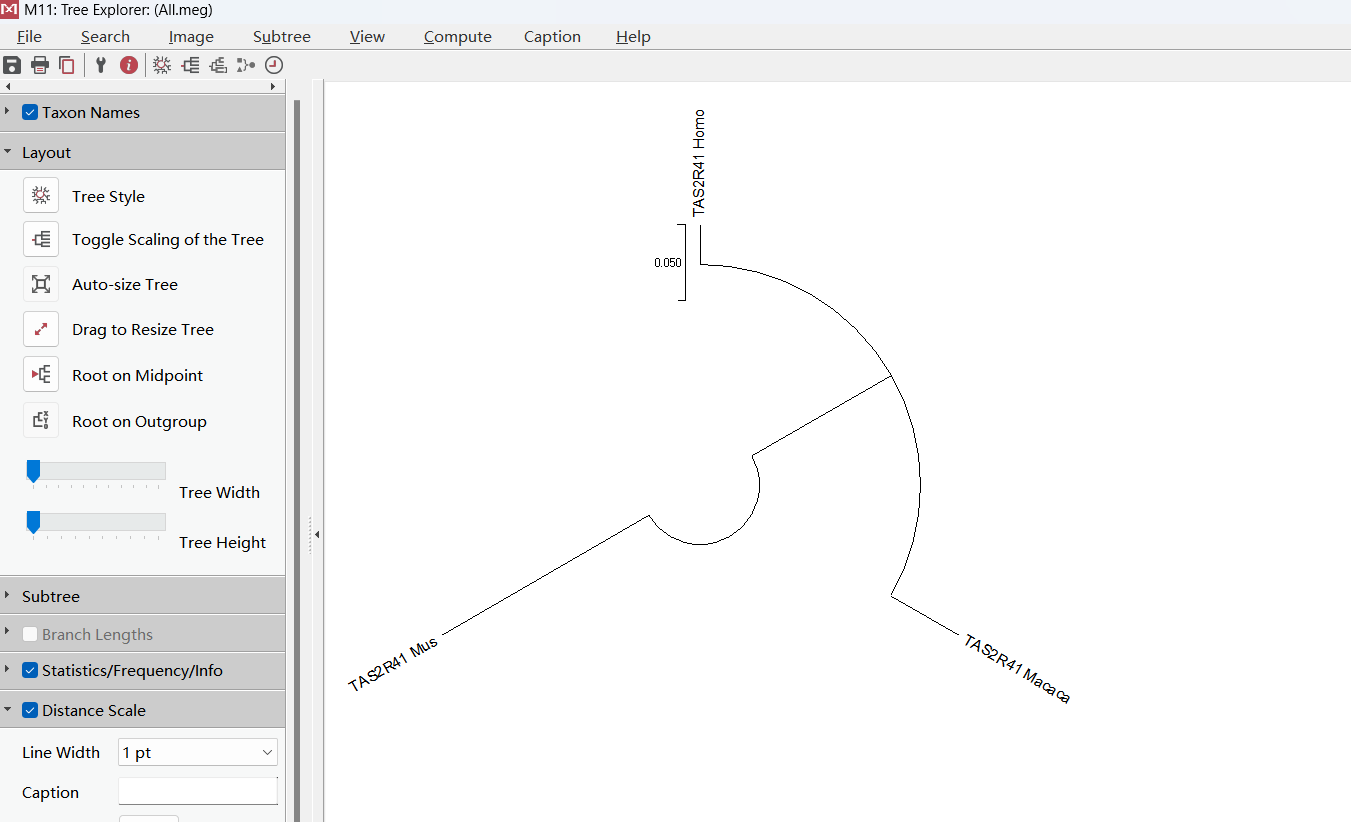
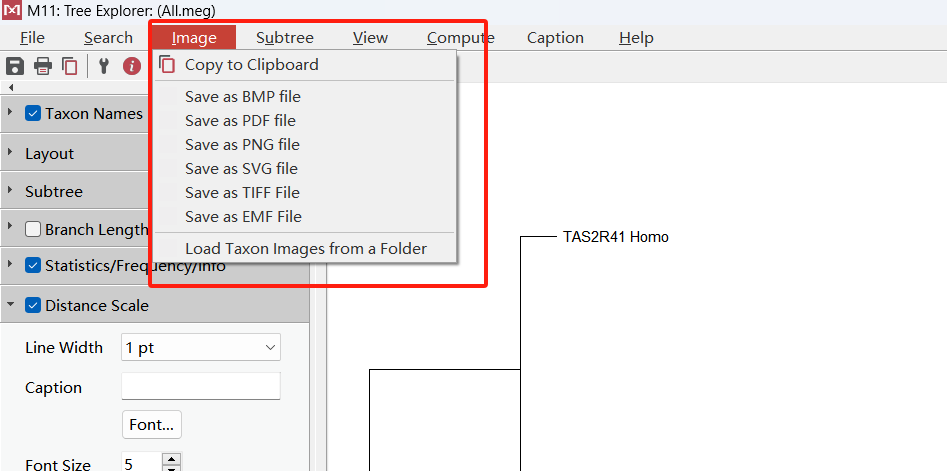
图片美化好后,可以选择Image菜单进行图片的导出。
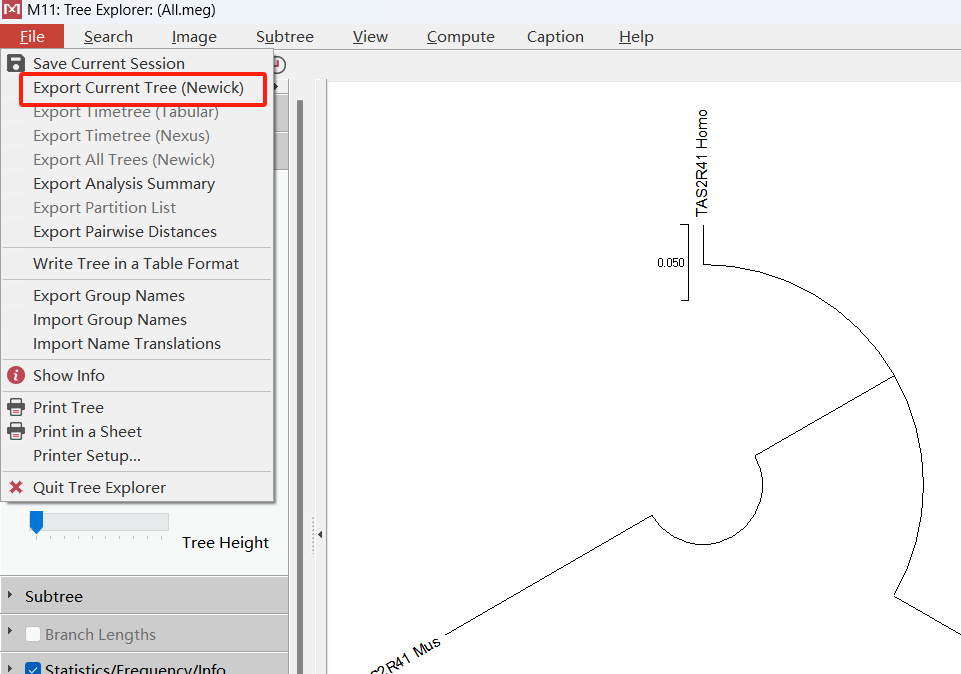
5. 导出nwk文件
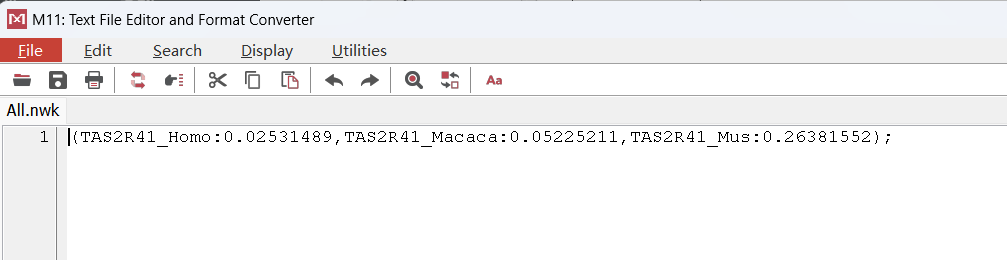
此外,可以将进化树导出nwk文件,点击File中的Export Current Tree(Newick)导出结果,进一步在其它工具中进行美化,如iTOL等网站。
Tips: nwk格式文件内容如下:
总结
通过以上步骤,完成了基因多物种比对与分子进化树的构建。从序列下载到比对分析,再到进化树的生成与美化,这些步骤展示了DNA序列比对的基本流程。希望这篇教程能帮助大家快速上手DNA多序列比对及基因进化分析,探索更多基因奥秘!
References
[1] NCBI官网: https://www.ncbi.nlm.nih.gov/
- 本文固定链接: https://maimengkong.com/kyjc/1840.html
- 转载请注明: : 萌小白 2024年11月20日 于 卖萌控的博客 发表
- 百度已收录
