Structure是由斯坦福大学Pritchard实验室开发的一款群体结构分析软件。structure可通过贝叶斯聚类方法对每个样本的来源进行判断,进而反应群体的遗传结构。真正跑时burnin设为100000,跑100000代,k设为1-20,每轮运行10次。
序列文件用xmfa2struct转为structure可识别的格式,SSR用Genalex转为structure可识别的格式。
操作步骤
打开structure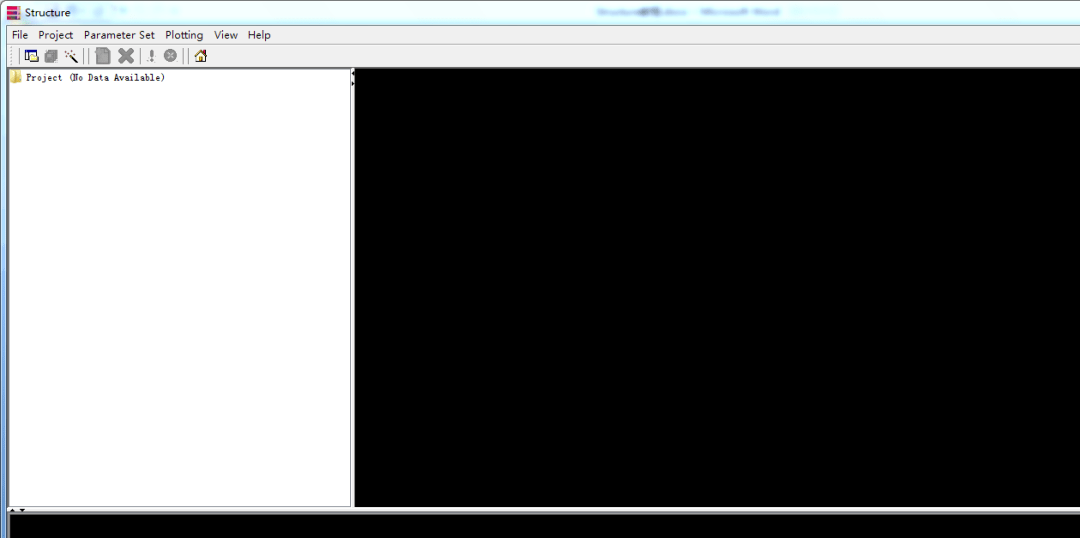 选择新建任务
选择新建任务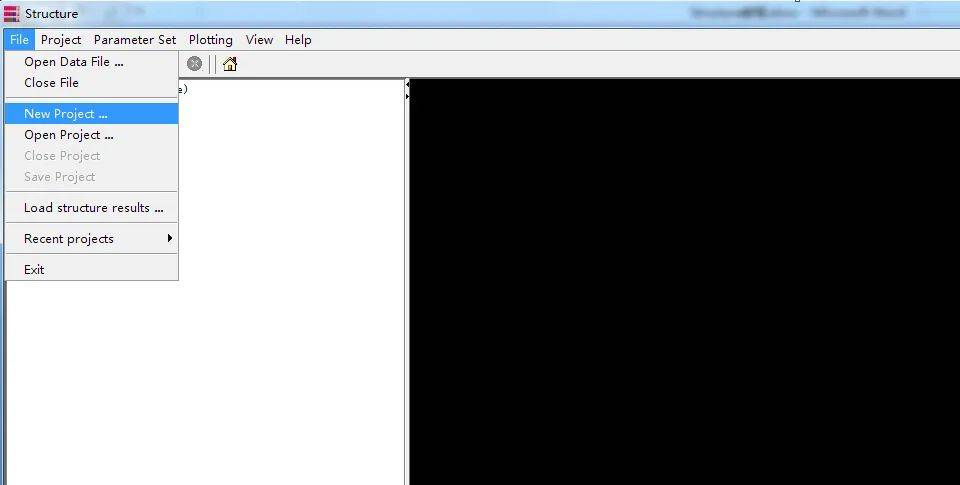 输入任务名,保存路径与输入文件
输入任务名,保存路径与输入文件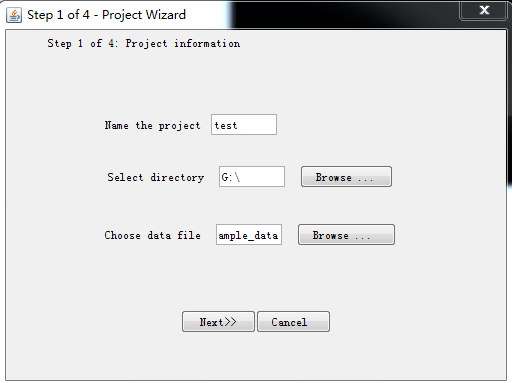 输入个体数,倍性,位点数,missing data的比重(一般都是-9)
输入个体数,倍性,位点数,missing data的比重(一般都是-9)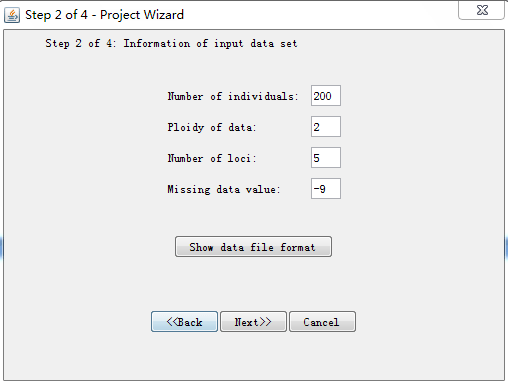 输入除了数据外多余的行的内容
输入除了数据外多余的行的内容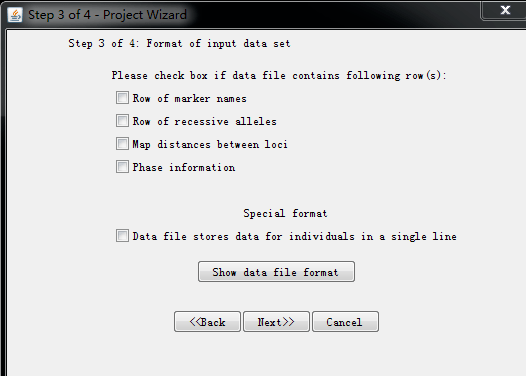 输入除了数据外多余的列代表的内容,点击finish
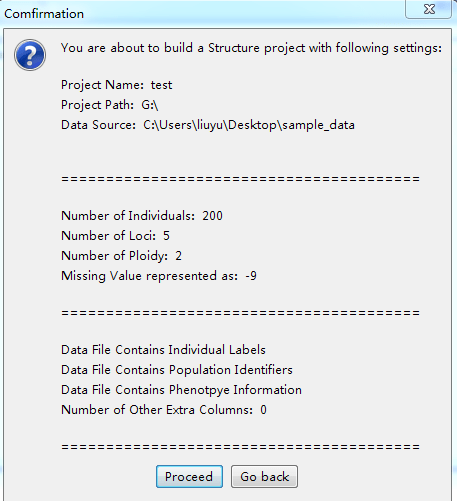
输入除了数据外多余的列代表的内容,点击finish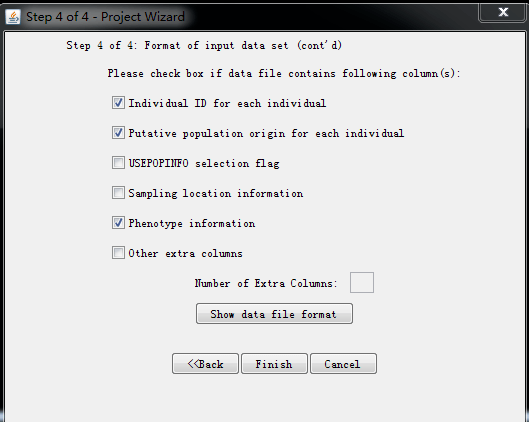 确认信息,点击proceed
确认信息,点击proceed
点击parameter set下的New 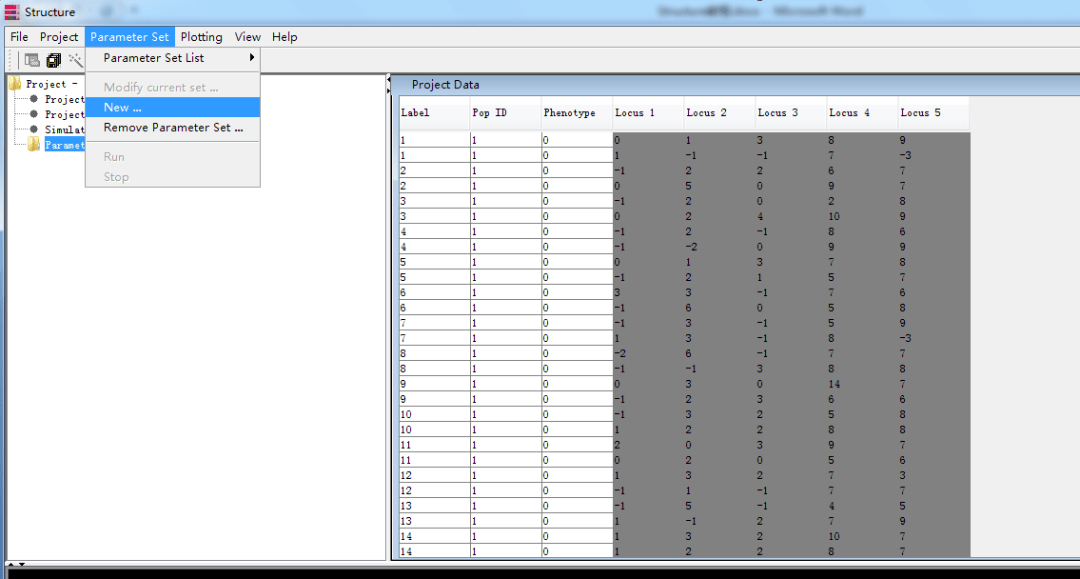 输入burnin多少代和burnin后再跑多少代,可以是5000与50000,其他模型之类的按照默认,点击OK
输入burnin多少代和burnin后再跑多少代,可以是5000与50000,其他模型之类的按照默认,点击OK 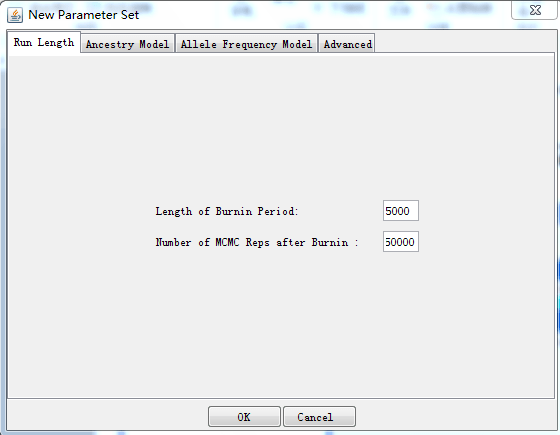
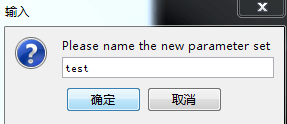
输入配置名,点确定
点击start a job 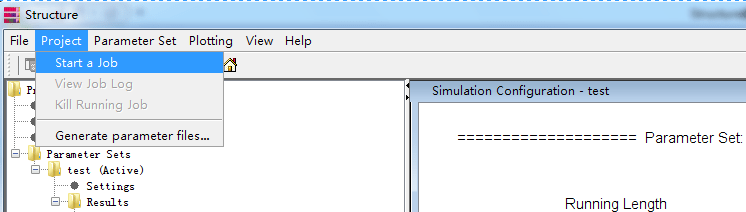 设定k为1到10,number of iterations为20次
设定k为1到10,number of iterations为20次 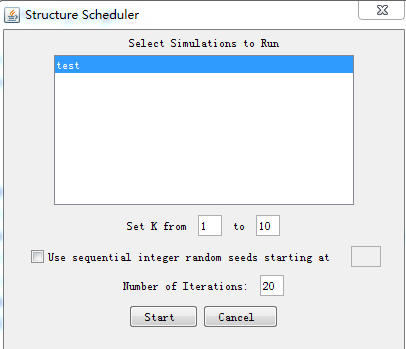
点击开始跑,等待跑完 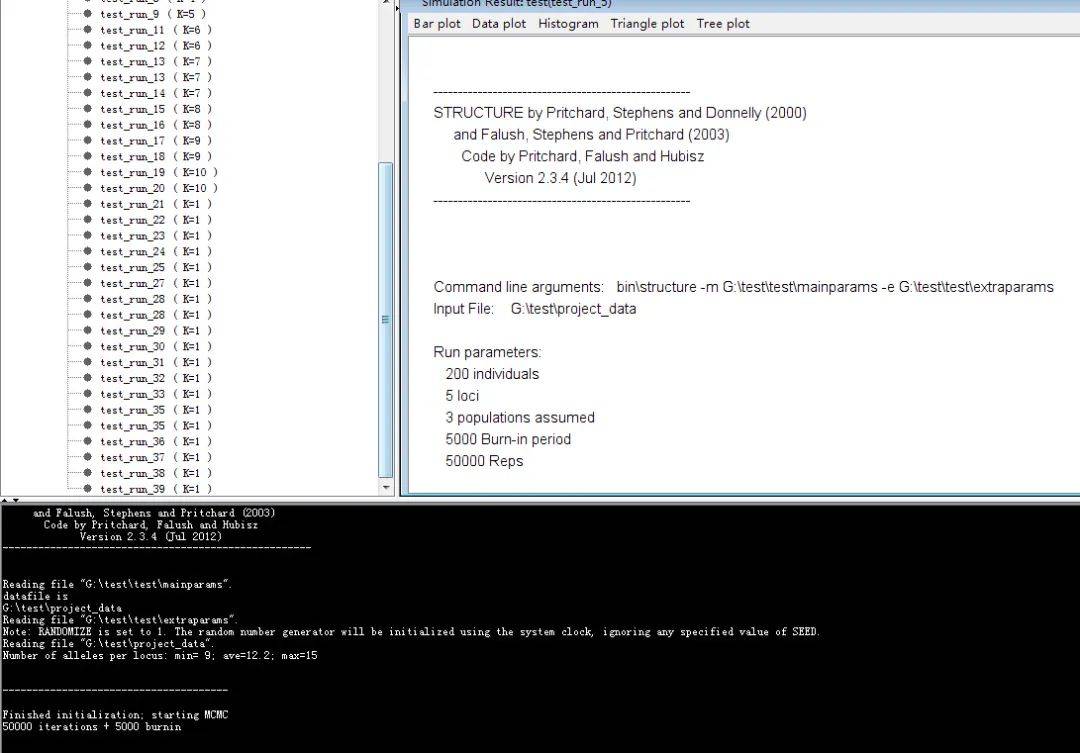 找到结果的目录,找到results文件夹,压缩为zip文件
找到结果的目录,找到results文件夹,压缩为zip文件 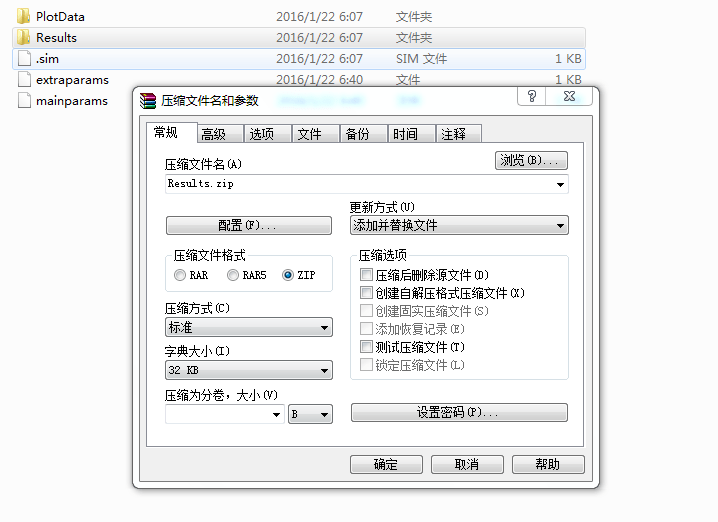 进入网站 http://taylor0.biology.ucla.edu/structureHarvester/# 选则results.zip,点击上传
进入网站 http://taylor0.biology.ucla.edu/structureHarvester/# 选则results.zip,点击上传 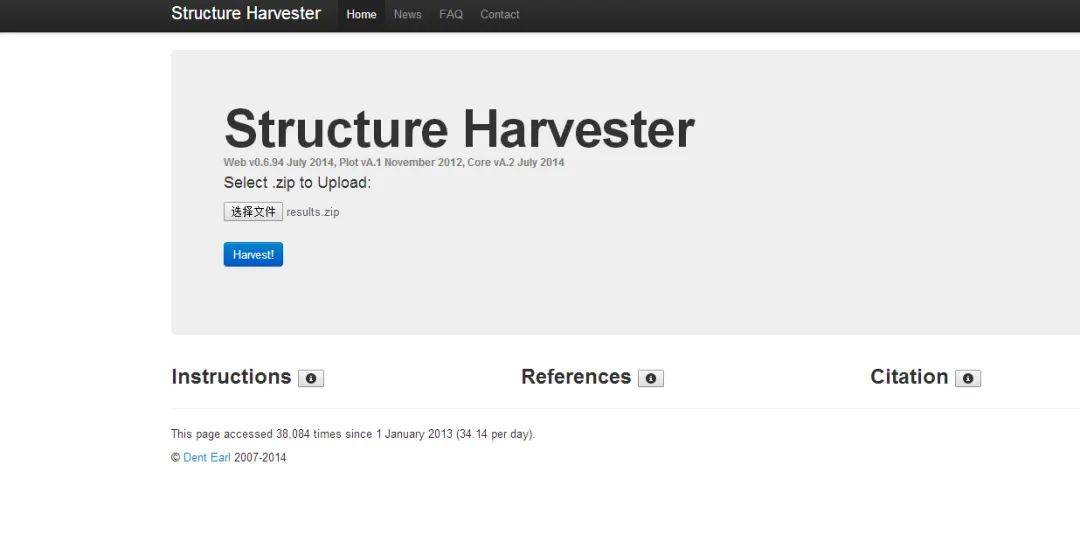 等待跑完
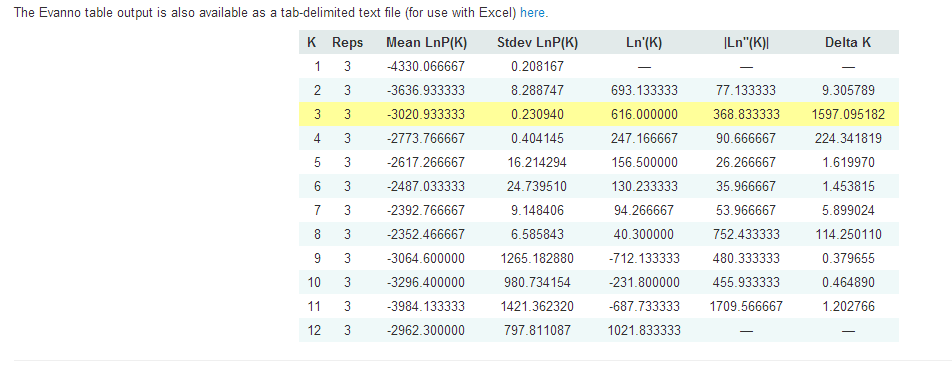
等待跑完  查看最近的K值是多少
查看最近的K值是多少 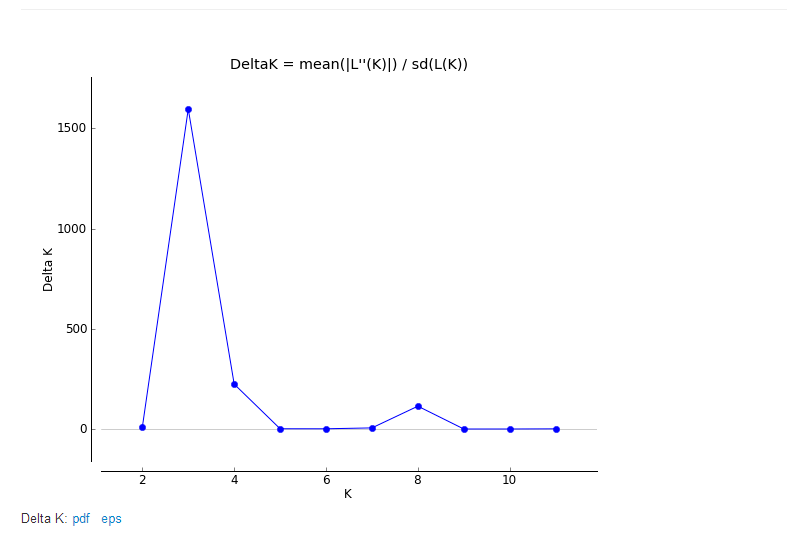
 返回structure,点击具有最近k值的结果文件
返回structure,点击具有最近k值的结果文件  点击bar plot下的show
点击bar plot下的show  结果出来了,图像可以以多种形式显示,并可保存。
结果出来了,图像可以以多种形式显示,并可保存。 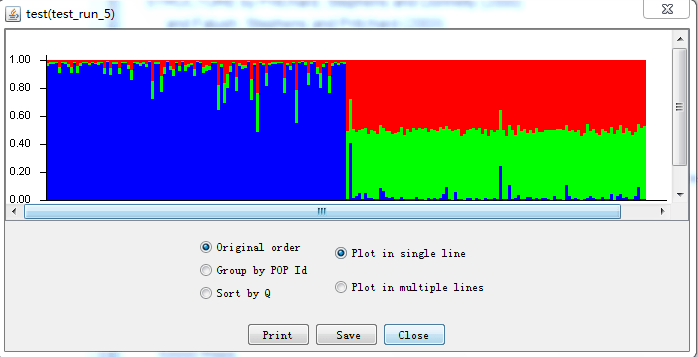 可以标上居群ID
可以标上居群ID 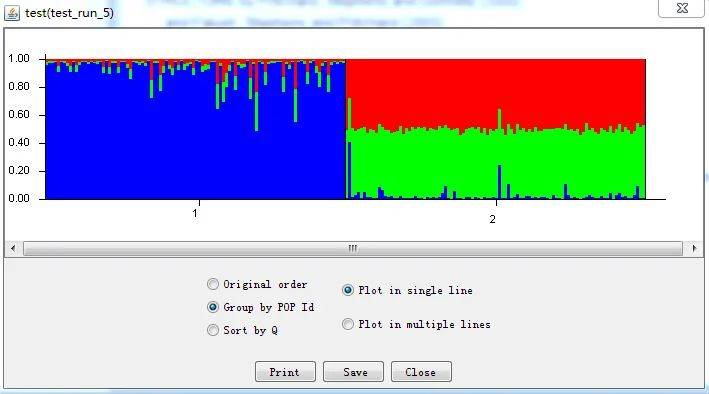 还可以根据Q值来划分
还可以根据Q值来划分 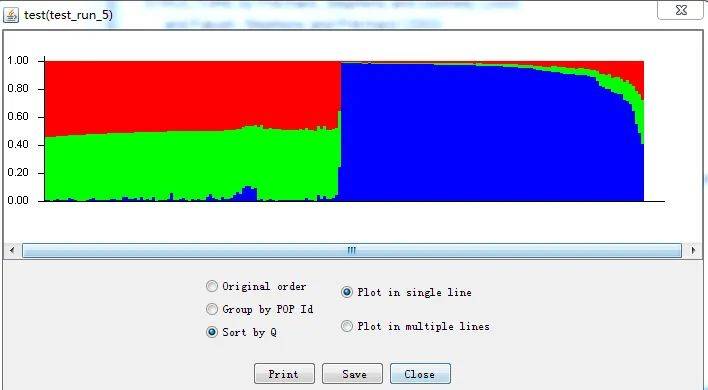 还可以分成多层来显示
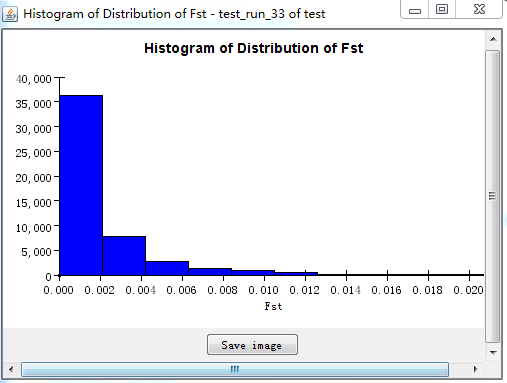
还可以分成多层来显示  此外,还有FST的图
此外,还有FST的图 
直方图
三角图 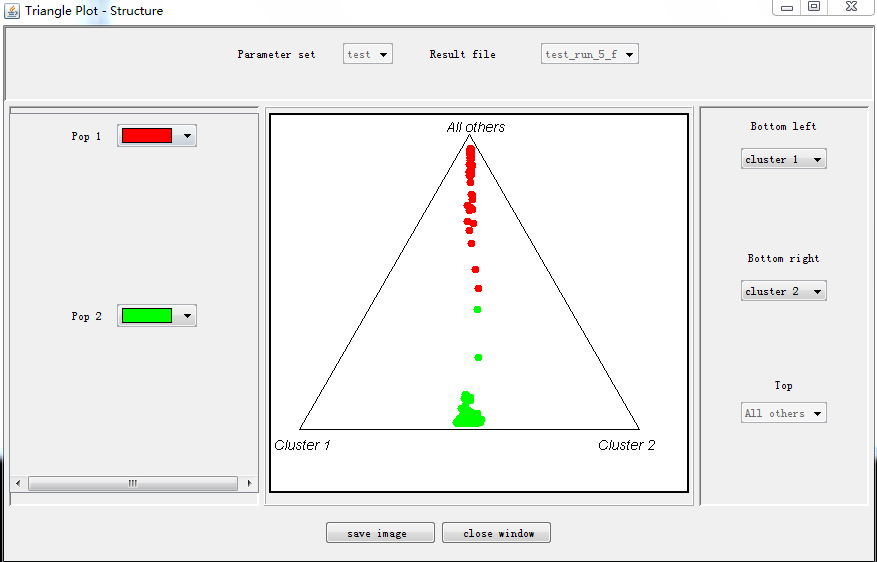 树图等
树图等 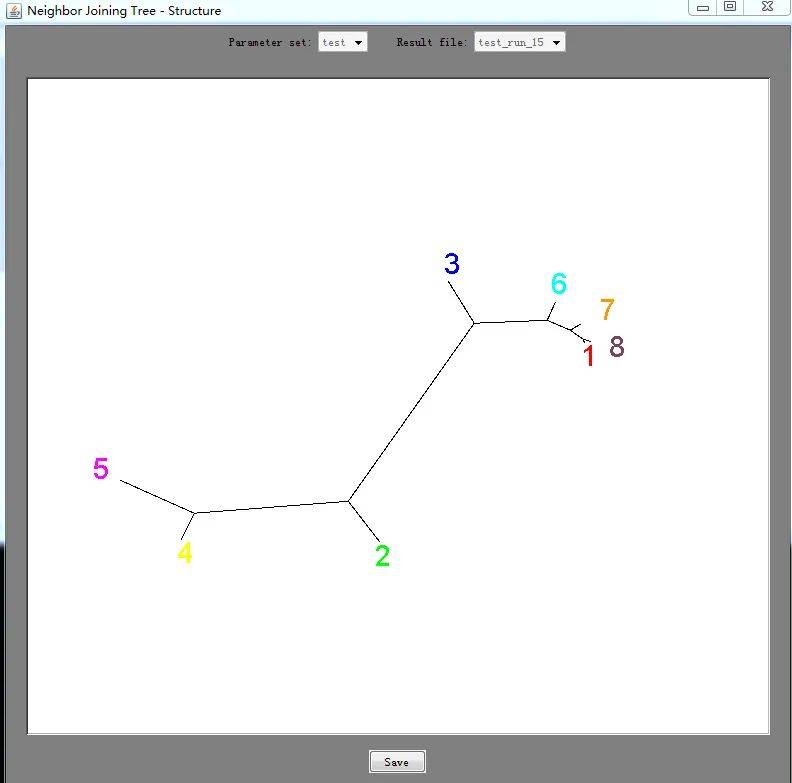 另外,也可用结果中的文件利用distruct软件进行美化处理
另外,也可用结果中的文件利用distruct软件进行美化处理 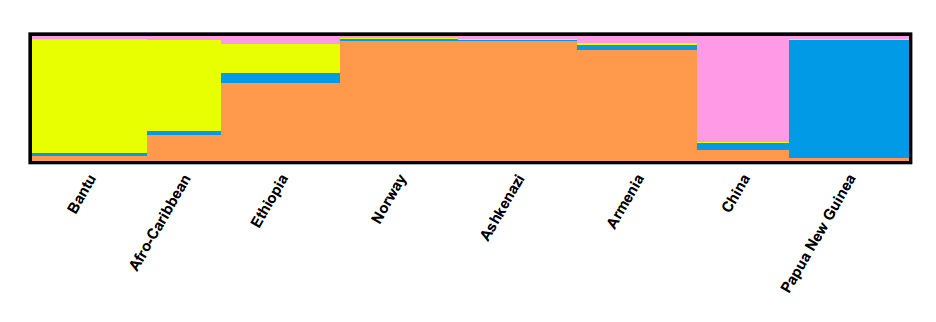
好了,以上就是Structure教程。如果文章对你有所帮助,请转发给你身边需要的人噢!
没有服务器想快速查看高通量比对数据?Tablet可以帮你
FASTQ格式详解
如何用Pathview画出高大上的基因与代谢通路热图?
使用PAML计算选择压力
测序数据到手后,如何高效的进行质量统计?
2021-03-15
用DOGMA对叶绿体或线粒体基因组进行注释
 扫码关注我们Get更多科研小工具
扫码关注我们Get更多科研小工具- 本文固定链接: https://maimengkong.com/kyjc/1638.html
- 转载请注明: : 萌小白 2023年10月8日 于 卖萌控的博客 发表
- 百度已收录
