在周一(4月2日)的《浅谈选择压力分析》一文周老师向大家介绍了选择压力分析的由来和用途,下面就可看下如何进行选择压力分析。按照群体数量,选择压力分析的方法主要可分成两类:DNA多样性的计算(单个群体内分析)和多样性水平在不同亚群间的比较(多群体分析)。
第一类方法
DNA多样性的计算(单个群体内分析)。在动植物重测序领域,选择压力分析的方法大多数是在同一个物种内,进行多样性统计和比较。最基础的方法,也是重测序文章中用的最多的方法π值的计算。
π值就是计算两两序列的差异度,然后求均值。下图有4条序列,它们如果进行两两比较,应该有6组比较。例如,序列1和2的差异度是0.1(10个碱基长度,1个差异位点),序列1和3的差异度是0.3。最终计算得到均值是0.233。这个小群体的π值=0.233。
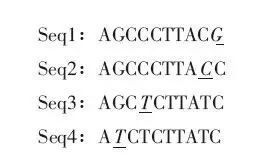
图1 多条序列的比较计算π值
这个π值就是代表了某个物种的某个群体的多样性程度(个体平均两两差异度的大小)。大部分动植物的π值都在1~5‰的水平。当然,这个数值只是整个基因组的均值。
在基因组的不同区域,由于受到不同的选择压,所以多样性水平实际上不同。例如下图,从野生种到栽培种的驯化过程中,基因组上只有第二个位点受到选择(蓝色基因型被淘汰,只有红色基因型被保留),所以只有这个位点的多样性显著降低。
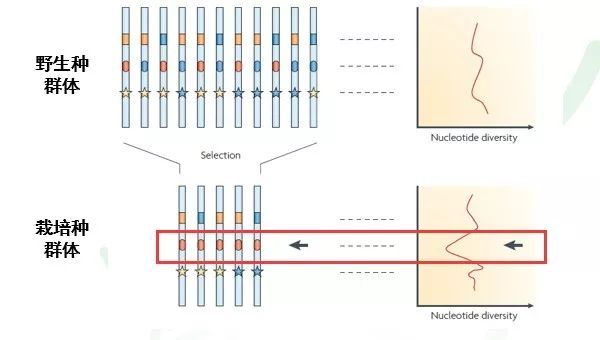
图2 基因组不同区域的多样性变化并不相同
为了寻找基因组上那些潜在最极端的受选择的区域,在重测序分析中通常我们会采用滑窗分析的方法(例如,以40kb为窗口在基因组上扫描,计算各个窗口区域π值的均值)计算基因组上不同区域的π值变化,将那些潜在π值最极端的区域(例如,最低的1%或5%)作为潜在受选择的区域。
当然,其他常见多样性分析的方法,包括ω ,Tajima’D,ZHp(用在混池测序里)等,相关方法的介绍可以见《DNA水平自然选择作用的检测》[1]
第二类方法
多样性水平在不同亚群间的比较(多群体分析)。多样性水平在亚群间的比较,包括线性相关性分析和亚群间差异比较两大类方法。
线性相关性分析类的方法,主要是计算各个位点基因型(或者基因型频率)与环境因子间的相关性(例如,海拔,温度,维度等),从而筛查与环境相关的受选择位点(例如相关性最强的前top1%的位点或区域)。这个方法在重测序文章中并不常见。更常见的是,在不同亚群间进行多样性差异比较的方法。
亚群体间多样性(π值)的比较,在重测序文献中最常见的方法是Fst(群体分化程度)和 π ratio(多样性的变化倍数)。从两个数值的计算方法,我们可以了解它们意义的不同。假设有一个物种,分为A和B两个亚群。
Fst公式:Fst=[π(亚群间)-π(亚群内)] / [π(亚群间)]
所以某个位点(或区间)的Fst数值越大,则说明在这个位点,亚群间两两个体的平均差异度要大于亚群内两两个体的差异度。其说明的问题就是:两个亚群已经发生了明显的分化(缺乏基因交流),亚群体内的个体较为相似,而亚群间的个体则差异较大。
从进化选择的角度来考虑,则说明在两个亚群体中,进化/驯化的力量对两个群体施加了不同的作用,使用A和B两个亚群在基因型(频率)上呈现了不同的偏好。
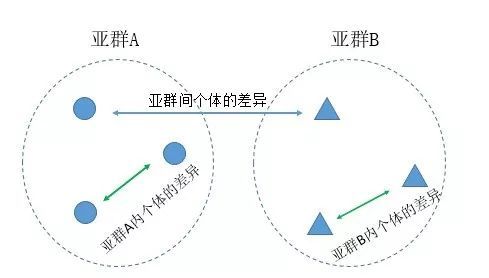
图3 两个亚群间各个差异度衡量方式的示意图
公式:π ratio= π(A)/π(B)
π ratio这个公式更加简单粗暴,直接代表某个位点(或区间)在两个亚群间的多样性差异倍数。相比Fst关心基因型的分化,π ratio关心多样性值的高低变化。因此,π ratio通常应用在驯化种和野生种的比较。
假设亚群A为野生种群体,亚群B为栽培种群体。通常,野生群A多样性较高,而栽培群体B是从野生种群体中育种选育而来,其多样性必然会降低。而B群体中,多样性降低最显著(例如 top1%)的基因组区域,往往与驯化改良的选择相关。
这些基因组区域常常携带产量高、生长快等相关的基因,在育种过程中人类会强烈选择优良的基因型,导致这些区域在栽培群体中的多样性更显著的下降。
例如在西红柿驯化历史研究的文章中[2],通过π ratio两组比较(西红柿野生群体PIM vs 地方品种群体CER,地方品种群体CER vs 商品种群体BIG)分别挖掘在西红柿驯化和改良两个阶段潜在受选择的基因。
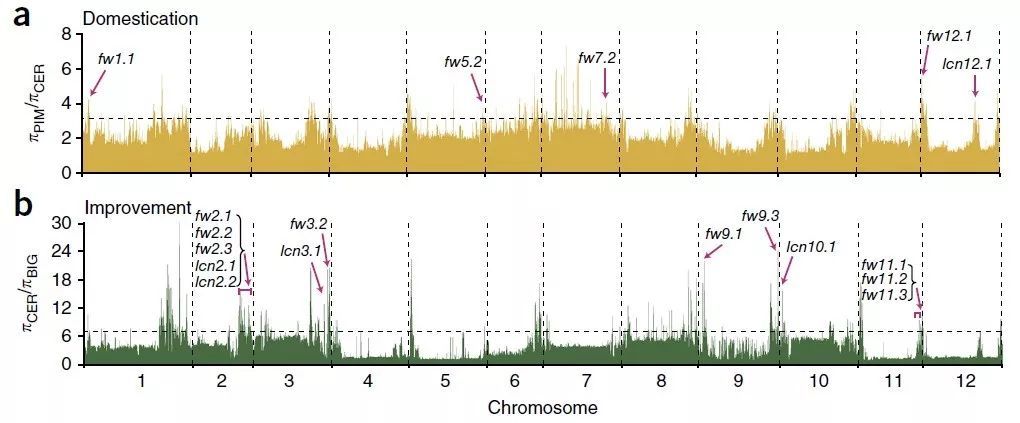
图4 利用π ratio挖掘西红柿驯化和改良两个阶段受选择的基因
以上的方法,都只是针对两个群体比较的方法。有的时候,可能会同时研究多个品种。那么,就可能会同时比较A品种与其他品种(B、C、D、E……)的分化差异。这个时候,则可以采用di分析的方法。di最早来源一篇研究10个狗品种选择分化的文章,用于解析任意一个狗品种与其他9个品种分化相关基因[3]。
为什么会有这么多方法?
上文我们介绍了选择压力分析的主流方法,但实际上在这些主流方法中还分化出了更多丰富的变种。那么,我们为什么需要这么多方法呢?
(1)不同方法适用范围不同,用于不同情况下的问题
例如,Fst可以广泛用于两个群体的分化。如果有多个群体相互比较,则可以采用di分析。但如果关心驯化过程中的多样性下降,π ratio的方法则更加有针对性。
(2)单个方法可能有较高假阳性,多个联合分析可以让结果更加可靠。
在自然群体中,除了因为选择压导致多样性变化,基因型频率随机波动导致的随机漂变也会导致多样性变化,导致结果假阳性。如果有2~3个方法联合解析,则可以降低假阳性。
例如,在狗适应性驯化研究文章中[4],作者就采用狗群体多样性下降(ZHp分析)和两个群体(狗和狼)分化程度上升两个指标的交集,来寻找狗驯化过程中受选择的基因,从而得到更加可靠的结果。
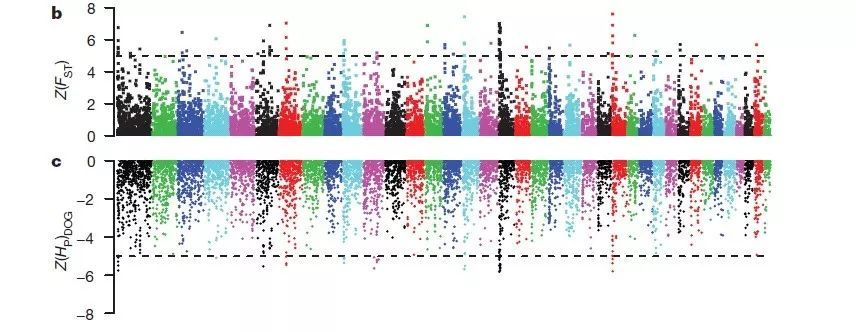
图5 两种方法结合寻找狗受选择的基因
正因为以上两点原因,基迪奥公司动植物群体选择压力分析结题报告中,提供包括π,ω ,Tajima’D,ZHp,Fst,π ratio,di在内的多种方法,便于用户可以根据自己的项目特点选择最适用的方法,挖掘目标材料受选择的基因。
最后,感兴趣的读者可通过点击 阅读原文直接观看Omicshare课堂的录播视频《群体遗传与进化选择技术交流》。也可以在基迪奥微信公众号底端菜单栏的“在线课堂”观看所有往期视屏。
今天的内容就到这里啦,接下来周老师会为大家介绍连锁分析、关联分析和选择压力分析三者的比较以及联合应用,敬请期待。
参考文献
[1] 周琦, 王文. DNA 水平自然选择作用的检测[J].动物学研究,2004, 25(1): 73-80.
[2] Lin T,Zhu G, Zhang J, et al. Genomic analyses provide insights into the history oftomato breeding[J]. Nature genetics, 2014, 46(11): 1220.
[3] Akey JM, Ruhe A L, Akey D T, et al. Tracking footprints of artificial selection inthe dog genome[J]. Proceedings of the National Academy of Sciences, 2010,107(3): 1160-1165.
[4] Axelsson E, Ratnakumar A, Arendt M L,et al. The genomic signature of dog domestication reveals adaptation to astarch-rich diet[J]. Nature, 2013, 495(7441): 360.- 本文固定链接: https://maimengkong.com/kyjc/1505.html
- 转载请注明: : 萌小白 2023年5月1日 于 卖萌控的博客 发表
- 百度已收录
