在我们平时的科研中,常常需要知道自己研究的基因组区段是否位于一些调控元件上,如enhancer,promoter或者特定蛋白结合位点(如TFBS)等。ENCODE (Encyclopedia of DNA Elements) 作为DNA调控元件百科全书整合了14,046个来自不同组织或细胞系的各类实验数据,并能通过UCSC genome browser快速可视化检索结果。
下面的时间里,编者就以检索HEK293细胞中H3K27ac这个组蛋白为例,为大家介绍ENCODE数据可视化过程。
第一步:访问ENCODE data portal主页(https://www.encodeproject.org/)
如图1,该页面介绍了ENCODE project所包含的各种数据类型以及数据来源,想要访问其中的数据,直接点击“Get Started” 即可。
图1,ENCODE data portal主页

第二步:筛选感兴趣实验类型和细胞系
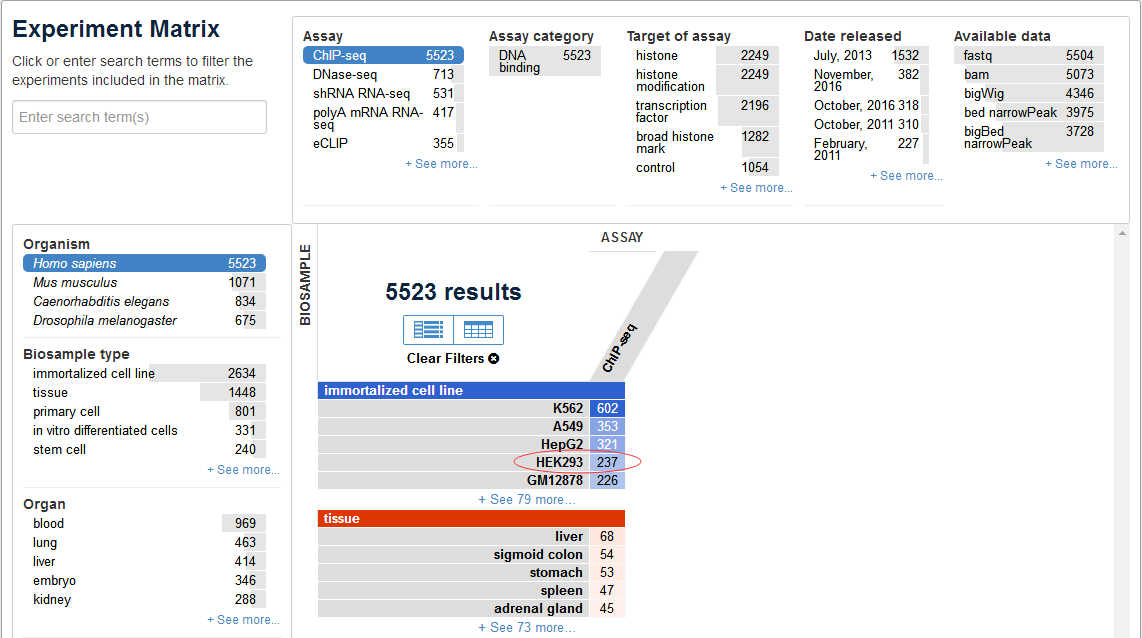
ENCODE data portal把数据按照矩阵格式存储,方便用户直接选取感兴趣数据。如图2,横排显示的是各种实验类型,竖排显示的是样本素材信息。我们直接点击横排的Chip-Seq以及竖排的Homo sapiens,然后选取HEK293(红色方框标注)。
图2,数据筛选
第三步:选择Chip-Seq目标蛋白类型
在第二步中,我们只是筛选到细胞系,这一步中,我们选择目标蛋白类型。由于组蛋白标记往往可以指示enhancer,所以这里我们以组蛋白为例进行检索(如图3)
图3,选择组蛋白
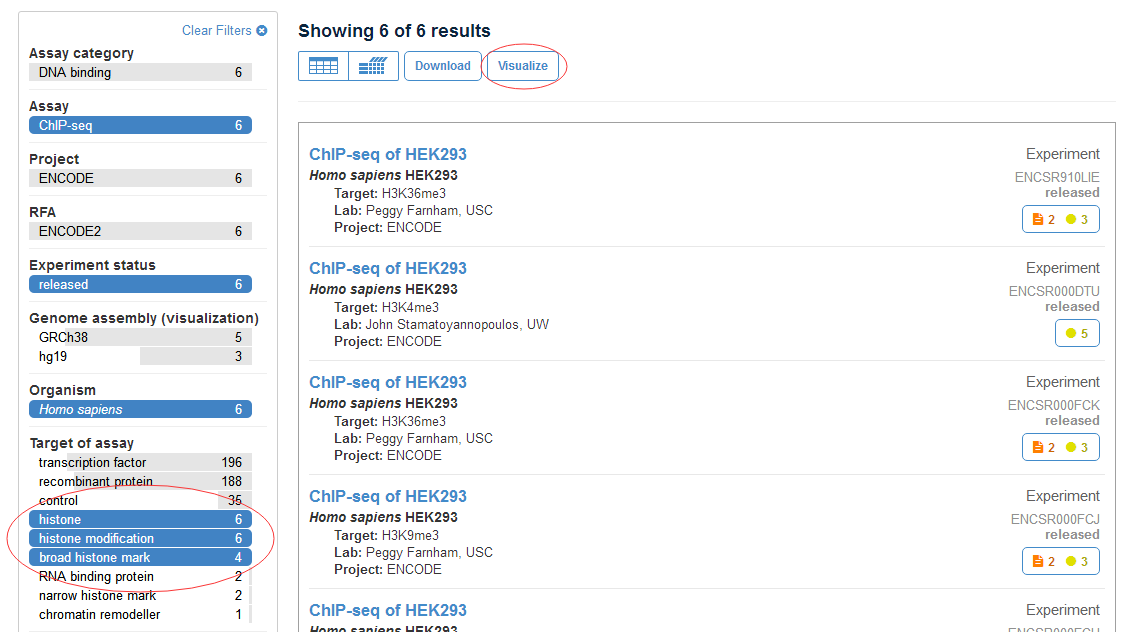
在完成以上筛选后,我们可以看到只有6个实验数据保留。这6个数据就是符合我们要求的数据,直接点击右上方“Visualize”就可以链接到UCSC genome browser。
第四步:控制UCSC显示条目
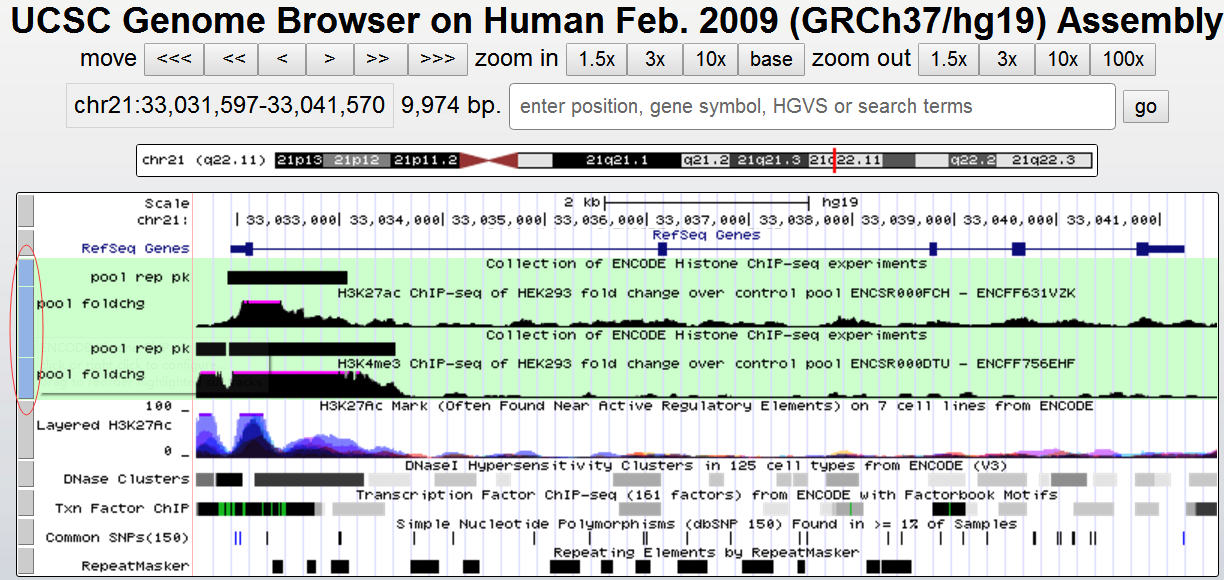
链接到UCSC后,我们发现显示效果并不理想。是因为UCSC会自动加上一些我们不需要的条目或者没有按照我们喜欢的方式显示条目。这就需要我们对有些条目进行处理。如图4,右击条目最左边竖线可以选择条目显示类型,如不需要,可以选择“hide track set”;
图4;点击红色框控制条目显示状态
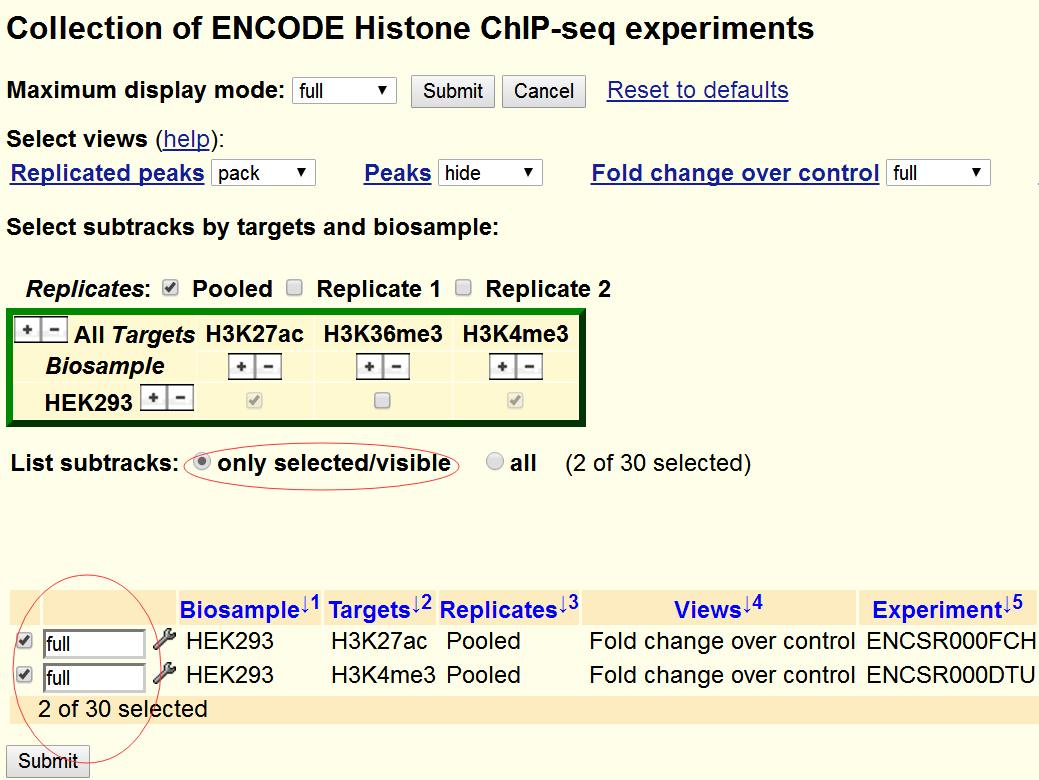
直接左键点击该竖线可以对该条目的显示内容做进一步的筛选,如图5,我们选取H3K27ac和H3K4m3作为靶蛋白,只保留跟control相比有差异的peak,提交即可。
图5,UCSC条目进一步筛选
第五步,get结果
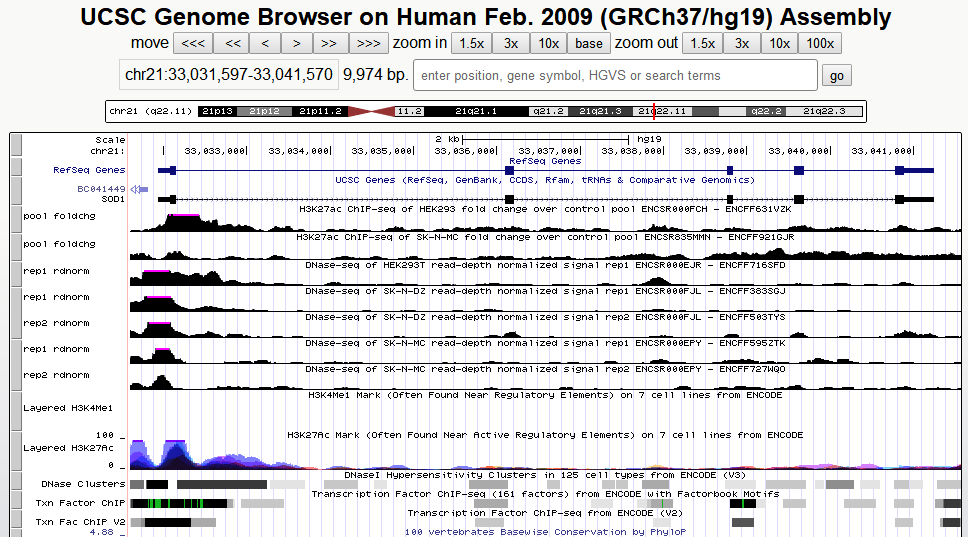
一旦你完成了所有的筛选,UCSC会记住这些筛选条件,这个时候你只需要不断地在基因组坐标栏输入感兴趣的基因组区段即可显示筛选到的内容了,如图6。
图6,最后结果展示
写在后面:
ENCODE是个非常庞大的数据库,涵盖了多种细胞及物种的数据,值得我们深入研究。小伙伴们赶快get起来这个庞大的数据资源吧~
转自生信草堂
- 本文固定链接: https://maimengkong.com/kyjc/1333.html
- 转载请注明: : 萌小白 2023年1月13日 于 卖萌控的博客 发表
- 百度已收录
