1. CHIP-seq
ChIP-Seq技术是研究蛋白质与DNA相互作用的有力工具,利用该技术可以检测转录因子与DNA的动态调控作用,各种组蛋白修饰,染色质调控因子等,转录因子与组蛋白修饰的ChIP-Seq示意图如下:
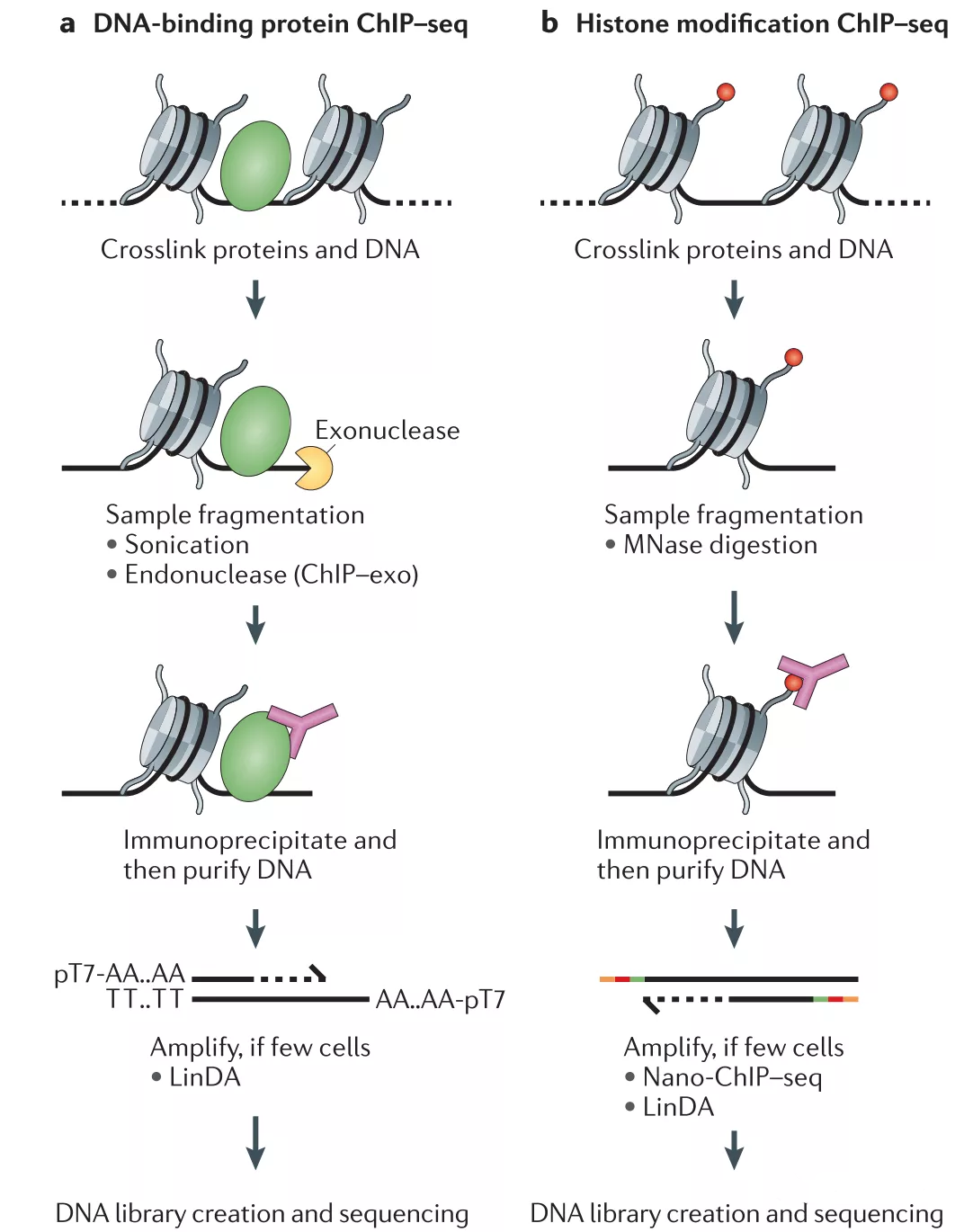
ChIP-Seq技术包括两部分:建库和测序,建库和测序的质量对于后续分析尤为关键,但是建库前ChIP实验的设计也同样重要。从实验的操作来讲ChIP主要分为两大类,一类是X-ChIP,一类是N-ChIP。其中N-ChIP不涉及甲醛交联,更适合那些和DNA有强相互作用的蛋白ChIP-seq实验,主要就是组蛋白;而X-ChIP采用甲醛交联就没有这个限制,广泛适用于转录因子, 组蛋白, 聚合酶等蛋白的结合片段测序。
实验流程、建库流程、分析流程:
1.1. Chip-seq可以告诉我们什么信息
chip-seq可以告诉我们一些什么信息?
基础分析系列:
-
我们实验处理后送样测序的数据基本情况(测序下机数据质量情况)
-
这些测序数据都分布在参考基因组的什么位置,以及比对结果如何(比对参考基因组情况)
-
这些reads的分布有哪些区域具有显著成峰(Peak)特征(CallPeak情况)
-
这些Peak区域可能影响到的基因是什么,这些Peak距离基因多远,又位于什么区域(Peak附近的临近基因注释结果及可视化情况统计)
-
这些基因都普遍具有什么样的功能(富集到什么通路上,临近注释基因集的富集结果)
-
这些Peak结合区域,都可能结合到一些什么样的蛋白(motif预测情况)
高级分析系列:
-
差异Peak分析:可以寻找到两组不同生物模型比较表观水平的改变差异
-
Chip-seq与RNA-seq联合分析:可以寻找一些表观水平差异与基因转录水平改变的关联性
-
超级增强子(SEs, Super-enhancers )鉴定分析:可以找到影响转录水平异常增加的一系列表观水平的相互作用区域
-
核心调控网络(CRC, Core transcription regulatory circuitry )分析:可以从SEs中通过调控网络关系找到的核心影响转录因子
1.1.1. chip seq 报告解读
我们的Chip-seq分析报告展示:
分析案例:
报告解读:
1.1.2. Peak_Motif分析意义(homer)
homer_motif 分析能给到我们什么信息?
以下我们将分两部分分别介绍 TF-Chip 和 Histone-Chip。
1.2. TF-Chip 与 Histone-Chip
1.2.1. TF Chip 研究的核心问题
是什么调控了我们的基因? 转录因子调控了谁?本质是蛋白质对DNA的调控。此处"DNA"包括能够翻译成蛋白质的蛋白编码基因、能够转录成lncRNA/circRNA/miRNA的基因。
TF(Transcription factor,转录因子):
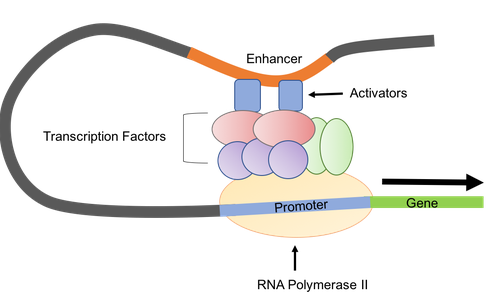
目前,两种高通量实验和一种计算方法能够解决这个调控的上游原因寻找问题:
-
Plan A:ChIP-seq,最直接,最有效
-
Plan B:DNase-seq/ATAC-seq 或 RNA-seq,曲线救国; 与ChIP-seq整合分析更准确
-
Plan C:基于motif预测
由于ChIP-seq的直接与有效性,我们此处着重介绍CHIP-seq方法:通过前期实验与建库测序得到原始下机数据,随后将CHIP实验交联蛋白附近的DNA片段对比上基因组的显著富集区域通过callPeak方式找出来,并对其进行一系列分析。
1.2.2. Histone chip 研究的核心问题
Histone chip 的背景知识
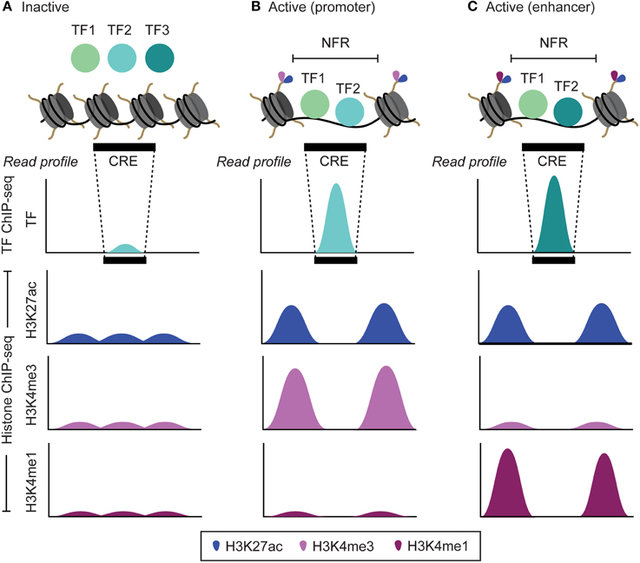
Histone chip 研究的核心是什么?
-
寻找组蛋白修饰对于我们基因调控的影响,寻找转录调控发生改变的根本原因。
Histone chip 整套流程可以告诉我们的什么信息?
-
基本内容同上,不同在于结果的意义:在基因组上哪些区域可能发生了组蛋白修饰。
1.3. SEs鉴定
超级增强子(SEs, Super-enhancers )鉴定分析:可以找到影响转录水平异常增加的一系列表观水平的相互作用区域
SEs鉴定能给出什么信息?
1.4. 高级分析系列
1.4.1. chip-seq的结果定量与比较
如何对 chip-seq 的结果定量? Chip seq的定量与比较(Diffbind)告诉了我们什么信息?
原理概述: 我们是如何计算定量结果与差异Peak的?
分析案例: Chip差异Peak分析结果及报告
1.4.2. RNA-seq 和 Chip-seq 的联合分析
RNA-seq 和 Chip-seq 的联合分析
-
意义在于寻找转录水平变化的根本原因,从表观水平的变化去解释。
-
“RNA seq 和 组蛋白Chip 联合分析” 与 “RNA seq 和 转录因子Chip 联合分析” 分析流程相同,不同在于得到结果的意义。
1.4.3. CRC分析
CRC分析能给出什么信息?
-
CRC(Core transcription regulatory circuitry ):核心调控网络分析
-
通过调控网络关系分析,可以从SEs中通过找到的核心影响转录因子。助力精确定位核心转录因子
2. RNA seq
2.1. RNA-seq能告诉我们一些什么信息?
转录组是指特定组织或细胞在某个时间或某个状态下转录出来的所有RNA的总和,主要包括mRNA和⾮编码RNA。转录组测序是基于Illumina测序平台,研究特定组织或细胞在某个时期转录出来的所有mRNA,是基因功能与结构研究的基础,对理解生物体的:发育和疾病的发生具有重要作用。
RNA测序(RNA-seq)在过去十年里逐渐成为全转录组水平分析表达和研究mRNA差异剪接必不可少的工具,应用于如单细胞基因表达、RNA翻译(translatome),RNA结构组(structurome), RNA-RNA/RNA-Protein的相互作用、空间转录组学(spatialomics)等多种RNA层面的研究(R. Stark, Grzelak, and Hadfield 2019)。
其中表达水平的探究是转录组领域最热门和基础的方向:利用转录组数据来识别转录本和表达定量,从而对造成细胞/组织/个体间不同状态的差异的内部原因进行诊断分析,挖掘关键基因。
在不同背景下比较mRNA水平:
-
同一物种,不同组织:研究基因在不同组织的表达情况,找到细胞组织特异性的基因;
-
同一物种,同一组织:研究基因在不同处理或条件下的表达变化,挖掘特异的功能基因,指导后续物种改良、疾病诊断等;
-
同一组织,不同物种:研究基因的进化关系;
-
时间序列实验:基因在不同时期的表达情况与其发育的关系,找到发育阶段特异性的基因;
RNA-seq能告诉我们一些什么信息?
-
各个基因在不同生物模型的表达情况
-
不同生物模型之间的显著差异表达基因有些什么
-
这些显著差异表达基因都能富集到什么生物学功能上
2.2. mRNA 建库,分析
建库与分析流程:mRNA实验建库流程与分析流程(polyA 建库)
分析案例: 转录组测序及分析报告
2.3. ncRNA seq 流程
2.3.1. ncRNA的建库与分析流程
ncRNA(micRNA lncRNA CIRCRNA)的建库与分析流程如下:
(miRNA 特异链 建库)
分析案例:
2.3.2. ncRNA的靶基因寻找方案
我们通过一系列分析筛选到的ncRNA列表后,并不能直接确定它能影响的生物学功能,因此,我们需要通过分析它对应能影响到的靶标基因,通过靶标基因来确定相关生物学功能。
但以上分析得到的ncRNA(micRNA lncRNA CIRCRNA)又如何找到其靶基因?
ncRNA(micRNA,lncRNA,CIRCRNA)靶标基因寻找方法:
2.4. go与kegg的意义与David网站
富集分析简介: func_comm/enrich/web_enrich.md
- 本文固定链接: https://maimengkong.com/kyjc/1296.html
- 转载请注明: : 萌小白 2022年11月20日 于 卖萌控的博客 发表
- 百度已收录
