Pfam: the protein families database11.501Nucleic Acids Res . 2014 Jan;42(Database issue):D222-30. doi: 10.1093/nar/gkt1223. Epub 2013 Nov 27.
Abstract
Pfam, available via servers in the UK (http://pfam.sanger.ac.uk/) and the USA (http://pfam.janelia.org/), is a widely used database of protein families, containing 14 831 manually curated entries in the current release, version 27.0. Since the last update article 2 years ago, we have generated 1182 new families and maintained sequence coverage of the UniProt Knowledgebase (UniProtKB) at nearly 80%, despite a 50% increase in the size of the underlying sequence database. Since our 2012 article describing Pfam, we have also undertaken a comprehensive review of the features that are provided by Pfam over and above the basic family data. For each feature, we determined the relevance, computational burden, usage statistics and the functionality of the feature in a website context. As a consequence of this review, we have removed some features, enhanced others and developed new ones to meet the changing demands of computational biology. Here, we describe the changes to Pfam content. Notably, we now provide family alignments based on four different representative proteome sequence data sets and a new interactive DNA search interface. We also discuss the mapping between Pfam and known 3D structures.
Pfam(http://pfam.xfam.org/)是一个被广泛使用的蛋白家族结构域数据库,其依赖于多序列比对和隐马尔可夫模型(HMMs)鉴定一个或多个蛋白质功能结构域。结构域的不同组合方式产生的蛋白质在自然界中各种不同。因此蛋白结构域的鉴别对分析蛋白质的功能来说尤其重要。
一
Pfam数据库概述
每个family以PF编号**标识,family可以分为以下5种类型:
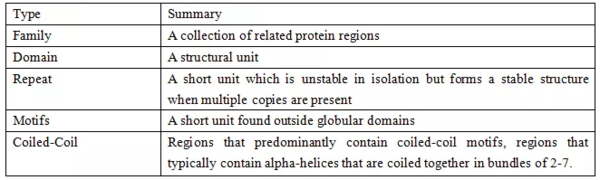
2、clans
3、proteones
物种的蛋白质组信息说明。查询蛋白质结构域,蛋白质结构域超级家族,物种蛋白质组信息。
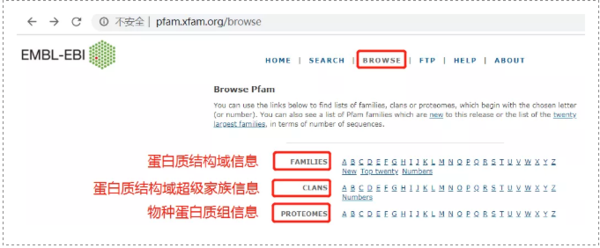
二
在线注释
1、Sequence
以一条蛋白质序列为例,查找这条蛋白质序列上的结构域,可以用Sequence入口查找:点击->Sequence->输入序列->Submit。
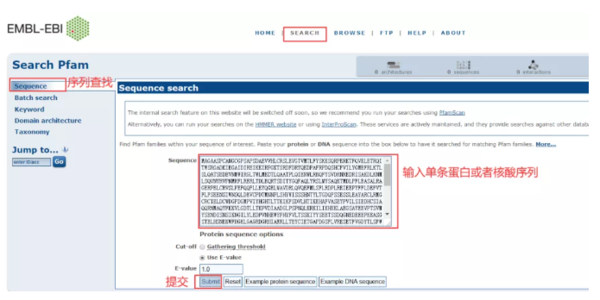
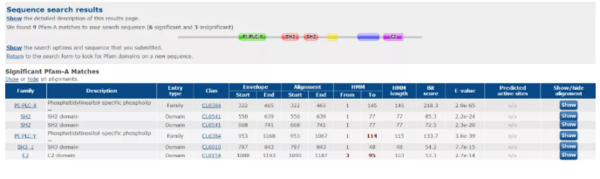
得到这条蛋白序列上的结构域信息,以及Pfam-A数据库比对上序列,如下:
3、Keyword
Keyword提供了通过关键词查找,例如输入关键词:apoptosis(细胞凋亡)。
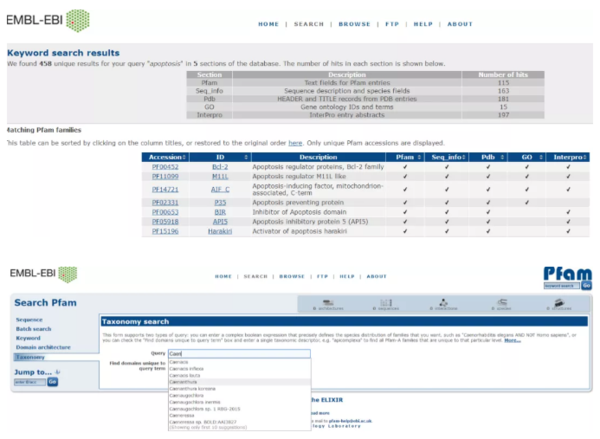
4、Taxonomy
Jump to是通过输入Pfam ID查找。
三
Pfam本地化配置
1、数据库和软件下载
√ PfamScan.pl工具(ftp://ftp.ebi.ac.uk/pub/databases/Pfam/Tools/PfamScan.tar.gz)
Pfam-A.hmm.gz
Pfam-A.hmm.dat.gz
Pfam-B.hmm.gz
Pfam-B.hmm.dat.gz
active_site.dat.gz
√ HMMER3(http://www.hmmer.org/download.html)
√ Anaconda3(https://repo.anaconda.com/archive/Anaconda3-2019.10-Linux-x86_64.sh)
2、安装
Anaconda3安装
sh Anaconda3-2019.10-Linux-x86_64.sh -b -p $HOME/anaconda3

修改环境变量
export PATH=$HOME/anaconda3/bin:$PATH

export PERL5LIB="$HOME/PfamScan:$PERL5LIB"

HMMER3的安装
conda install -c bioconda hmmer=3.1b2

Moose的安装
cpan Moose#注意该软件建议用root账号安装

hmmpress Pfam-A.hmm

perl $HOME/PfamScan/pfam_scan.pl -fasta xx.faa -dir $HOME/PfamScan/db -outfile xx.pfam.xls -clan_overlap -as -cpu 16 -e_seq 1e-5 -e_dom 1e-5

-dir Pfam_data_dir包含Pfam数据库文件的目录[必须]
-fasta fasta_file 包含序列的输入文件名,必须为蛋白序列 [必须]
-outfile output_file 输出文件名 [不指定则输出在命令行中]
-e_seq 序列E-value阈值 [不指定则使用默认阈值]
-e_dom 结构域E-value阈值 [不指定则使用默认阈值]
-b_seq 序列bit score阈值 [不指定则使用默认阈值]
-b_dom 结构域bit score阈值[不指定则使用默认阈值]
-clan_overlap 允许不同上级分类的序列重叠 [默认关闭]
-align 在结果中显示比对片段 [默认关闭]
-as 预测Pfam-A数据库匹配的active sites[默认关闭]
-cpu 并行分析的CPU数目 [默认全部]
-translate [mode] 将输入序列视为DNA,并在搜索前使用6框翻译的方法进行转换。如果翻译模式[mode]被指定,则必须为"all"或者"orf"。"all"表示完整翻译,包括终止子并且不产生单独的ORFs;"orf"表示只翻译和报告长度大于20的ORFs。[默认关闭]
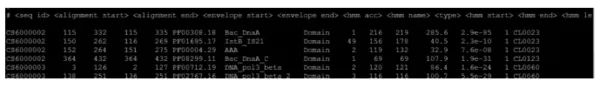
输出结果说明:
(1) seq_id:蛋白序列编号
(2) alignment start:蛋白序列比对的起始位置
(3) alignment end:蛋白序列比对的终止位置
(4) envelope start:蛋白序列结构域的起始位置
(5) envelope end:蛋白序列结构域的终止位置
(6) hmm acc:比对到pfam结构域的ID
(7) hmm name:pfam结构域名称
(8) type:pfam结构域类型
(9) hmm start:比对到结构域的起始位置
(10) hmm end:比对到结构域的终止位置
(11) hmm length:pfam结构域的长度
(12) bit score:比对打分分值
(13) E-value:比对的E值
(14) Significance:比对序列的显著性
(15) Clan:蛋白结构域超级家族名称
(16) predicted_active_site_residues:比对的序列是否位于酶的活性部位
- 本文固定链接: https://maimengkong.com/kyjc/1121.html
- 转载请注明: : 萌小白 2022年7月10日 于 卖萌控的博客 发表
- 百度已收录
