之前介绍了如何通过使用GDCRNATools这个包构建ceRNA网络,可以说整个过程非常简单和明了,对于我们学医的小朋友来讲,是个极好用的工具。但是这个包除了构建网络以外,还有很多附加的功能。要知道一篇ceRNA的文章,不单单只有那张网络图而已,还需要其他内容进行修饰和填充。那我们来看看这个包到底还有哪些功能?
前两期链接:
一
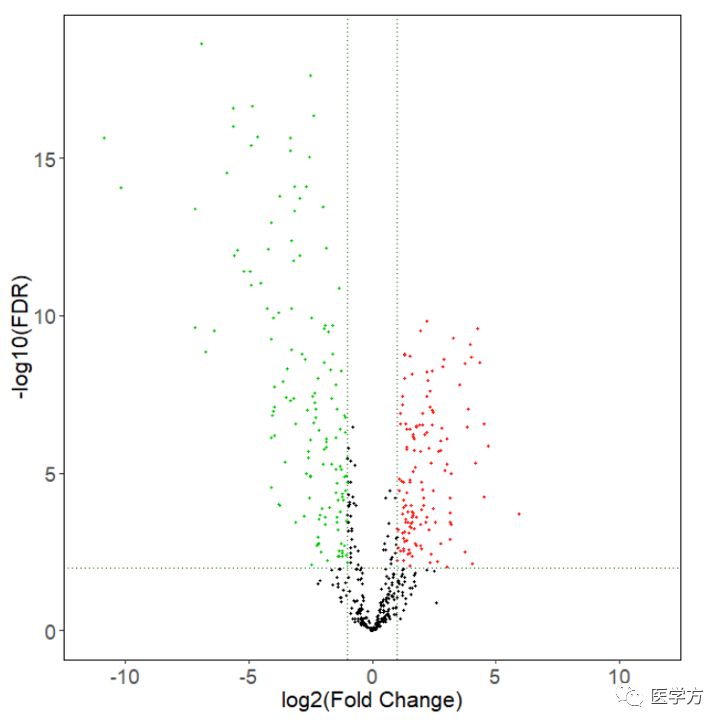
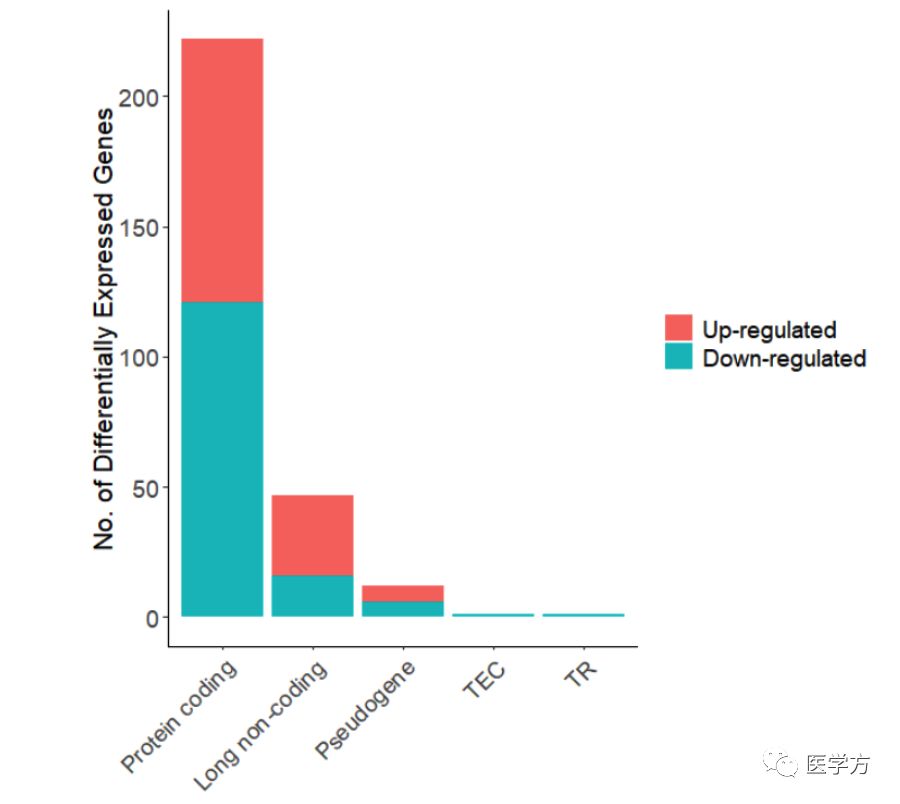
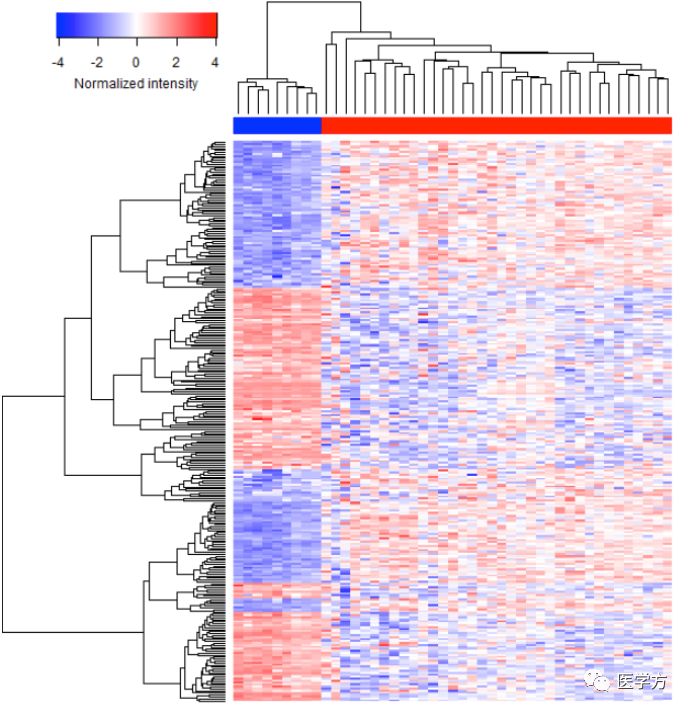
这三条代码产生了下面几张图,具体就不解释了,一看就知道哈
二

输入上述的代码,这个时候会跳出一个交互界面,该界面反映的就是在差异的lncRNA与差异的蛋白编码的mRNA表达量之间的相关关系。这里会自动给出相关系数和P值。在这个交互界面里,大家可以鼠标点击选择任一一个差异的lncRNA或mRNA,界面里都会很快帮你画出相关关系的图。
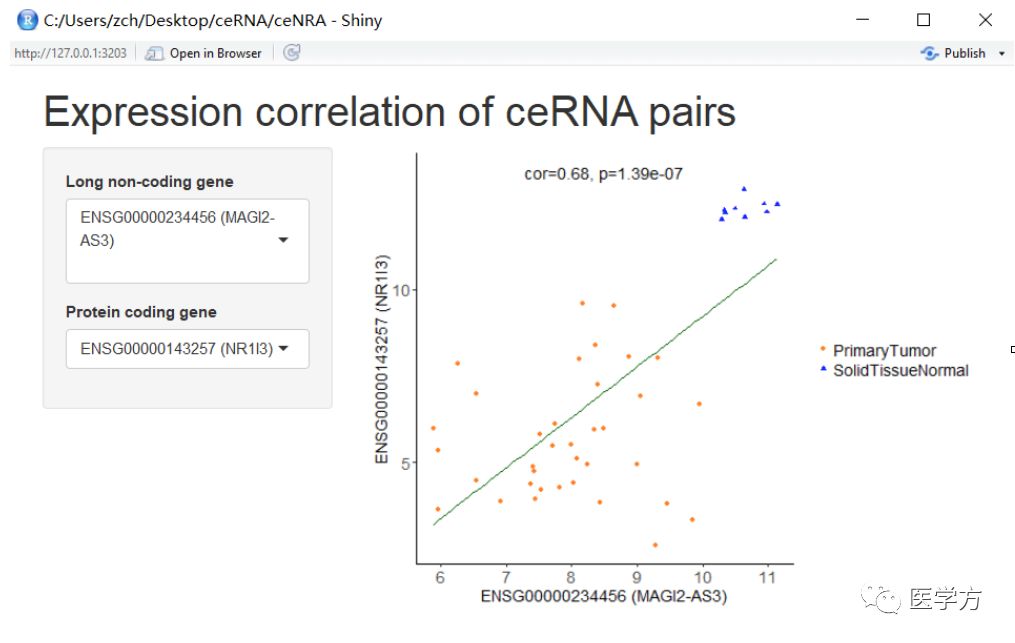
三
生存分析

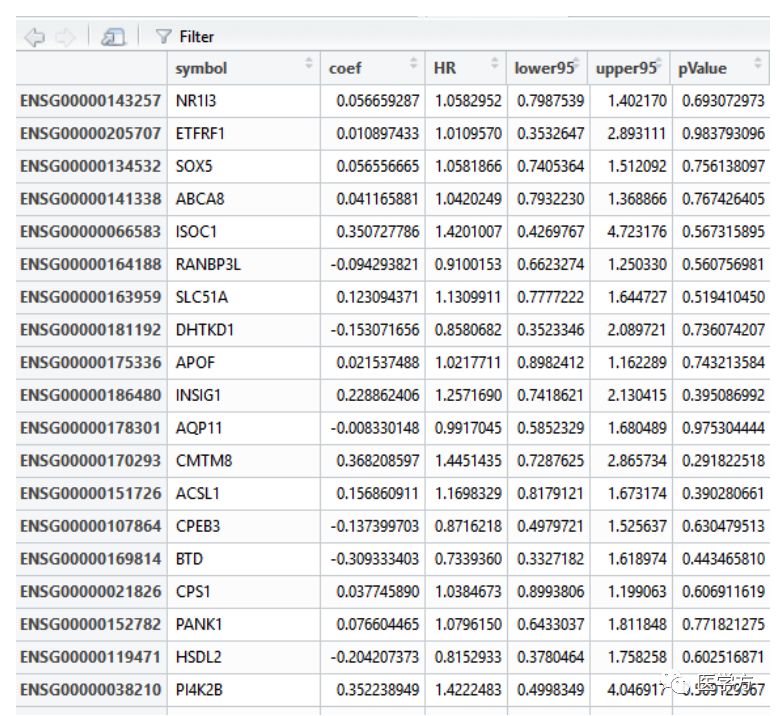
上述两条代码非常类似,只是方法不太一样,一个用了cox单因素的方法,而另外一个用了K-M的方法筛选与预后相关的差异基因。最后结果会显示基因的名字,系数,HR及其置信区间,还有p值。大家根据最后的p值来挑选出与预后相关的基因。
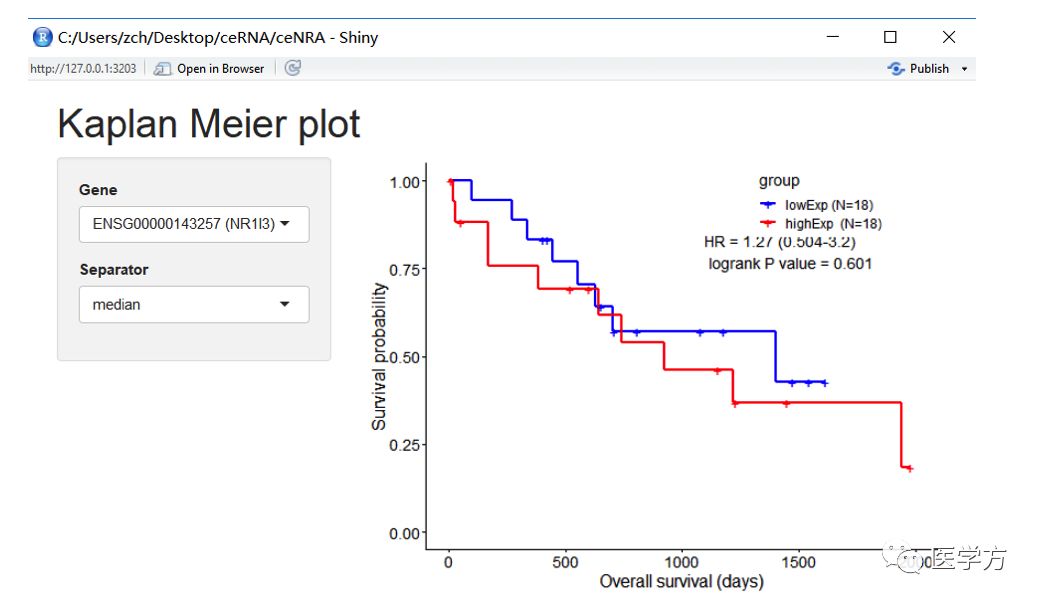
另外,大家输入下面的代码,也会跳出一个交互界面。在这个界面中,大家也是可以任意选择一个基因,随即就会跳出生存曲线,同样会给出HR值,p值。值得简单一提的是,这边做生存分析是根据该基因表达量的高低选择中位数来区分的,进行比较两组之间的生存预后。
四
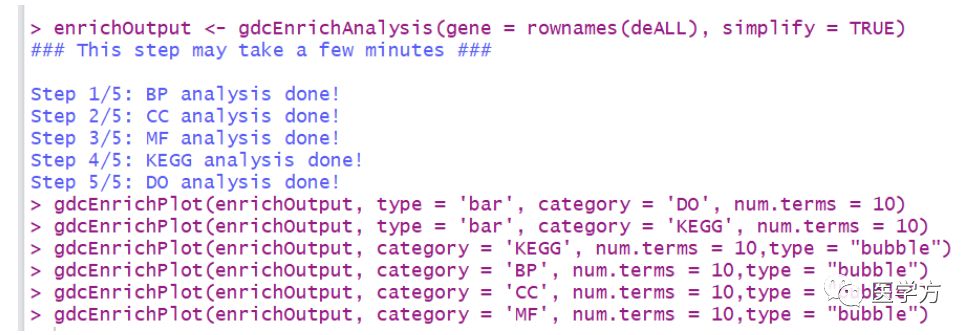
上述的代码主要执行的是我们差异的基因的功能分析,然后用gdcEnrichPlot这个函数进行绘制。其中category这项,大家可以选择BP,CC,MF,KEGG和DO,这些选项都是大家平时经常在用的分析。然后type就是我们结果的展示的形式,可以是柱状图(bar),也可以是气泡图(bubble)。大家根据自己的需要进行选择。以下就是功能富集的几张结果图。
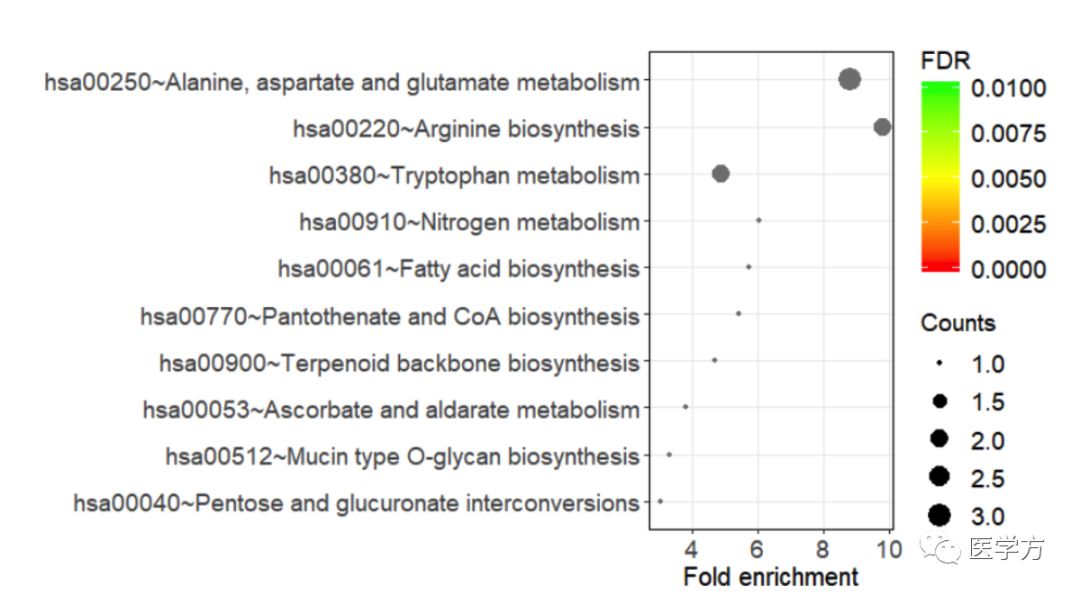
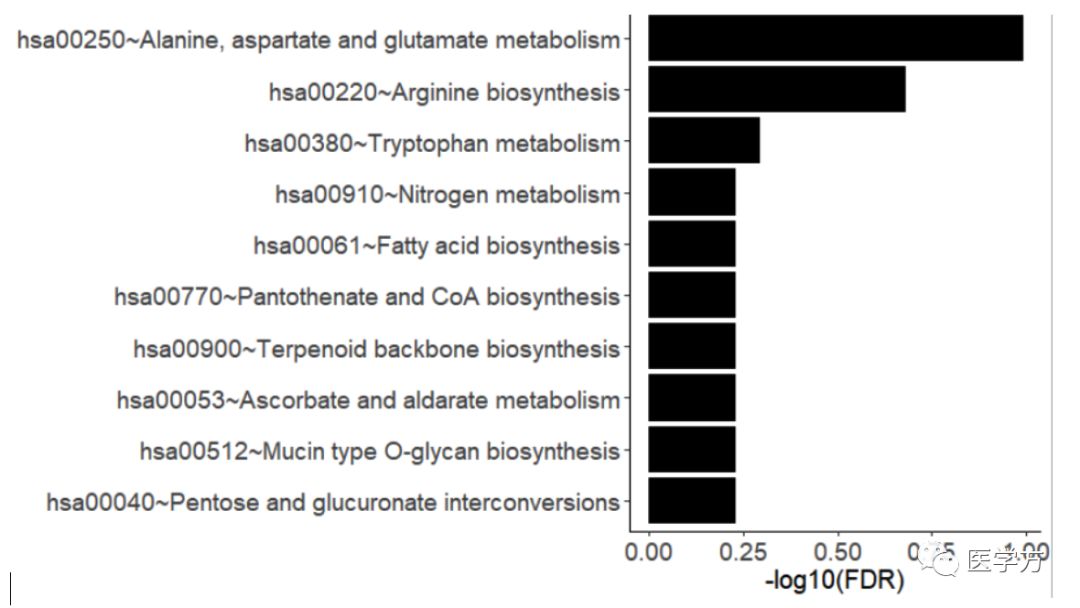
最后,大家有时候会在很多文章中会看到某个基因的通路图,一看上去就很高大上,其实这个功能也可以通过我们这个包进行实现。大家只有输入下面的代码即可,最后也会跳出一个交互界面,大家鼠标点击任何一个基因,这个界面会立刻跳出该基因所在的通路图。

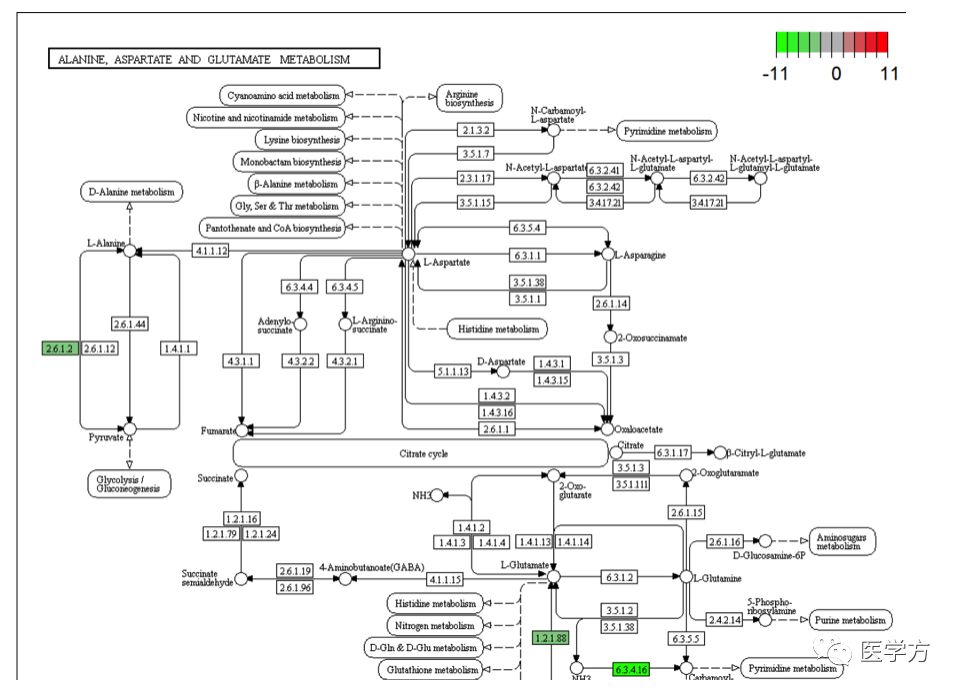
总结
以上就是这个包的全部内容,但是想提醒大家的是,虽然这个包非常人性化,提供了很多交互界面,甚至很多图都可以通过一两条代码就能产生。但是大家也千万别太过依赖于这些功能。毕竟很多文章都需要我们做一些个性化的结果和图。这就要求我们灵活应该这些代码的同时,得利用这些中间文件,定制我们自己文章所需要的结果和图。只有这样,才能真正意义上掌握了这个包。
转自医学方
- 本文固定链接: https://maimengkong.com/kyjc/1013.html
- 转载请注明: : 萌小白 2022年6月23日 于 卖萌控的博客 发表
- 百度已收录
